Реконструкция филогенетических деревьев
С помощью таксономического сервиса NCBI определим к каким таксонам относятся отобранные ранее бактерии. Результаты преставлены в табл.1.
Табл.1. Таксономия отобранных видов
| № | Название | Мнемоника | Тип | Класс | Отряд | Семейство |
| 1 | Bacillus anthracis | BACAN | Firmicutes | Bacilli | Bacillales | Bacillaceae |
| 2 | Bacillus subtilis | BACSU | Firmicutes | Bacilli | Bacillales | Bacillaceae |
| 3 | Clostridium botulinum | CLOB1 | Firmicutes | Clostridia | Clostridiales | Clostridiaceae |
| 4 | Clostridium tetani | CLOTE | Firmicutes | Clostridia | Clostridiales | Clostridiaceae |
| 5 | Enterococcus faecalis | ENTFA | Firmicutes | Bacilli | Lactobacillales | Enterococcaceae |
| 6 | Finegoldia magna | FINM2 | Firmicutes | Clostridia | Clostridiales | Clostridiales Family XI. Incertae Sedis |
| 7 | Geobacillus kaustophilus | GEOKA | Firmicutes | Bacilli | Bacillales | Bacillaceae |
| 8 | Lactobacillus acidophilus | LACAC | Firmicutes | Bacilli | Lactobacillales | Lactobacillaceae |
На рис.1. представлено филогенетическое дерево, на котором обозначены таксоны. Таким образом, пять организмов относятся к классу Bacilli, из них три к отряду Bacillales и два к отряду Lactobacillales, и три к классу - Clostridia, к отряду Clostridiales.
Рис.1. Филогенетическое дерево с указанием таксонов. Красным обозначены классы, синим - отряды, зеленым - семейства.
С помощью Swiss-Prot получим последовательности рибосомальных белков S2 из восьми отобранных бактерий. Поместим все последовательности в один файл sequences.fasta. Импортируем невыровненные последовательности в JalView и воспользуемся программой MUSCLE, чтобы получить выравнивание, которое представлено на рис.2. Также выравнивание преставлено в fasta-файле alignment.fasta и проекте JalView alignment.jar.

Рис.2. Выравнивание рибосомальных белков S2 в "блочной" форме из восьми отобранных ранее бактерий. Выравнивание окрашено по проценту идентичности.
Реконструируем филогенетическое дерево четырьмя методами, доступными из JalView. Далее поочередно сравним полученное дерево с правильно составленнным.
| Average distance tree using % identity | Правильное дерево |
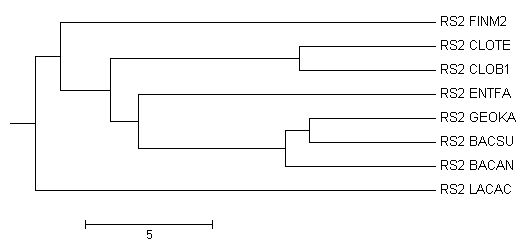
|
| Average distance tree using BLOSUM62 | Правильное дерево |
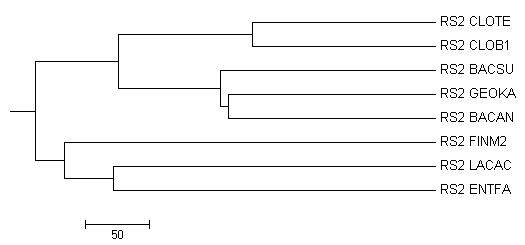
|
| Neighbour joining tree using % identity | Правильное дерево |
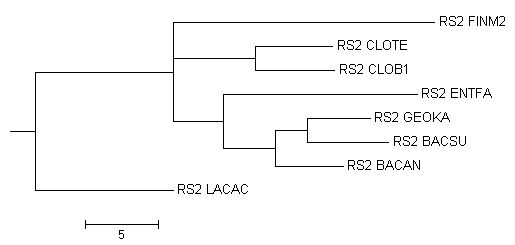
|
| Neighbour joining tree using BLOSUM62 | Правильное дерево |
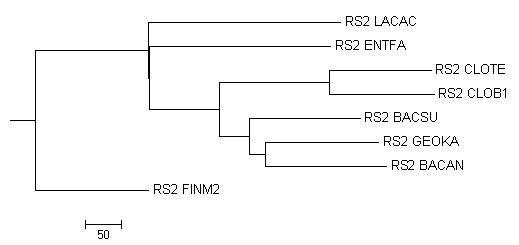
|
Реконструируем дерево методом "Maximum Parsimony", для этого в программу MEGA импортируем выравнивание в fasta-формате alignment.fasta. Полученное дерево представлено на рис.2. Далее укореним дерево в наиболее правильную ветвь(рис.3).
Рис.2. Дерево, полученное методом "Maximum Parsimony"
Рис.3. Укорененное дерево
Как можно заметить, дерево отличается от правильного, но незначительно. Из пяти нетривиальных ветвей, присутствующих в правильном дереве, три присутствуют и в реконструированном методом "Maximum Parsimony":
Присутствуют: {GEOKA,BACAN,BACSU} против {LACAC,ENTFA,FINM2,CLOTE,CLOB1}, {FINM2,CLOTE,CLOB1} против {GEOKA,BACAN,BACSU,LACAC,ENTFA}, {CLOTE,CLOB1} против {GEOKA,BACAN,BACSU,LACAC,ENTFA,FINM2}.
Отсутствуют: {BACAN,BACSU} против {GEOKA,LACAC,ENTFA,FINM2,CLOTE,CLOB1}, {LACAC,ENTFA} против {GEOKA,BACAN,BACSU,FINM2,CLOTE,CLOB1}.
Это объясняется тем, что для реконструкции дерева, мы рассматриваем только один рибосомный белок S2.