Известны два основных типа вторичной структуры трансмембранных белков: альфа-спирали и бета-баррели. В таблице 1 представлены основные параметры шести случайных белков: трех трансмембранных бета-баррелей и трех трансмембранных альфа-спиральных белков, найденных с помощью базы данных OPM .
Таблица 1. Параметры для трансмембранных белков
| PDB код | Тип | Какая мембрана | Толщина гидрофобной части мембраны в ангстремах | Медиана числа остатков в одном трансмембранном участке |
| 2m3g | спираль | Внутренняя мембрана Nostoc sp. | 31.8 ± 0.8 | 18 |
| 4dkl | спираль | Плазматическая мембрана Mus musculus | 32.0 ± 1.0 | 25 |
| 3a7k | спираль | Мембрана архей Natronomonas pharaonis | 33.8 ± 1.4 | 20 |
| 1qj8 | баррель | Внешняя мембрана грам-отрицательных бактерий Escherichia coli | 23.6 ± 2.8 | 9 |
| 1uyn | баррель | Внешняя мембрана грам-отрицательных бактерий Neisseria meningitidis | 25.3 ± 1.3 | 10 |
| 2wjr | баррель | Внешняя мембрана грам-отрицательных бактерий Escherichia coli | 23.3 ± 1.5 | 9,5 |
Из таблицы видно, что для альфа-спиралей среднее количество трансмембранных остатков равно 21, а для бета-тяжей - около 9 остатков.
Для начала определили филум, к которому принадлежит белок Pesticin receptor FyuA. Он принадлежит филуму Proteobacteria. Далее осуществили поиск BLAST, в качестве исходной последовательности используя мой белок. При этом немного изменили параметры программы: увеличили максимальное количество хитов (max target hits), которые будет искать программа, со стандартных 100 до 1000 и установили e-value равный 1. В поле Organism введите название филума Proteobacteria и выставили галочку напротив "Exclude", тем самым мы запретили BLAST выдавать сходные белки из того же филума. Добавили еще одно поле Organism и аналогичным образом исключили из поля поиска всех эукариот (Eukaryota). В качестве базы данных использовали RefSeq. Далее было отобрано 14 белков вручную, по возможности учитывая таксономию, так как бласт выдает ошибку при открытии страницы с деревом.
Полученная выбора: gomo.fasta
Воспользуемся базой данных OPM для того, чтобы найти в ней информацию о белке Pesticin receptor FyuA. На основании полученных данных была заполнена таблица 2, на рисунке 1 представлена пространственная структура белка.
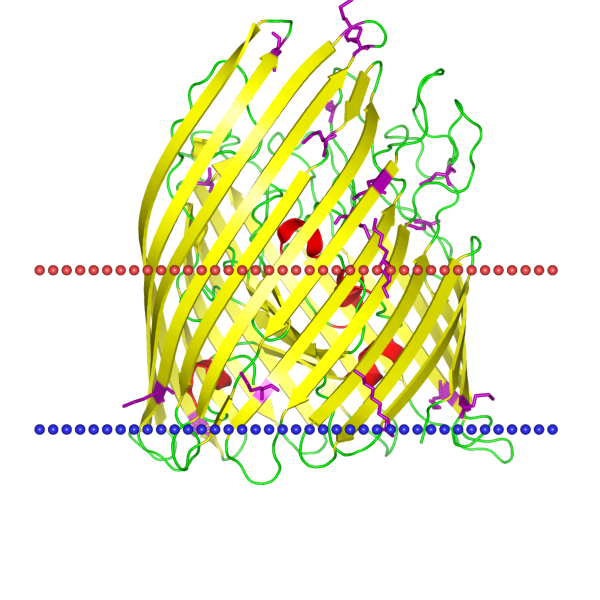
Рис.1. Пространственная структура Pesticin receptor FyuA, полученная с сайта базы данных OPM.
Таблица 2. Описание структуры трансмембранного белка Pesticin receptor FyuA
| PDB ID | Организм | Тип мембраны | TC-код | Угол наклона спиралей (бета-тяжей) к нормали | Количество трансмембранных спиралей (бета-тяжей в бочонке) |
| 4EPA | Yersinia pestis | Внешняя | 1.B.14 | 3 ± 0° | 22 |
Описание каждого из полей TC-кода для белка 4EPA:
- 1 - каналы/поры
- 1.B - бета-баррельные каналы
- 1.В.14 - семейство белков Outer Membrane Receptor, расположенных на внешней мембране грам-отрицательных бактерий. Они воспринимают сигналы из внешней среды и переносят их между двумя слоями бислоя к цитоплазме.
Для начала построили множественное выравнивание отобранных гомологов с помощью программы Muscle. Загрузили множественное выравнивание в программу JalView. Далее добавили к выравниванию дополнительную аннотацию "TM_REAL", указывающую на положения трансмембранных спиралей. Переместили исходный белок, для которого есть структура, в верхнюю строку выравнивания. Прикрепили к нему структуру 4EPA, единственную цепь А. Используя появившуюся связь между последовательностью и структурой, пометили участки выравнивания, отвечающие трансмембранным спиралям в белке со структурой в строке-аннотации буквой "М". Выбрали цветовую схему, позволяющую визуально различать гидрофобные и гидрофильные остатки. Затем установили галочку на "By Conservation" (теперь интенсивность цвета зависит от того, насколько позиция консервативна). Установили белок так, чтобы его часть, ориентированная в n-сторону мембраны(внутреннюю) оказалась сверху, а ориентированная в p-сторону(внешнюю) - снизу. Сохранили полученное изображение структуры белка (рис. 2).
Рис.2. Пространственная структура Pesticin receptor FyuA(4EPA), окрашенная по гидрофобности с уровнем консервативности 18%. Более гидрофобные участки белка окрашены синим, более гидрофильные - красным.
Далее выберем любой из гомолоов, например, белок YP_004113197.1 и получим для него предсказания TMHMM. Но программа не нашла трансмембранных участков для этого белка (рис.4). Тогда то же самое проделаем для оставшихся 13-и гомологов, но результат оказался таим же, т.е. программа не выдает предсказанные участки. Это вызывает подозрения, поэтому помещаем последовательность исследуемого белка и видим, что и для него не находятся трансмембранные участки (рис.5). Если посмотреть на выравнивание (рис.6), то можно заметить, что в трансмембранных участках помимо гидрофобных, достаточно много гидрофильных аминокислот. Возможно это сбивает столку программу и затрудняет ее работу.
Рис.4.Результат предсказания TMHMM для YP_004113197.1
Рис.5. Результат предсказания TMHMM для Pesticin receptor FyuA(4EPA)
Рис.6. Выравнивание выборки гомологов белка Pesticin receptor FyuA(4EPA). Раскраска по гидрофобности с уровнем консервативности 18%. Показана аннотация TM_REAL, буква М индексируют трансмембранные участки.
Итоговый файл c выравниванием формата JAR доступен по ссылке:выравнивание.
Большинство участков, встречающихся в трансмембранных бета-тяжах, не особо консервативны, но встречаются и очень консервативные фрагменты.Ниже в таблице 3 представлены частоты встречаемости различных аминокислот в трансмембранных тяжах белка .
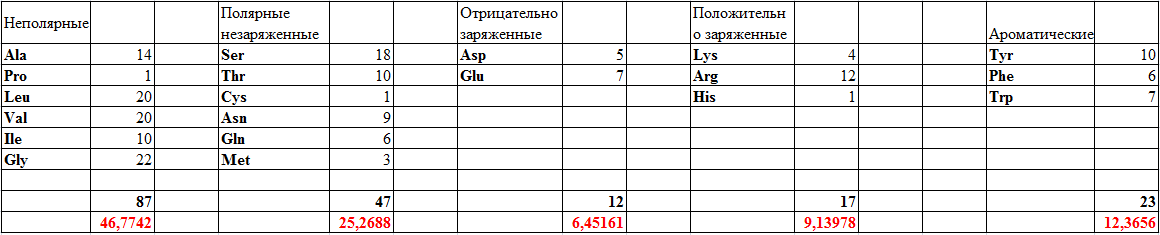
Таблица 3. Частоты встречаемости различных аминокислот в трансмембранных спиралях белка Pesticin receptor FyuA(4EPA).
Из приведённой таблицы отчётливо видно, что в трансмембранных участках преимущественно расположены неполярные аминокислоты с гидрофобными радикалами(46,8%).
Концевые участки более консервативны, чем трансмембранные. Это может быть связано с какой-то дополнительной функцией белка.
Дополнительные задания:
- Средний размер дыни 15 см, размер эукариотической клетки - 10-50 мкм, возьмем в среднем 30 . Толщина мембраны - 7-8 нм. Таким образом, для дыни: (8*10-9 * 15*10-2)/30*10-6= 4*10-5, т.е. 40 мкм. Это значит, мембрану можно сравнить с одним поверхностным слоем дыни толщиной в одну клетку.
- Правило positive inside для трансмембранных белков состоит в том, что часть белка, расположенная с n-стороны от мембраны(внутренняя), содержит больше положительно заряженных остатков, т.е. лизина, аргинина и гистидина.
Рассмотрим остатки лизина, аргинина и гистидина в трансмембранных участках данного белка с помощью средств Jmоl и определим с какой стороны мембраны число положительно заряженных остатков больше (рис.3).
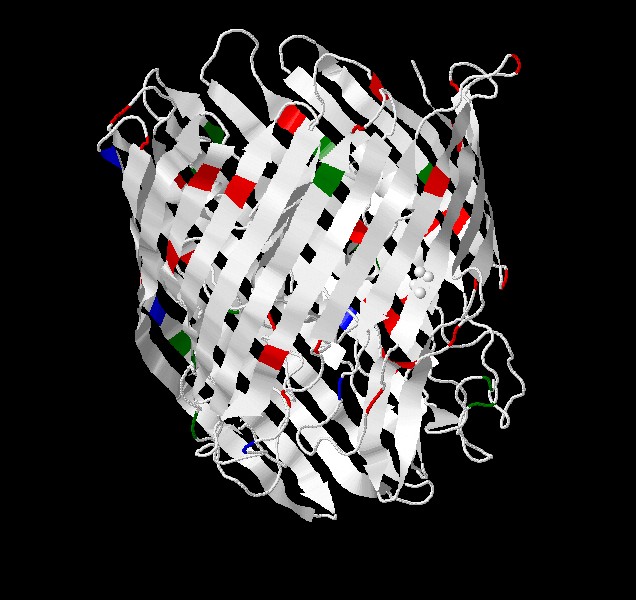
Рис.3. Трёхмерной структурной белка 4ЕРА. Красным цветом обозначены аргинины, синим- гистидины и зеленым-лизины. Белок расположен так, что его часть, ориентированная в n-сторону мембраны(внутреннюю) оказалась сверху, а ориентированная в p-сторону(внешнюю) - снизу.
Видно, что с n-стороны мембраны положительно заряженных остатков больше, чем с p-стороны.
