По данным, приведенными в PDB файле, цепь А белка 2D8L состоит из 17-и альфа-спиралей и 3-х антипараллельных бета-листов. Для определения вторичной структуры белка 2D8L была использована программа Stride, ее результатом явилась следующая выдача: Stride.html. Сравнение результатов определения элементов вторичной структуры приведены в таблице 1.
Таблица 1. Сравнение результатов определения элементов вторичной структуры
| № элемента вторичной структуры | Результат Stride (номера остатков) | Аннотация в pdb-файле (номера остатков) | ||
| Альфа-спираль | 1 | 13 - 27 | 12 - 28 | |
| 2 | 142 - 159 | 142 - 160 | ||
| Бета-лист | 1 | 139 - 141 | 139 - 141 | |
| 190 - 192 | 190 - 192 |
Таким образом, границы, определенные с помощью Stride и в аннотации PDB-файла схожи. Границы бета-листов полностью совпадают, а границы альфа-спиралей отличаются на 1-2 остатка. Для двух альфа-спиралей с границами 86-94 и 95-104, согласно pdb-файлу, программой Stride было определено, что их части 87-89, 91-95 являются 3^10-спиралями, а часть 96-103 альфа-спиралью.
С помощью программы SheeP построили карту бета-листа цепи А структуры 2D8L. Изображение одной из трех карт показано на рис.1.
Рис.1. Карта бета-листа
На рис.2 изображен бета-лист, показанный на карте. Он состоит из 3-х тяжей. На рис.3. показан хребет, соответствующий первому столбцу на карте, то есть аминокислотами Glu193 и Tyr138.
Рис.2. Бета-лист в структуре 2D8L (выделен желтым)
Рис.3. Бета-лист в структуре 2D8L и Cα-атомы,соответствующие одному из его «хребтов» (показаны в шаровой модели)
Далее с помощью Jmol были найдены остатки листа, обращенные к гидрофобному ядру показаны серым, а в гидрофильную сторону красным.
Рис.4. Бета-лист в структуре 2D8L(остатки листа, , обращенные к гидрофобному ядру показаны серым, а в гидрофильную сторону красным).
Далее для данного бета-листа на основании данных программы Stride была получена карта бета-листа (рис.5) и схема водородных связей (рис.6):2D8L.pdb.map.
Рис.5. Карта бета-листа в структуре 2D8L, построенная на основании результатов Stride.
Рис.6. Схема водородных связей тяжей бета-листа.
Совмещение структур
- Построение совмещения структуры 2D8L и четырех гомологов
- По структуре - str_align.fasta
- По последовательностям - tcoffee_align.fasta
- Поиск структурных гомологов для домена 1dek A: 33-154
- Совмещение структур альфа- и бета- цепочек константного домена T-клеточного рецептора
С помощью сервиса PDBeFold были выбраны 4 структурных гомолога 2D8L: 3QWT, 3REN, 4WU0, 2D5J. Для гомологов и самого белка были загружены выравнивание последовательностей по совмещению структур и само совмещение структур (рис. 7).

Рис.7.Cовмещение структур гомологов 2D8L в JMol, предоставленное сервисом PDBeFold.
Выравнивание по структуре (большие буквы в файле с выравниванием) можно сравнить с выравниванием последовательностей, выполненным, например, в программе JalView (с помощью Tcoffee со стандартными параметрами) (рис.8, 9).
Выравнивания:

Рис.8.Выравнивание по структуре гомологов 2D8L в JalView.

Рис.9. Выравнивание последовательностей гомологов 2D8L в JalView.
Выравнивания по последовательности и структуре не совпадают. Можно заметить, что по структурам консервативный Gly92 выравнивается во всех последовательностях, чего не скажешь о выравнивании TCoffee (Gly84).
С помощью сервиса PDBeFold был осуществлен поиск стрктурных гомологов для домена 1dek A: 33-154, всего получилось 28 находок, однако искомый домен не был найден. Это связано с тем, что что по умолчанию в параметрах PDBeFold необходимо совпадение последовательностей 70%, как от длины находки, так и от длины запроса. Указанный кусочек просто меньше 70% от домена.
Выдача PDBeFold: 1dek.dat
Для совмещения по заданному выравниванию были выбраны структуры константного домена человеческого Т-клеточного рецептора из цепи альфа (1qse region d:118-206) и из цепи бета (2esv region e:119-247). С помощью Sheep были построены карты бета-листов для этих структур. Карты, соответствующие указанным участкам показаны на рис.10 и рис.11.
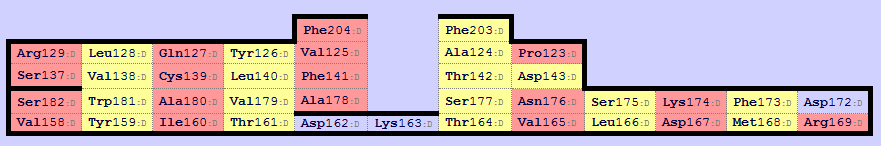
Рис.10. Карта бета-листа в структуре 1qse d:118-206.

Рис.11. Карта бета-листов в структуре 2esv e:119-247.
Бета-листу альфа-цепочки соответствует первый бета-лист бета-цепочки. Консервативный остаток Cys139 в альфа-цепочке и Cys148 в бета-цепочке.
Совмещение цепочек было построено в Pymol с помощью следующих команд:
create 1qse_part, /1QSE//D and resi 118-206
create 2esv_part, /2ESV//E and resi 119-247
create sheet_2esv, 2esv_part and name CA and resi 129-131+147-149+174-176+194-196
create sheet_1qse, 1qse_part and name CA and resi 126-128+138-140+159-161+179-181
pair_fit sheet_1qse, sheet_2esv
Получаем, что RMSD совмещения = 0.533. Совмещение сохранено в файле: pair_fit.pse
Нахождение гидрофобных кластеров
С помощью программы Clud были найдены гидрофобные кластеры в структуре 2D8L. С порогом расстояния 5 и минимальным размером кластера 3 было получено 4 гидрофобных ядра. Одно из них очень крупное и включает все остатки белка (рис.12). Другие гидрофобные ядра принадлежат петле (рис.13), бета-листу (рис.14) и части альфа-спирали (рис.15).
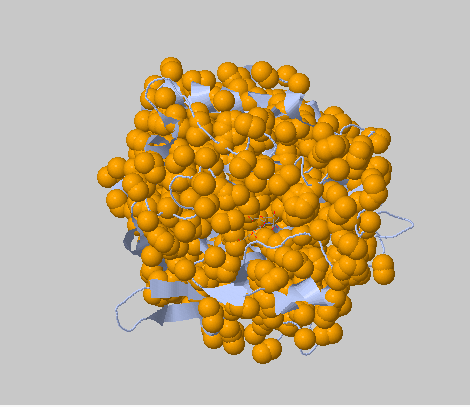
Рис.12.Гидрофобные кластеры в структуре 2D8L

Рис.13.Гидрофобные кластеры в структуре 2D8L
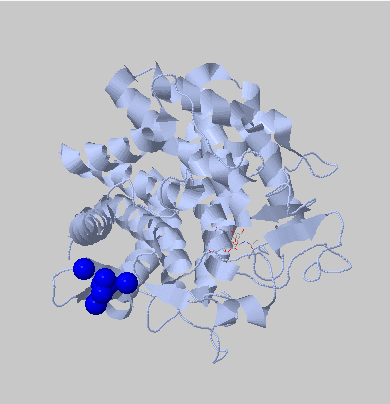
Рис.14.Гидрофобные кластеры в структуре 2D8L
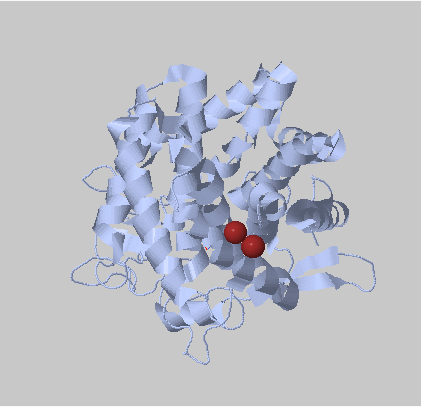
Рис.15.Гидрофобные кластеры в структуре 2D8L
Также были найдены гидрофобные ядра между спиралями структуры 3E7K – домена тетрамеризации TRPM7 катионных каналов, с порогом расстояния 5 и минимальным размером кластера 3, было найдено 10 кластеров. Два наиболее крупные из них расположены между спиралями, что обеспечивает их контакт (рис.16).
Рис.16.Гидрофобные кластеры в структуре 3e7k
Построение поверхности, раскраска участка поверхности: pymol
Для выполнения задания была выбрана структура 1ZAY ДНК-связывающего белка, репрессора транскрипции. Димер был получен с помощью команды:
symexp sym, 1ZAY, 1ZAY, 4
C помощью программы PyMol были созданы изображения, демонстрирующие контакт мономеров белка между собой (рис.17) и контакт белка с ДНК (рис.18, 19).
Файл с построенными поверхностями: surface.pse
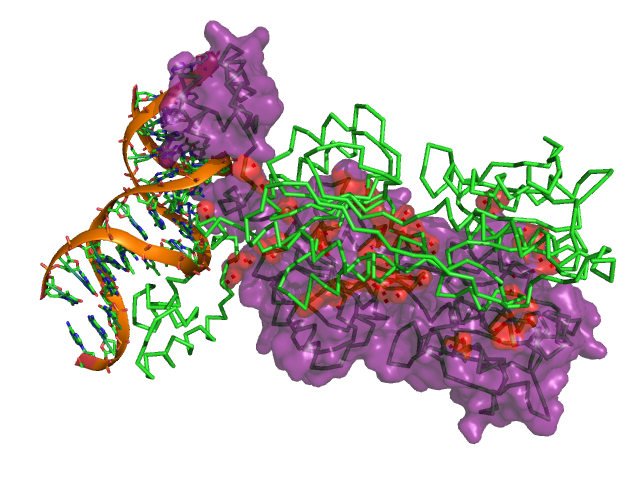
Рис.17. Поверхности контакта мономера белка с симметричным мономером на фоне остовной (ribbon) модели мономера
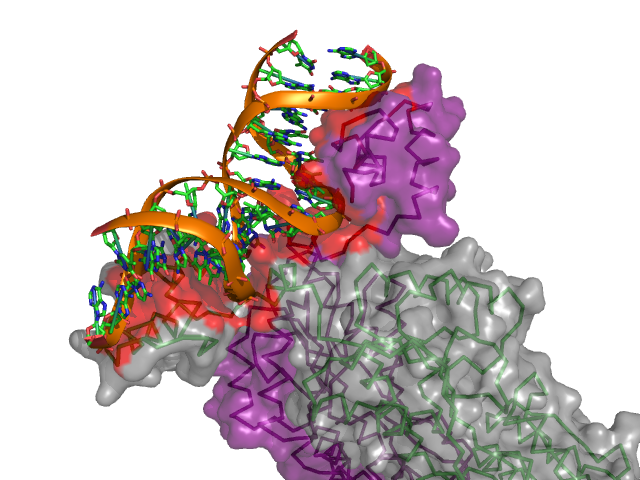
Рис.18.Поверхности контакта димера белков с двойной спиралью ДНК на фоне остовной модели части белка, вовлечённой в контакт
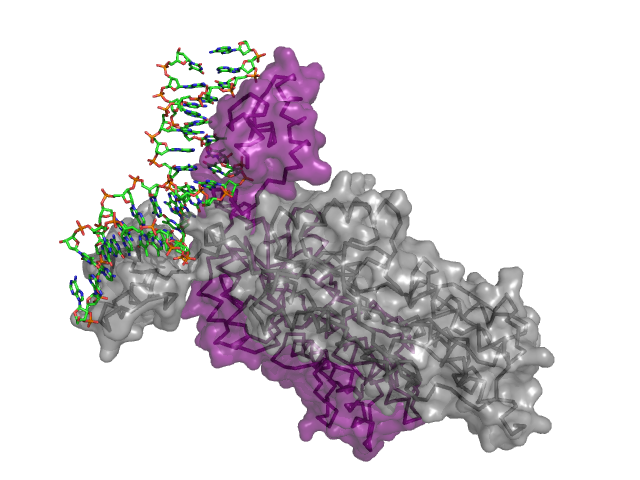
Рис.19.Поверхности контакта ДНК с димером белков на фоне проволочной (sticks) модели двойной спирали
С помощью сервиса Clud в структуре 1ZAY были определены гидрофобные кластеры объёмом не менее 10 атомов на интерфейсе мономеров белка (distance threshold – 5 ангстрем) (рис.20). Таких кластеров было найдено 8.
Рис.20. Гидрофобные кластеры структуры 1ZAY
Сравнение доменов SCOP/SCOPe, ECOD, CATH и Pfam
Структура 2D8L соcтоит из субъединицы A, для которой был произведен поиск доменов в Pfam, SCOPe, ECOD, CATH. Подробная информация о координатах доменов представлена в таблице 2.
Таблица 2. Координаты доменов SCOPe, ECOD, CATH и Pfam для цепи A модели структуры 2DL
| Сервис | ID домена | Координаты домена |
| Pfam | Glyco_hydro_88 | 16-368 |
| SCOPe | d2d8la_ (2d8l A:) | 11-373 |
| CATH | 2d8lA00 | 11-373 |
| ECOD | e2d8lA1 | 11-373 |
Таким образом, границы доменов, найденных с помощью SCOPe, ECOD, CATH совпадают, при этом домен занимает всю расшифрованную часть структуры белка (рис.21). Сервисом Pfam были предсказаны более узкие границы домена (рис.22), это прежде всего связано с тем, что Pfam предсказвает функциональные домены, а SCOPe, ECOD, CATH структурные.
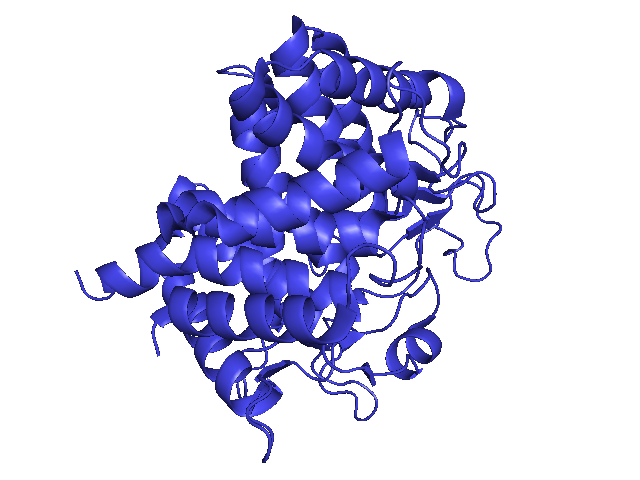
Рис.21. Структура 2D8L, домен, предсказанный SCOPe, ECOD, CATH выделен синим цветом, что совпадает со всей структурой белка

Рис.22. Структура 2D8L, домен, предсказанный Pfam выделен зеленым цветом
Использование сайта PDB
На сайте PDB был осуществлен поиск последовательностей белков, структуры которых получены с помощью электронной микроскопии. Для этого в разделе advanced search были заданы параметры Experimental Method --> ELECTRON MICROSCOPY. Было найдено 965 структур, последовательности которых были сохранены в файле: all_microscopy.fasta.
В PDBeFold осуществили поиск структурных гомологов 2D8L,их оказалось 48. Результаты были отсортированы по RMSD и сохранены в файл : yter.dat
Далее был осуществлен поиск структурных гомологов для структуры 2D8L с помощью программы jFATCAT в PDB. Однако для структуры 2D8L не было обнаружено ни одного гомолога. В таком случае на сайте было предложено выбрать структуру 1NC5, для которой было найдено 207 гомологов с p-value < 0.001. Сервис PDBeFold обнаружил для структуры 1NC5 48 гомологов.
Таким образом, среди результатов jFATCAT и PDBeFold есть 32 совпадения. Совпадения были посчитаны с помощью средст Exel, основываясь на выдачах программ.
Расчет совпадений: 1nc5.xlsx
Список из PDBeFold: 1nc5.dat