- cat NEIMA.fasta PASMU.fasta ROSDO.fasta POLAQ.fasta YERPE.fasta BRUSU.fasta AROAE.fasta > ~/term4/pr4/prot.fasta
- makeblastdb -dbtype prot -in prot.fasta -out col
- blastp -query CLPX.fasta -db col -evalue 0.0001 -out result.txt
Практикум 4. Ортологи и паралоги
Нахождение гомологов
Для начала нужно было найти гомологи белка CLPX_ECOLI в протеомах выбранных организмов. Сделала я это с помощью команд ниже:
Вот файл с выдачей blast
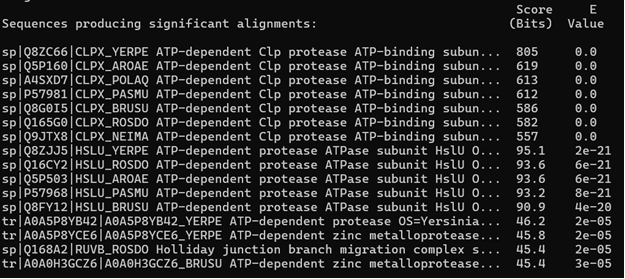
Реконструкция и визуализация
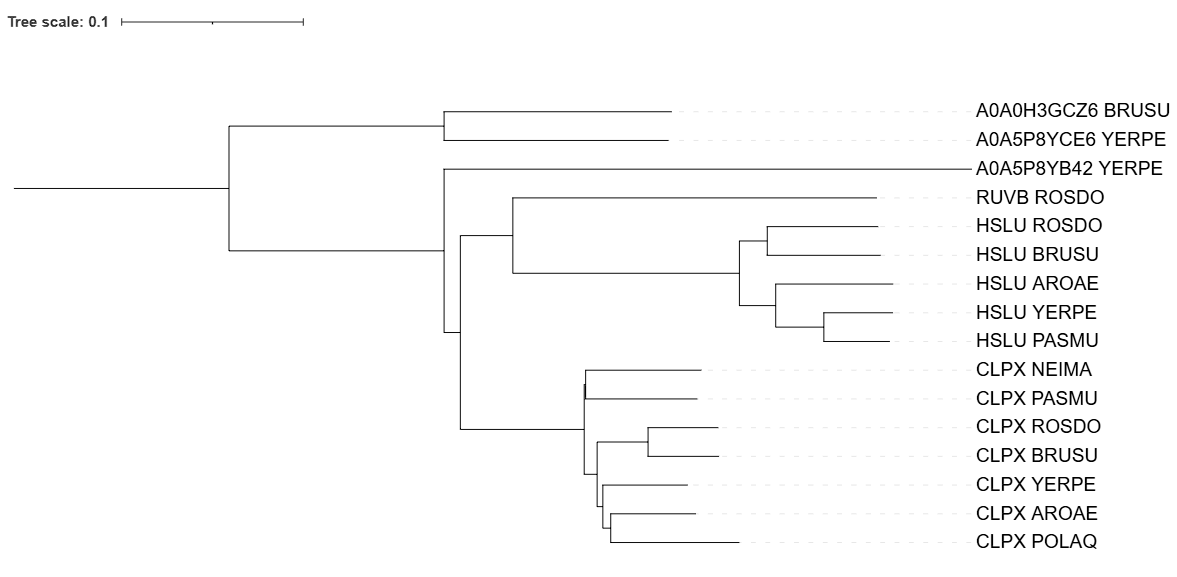
Я изменила названия находок на их мнемоники, затем выравнивал программой muscle. Построила дерево командой fastme с моделью p-distance. Визуализировала дерево с помощью программы ITOL и укоренила в среднюю точку.
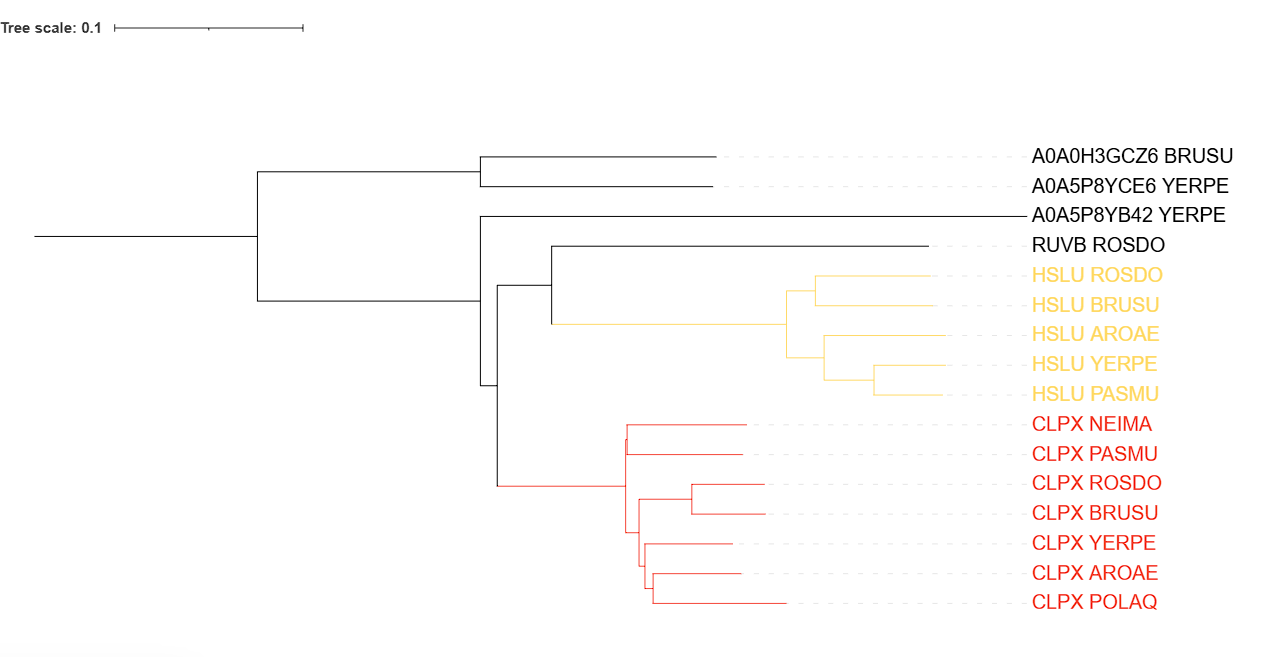
На Рис. 3. показаны с помощью красного и желтого цветов различные ортологические группы (красным - CLPX, желтым - HSLU).
Примеры пар ортологов: HSLU_BRUSU и HSLU_ROSDO, CLPX_NEIMA и CLPX_PALMU, CLPX_YERPE и CLPX_AROAE
Примеры пар парологов: CLPX_ROSDO и HSLU_ROSDO, CLPX_YERPE и HSLU_YERPE, HSLU_PASMU и CLPX_PASMU
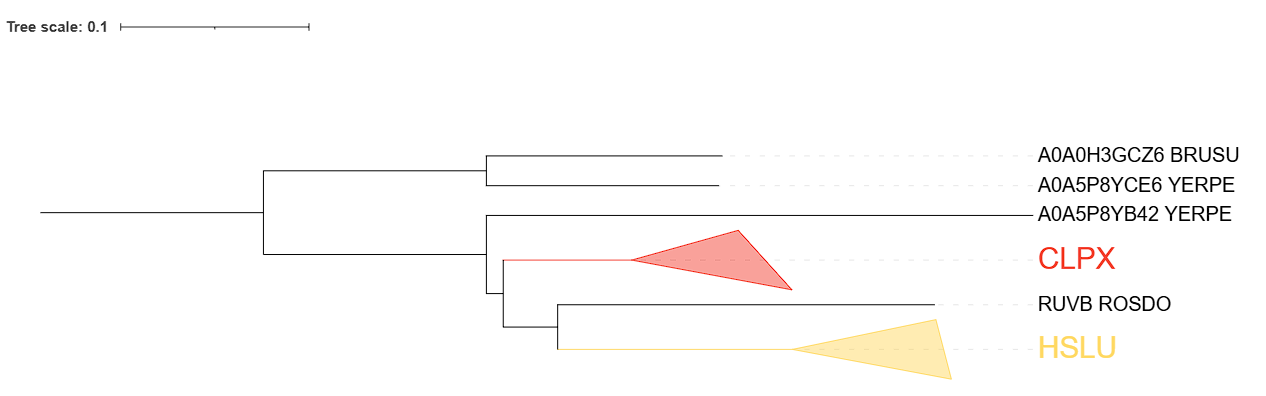
В итоге в у меня получились две большие ортологические группы (CLPX и HSLU).
CLPX: ATP-dependent Clp protease ATP-binding subunit ClpX, АТФ связывающая субъединица АТФ зависимой Clp протеазы.
Состав CLPX: CLPX_NEIMA, CLPX_PASMU, CLPX_ROSDO, CLPX_BRUSU, CLPX_YERPE, CLPX_AROAE, CLPX_POLAQ
Соответствует филогении за исключением одной группы клады: ((NEIMA, PASMU), (YERPE,(AROAE, POLAQ))) реконструирована неверно, должно быть ((YERPE, PASMU), (NEIMA, (AROAE, POLAQ))).
HSLU: ATP-dependent protease ATPase subunit HslU, АТФ зависящая субъединица АТФ зависимой протеазы HslU.
Состав HSLU: HSLU_ROSDO, HSLU_BRUSU, HSLU_AROAE, HSLU_YERPE, HSLU_PASMU
Полностью соответствует филогении.