Практикум 4
Составление списка гомологичных белков, включающих паралоги
Все последовательности были собраны в один файл. Затем последовательности были проиндексированы с помощью программы makeblastdb
После локального запуска программы blatp был получен список гомологов белка CLPX_ECOLI
Параметры запроса:
Порог на E-value = 0.001
Результат работы программы собран в файле
Реконструкция и визуализация
Находки были собраны в один файл. Реконструкция филогенетического дерева проводилась с помощью онлайн-ресурса NGPhylogeny.
Параметры:
"Gamma distributed rates across sites" - No
"Starting tree" - BIONJ
"No refinement"
100 бутстреп реплик
Newick - формула
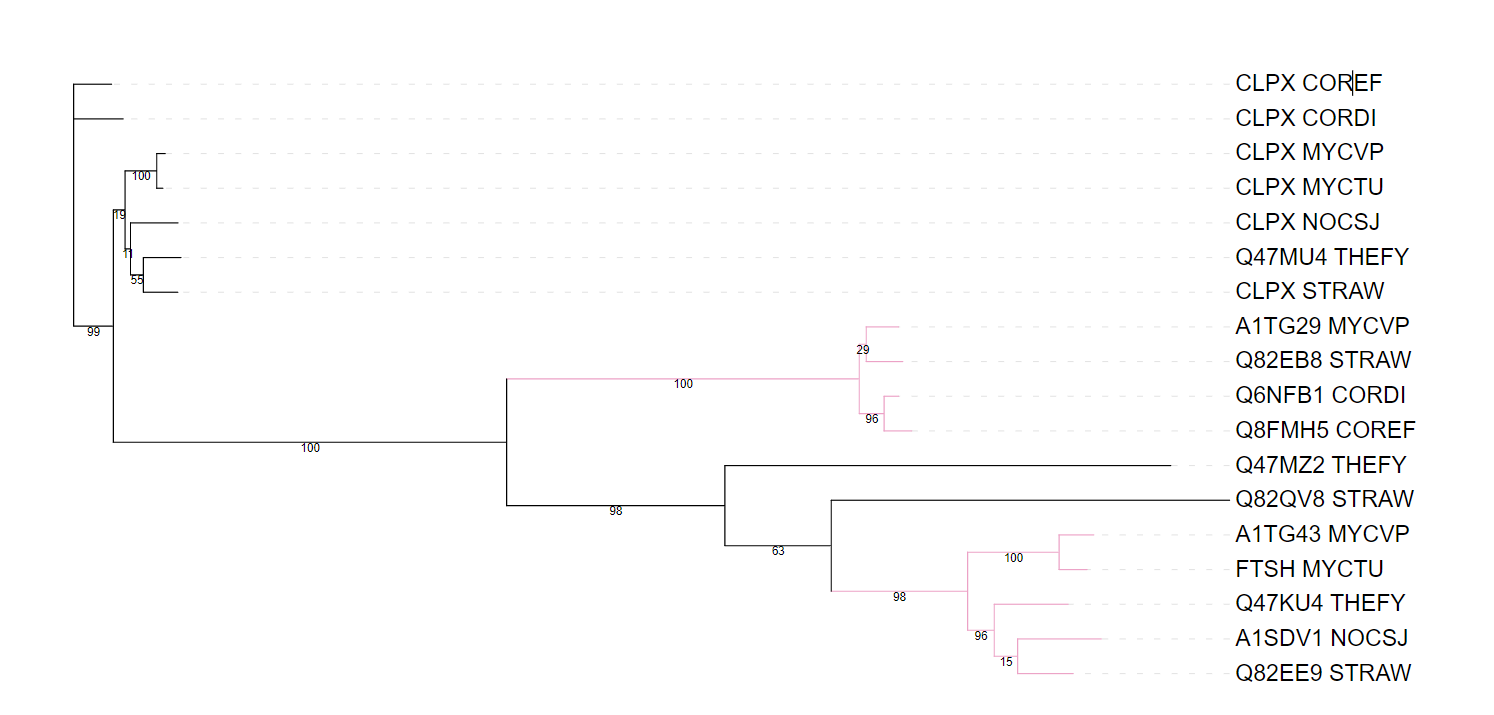
Ортологи:
A1TG29 MYCVP и Q82EB8 STRAW
Q6NFB1 CORDI и Q8FMH5 COREF
A1TG43 MYCVP и FTSH MYCTU
A1SDV1 NOCSJ и Q82EE9 STRAW
Паралоги:
CLPX MYCVP и A1TG29 MYCVP
CLPX CORDI и Q6NFB1 CORDI
CLPX STRAW и Q82EE9 STRAW
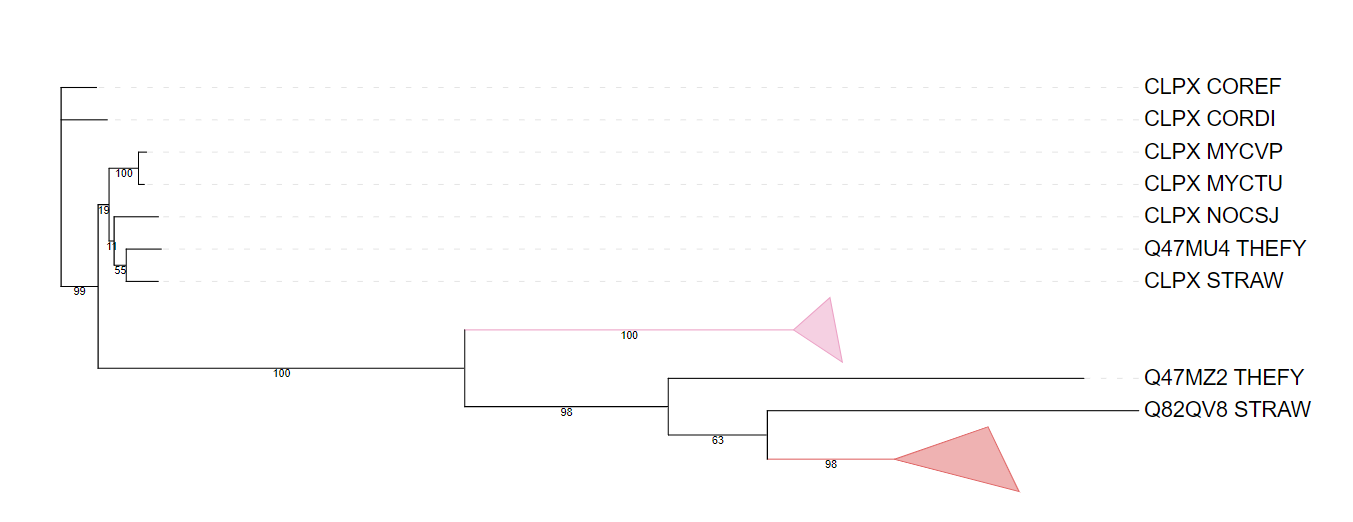
Ссылка на страницу с эталонным деревом
Для группы АТФ-зависимых Clp протеаз эталонному дереву соответствует ветвь {COREF, CORDI}. Также в розовой группе содержатся ветвь, не соответствующая эталонному дереву:{MYCVP,STRAW}. Для группы АТФ-зависимых цинковых металлопротеаз ветвь {MYCVP,MYCTU} и {THEFY,NOCSJ,STRAW} соответствуют эталонному дереву. В целом, можно сказать, что красная ветвь достаточно хорошо соответствует филогении.