Практикум 10. Сравнить состав систем рестрикции модификации, закодированных в двух штаммах одного видаЗадание 1.Этап 1. Название вида бактерии - Roseburia hominis GenBank AC - CP003040.1 Надо посчитать ожидаемое количество и контраст всех сайтов транскрипции из списка в геноме моей бактерии. Последовательность генома лежит в папке chr_fasta. Я загрузила CP003040.1 и лист с сайтами и получила выход - tmpAnwGLk.tsv (первый столбец - сайты, наблюдаемое число, ожидание Карлинга, соотношение Карлинга, общее число) смотрим контараст по ratio - отбираем те, что меньше 0,78, тогда получается 18 сайтов(расставила по увеличению соотношения и покрасила) сами сайты - new_sites.list таблица - input_file Этап 2. Последовательности контигов - Roseburia_hominis.fasta Делаем тоже самое что и раньше, выход - tmpaE_4zx.tsv; меньше 0,78 - 37 штук; отобранные сайты - new_sites_contig.list; таблица - input_file_contig.xlsx Этап 3. Количество сайтов, найденных в геноме - 4, количество сайтов, найденных в контигах - 23, и там и там - 14. Получается, что больше всего сайтов Р-М было найдено в бактерии Roseburia hominis. Задание 2.Найдите последовательности Шайн – Дальгарно в геноме бактерии Alicycliphilus denitrificans K601. На сайте NCBI сборка этой бактерии - ASM20464v1, а именно бактерия Alicycliphilus denitrificans K601 (b-proteobacteria) ссылка на информацию скачала файл с хромосомой - sequence.fasta особенности - CDS - sequence.txt запустила скрипт и получила файл с координатами кодирующих последовательностей - CDSs.xls. Всего найдено 4607 Отобрала около 200 последовательностей, у которых длина превышает 500.Эти последовательности будут использованы для построения мотива - CDSs_top.xls Параметры MEME: длина мотива - 6-10 н, поиск только по данной цепи(даны кодирующие последовательности), количество ожидаемых мотивов - 3. Получены 3 мотива MEME:
Мотив по MEMEПолучены 3 мотива MAST:
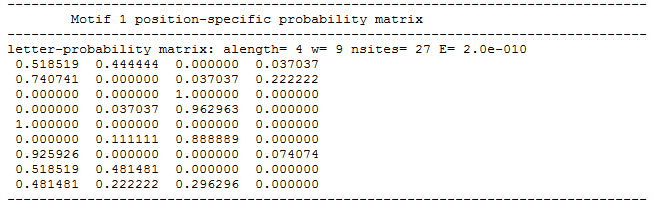
Мотив по MASTПервый мотив и есть последовательность Ш-Д - motif_1_meme.png и выдача с сайта При поиске с параметром 1 мотив мы получили позиционную матрицу весов(PWM) для первого мотива - Позиционнаяа матрица весовДалее провели поиск по всем генам с помощью FIMO используя полученную матрицу весов и расширив границы поиска с -20 до 1 до начала кодирующей последовательности.Порог e-value - 0.01. Найдено генов : Результаты представлены здесь и в этом файле Задание 3.Определите сайты связывания данного транскрипционного фактора в данном участке хромосомы человекаФайл с ридами - chipseq_chunk19.fastq. С помощью программы FastQC я сделала контроль качества прочтений. Командой "fastqc chipseq_chunk19.fasta" были получены файлы chipseq_chunk19_fastqc.zip и chipseq_chunk19_fastqc.html
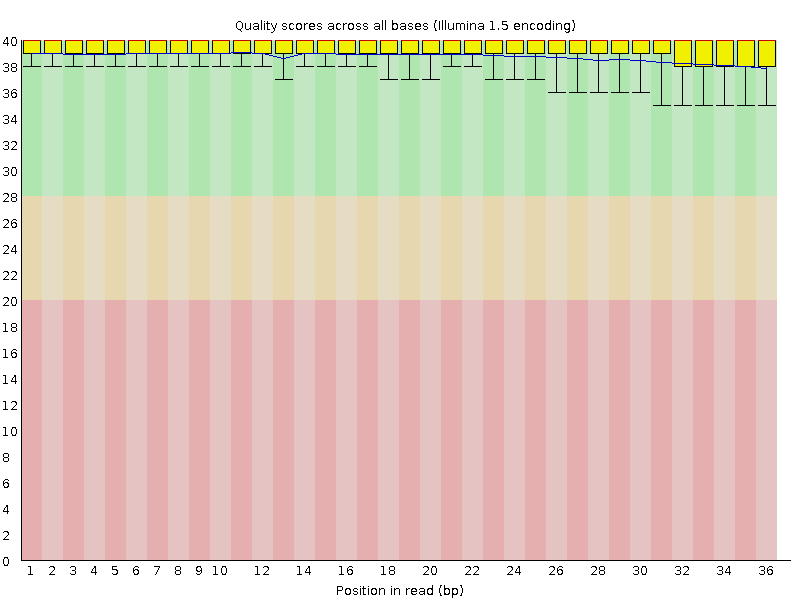
График качества чтенийЧисло чтений:7337; длина ридов: 36 Далее провела картирование чтений на геном hg19 с помощью команды "bwa mem /srv/databases/ngs/hg19/GRCh37.p13.genome.fa chipseq_chunk19.fastq > chipseq_chunk19.sam" Получен файл chipseq_chunk19.sam. Далее при помощи команд проведен анализ данных из этого файла: $ samtools view -bSo chipseq_chunk19.bam chipseq_chunk19.sam (перевод в бинарный формат) $ samtools sort chipseq_chunk19.bam -T chip_temp -o chipseq_chunk19.sorted.bam (сортировка выравнивания чтений по координате) $ samtools index chipseq_chunk19.sorted.bam (индексирование данных) $ samtools idxstats chipseq_chunk19.sorted.bam > chipseq_chunk19.idxstats (определение числа чтений откартированных на геном) $ samtools view -c chipseq_chunk19.sorted.bam (общее число откартировавшихся чтений) Откартировались все риды - 7337. Далее искали пики программой MACS через команду "macs2 callpeak -t chipseq_chunk19.sorted.bam". Но пиков оказалось мало, поэтому использовались другие параметры "macs2 callpeak -t chipseq_chunk19.sorted.bam --nomodel -nchunk19" Получены файлы chunk19_peaks.narrowPeak, chunk19_peaks.xls и chunk19_summits.bed Найдено 7 пиков. "track type=narrowPeak visibility=3 db=hg19 name="my_peaks" description="Peaks from chunk X" browser position chr8:37500000-38300000" - фраза добавлена в файл chunk19_peaks.narrowPeak, что бы провести дальнейшие манипуляции.
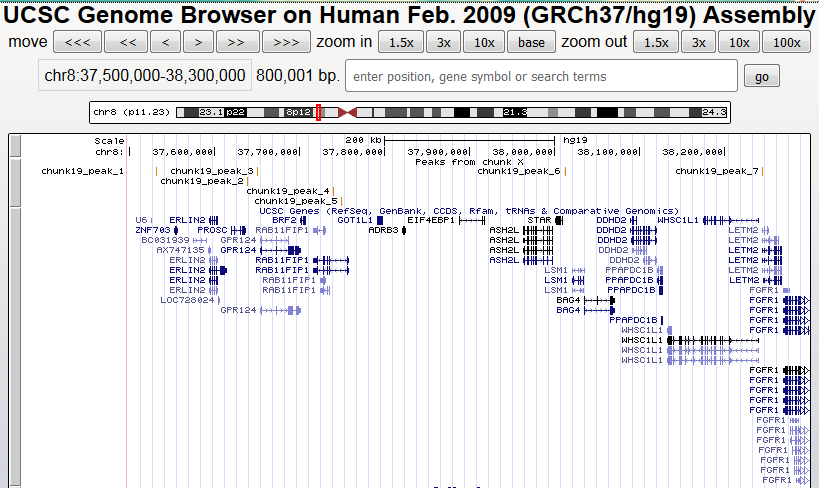
Визуализация полученных пиков с помощью UCSCGenomeBrowser
Вершины пиковИнформация по нескольким пикам представлена в таблице
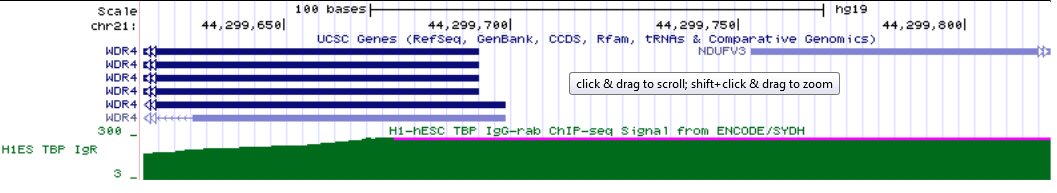
Чем меньше p-value пика, тем он достовернее. Более достоверным пикам соответствует большее значение –log10p-value. В таблице более достоверным пиком является пик 2. Пик 1 не перекрывается с генами. Рядом с пиками 2-5 расположены гены ERLIN2(кодирует член семейства SPFH домена липидных белков), BRF2(кодирует одну из субьъединиц ДНК полимеразы 3).Рядом с пиком 7 расположен ген WHSC1L1(связан с синдромом Вольфа-Хирхшорна). Задание 4.В геноме человека найдите три гена, транскрипция которых инициируется с помощью TATA-бокс связывающего белка, и один - без сигнала TATA-бокса в промоторной областиTATA-бокс связывающий фактор TBP - архейный и эукариотический белок, узнающий восьминуклеотидный сигнал в ДНК с консенсусом TATA(А/Т)A(A/T)(A/G). Не все промоторы имеют сигнал TATA-box. Ген WDR4, chr21:44,263,190-44,299,693; длина 36,504 п.о., на - цепи, 12 экзонов.
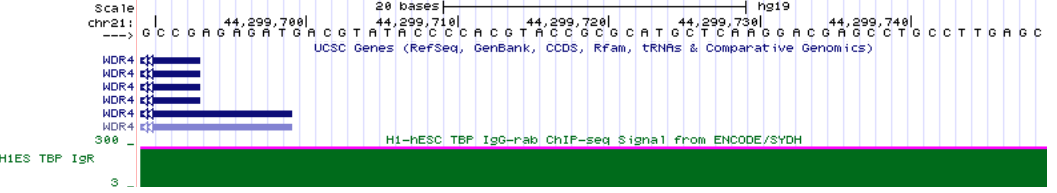
Ген WDR4
Ген WDR4Ген ADAMTS1, hg19 chr21:28,208,606-28,217,728; длина 9,123 п.о., на - цепи, 9 экзонов.
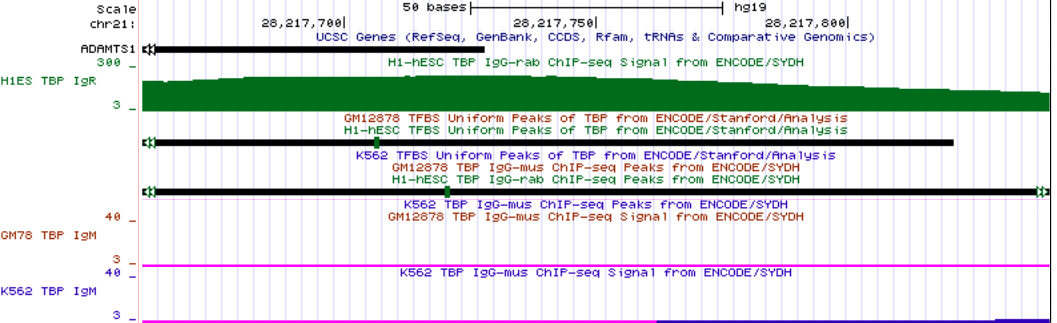
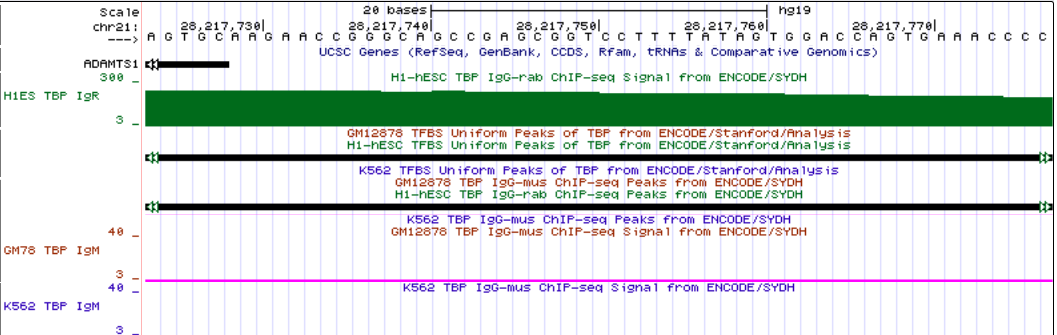
Ген ADAMTS1
Ген ADAMTS1Ген APP, hg19 chr21:27,252,861-27,543,446; длина 290,586 п.о., на - цепи, 16 экзонов.
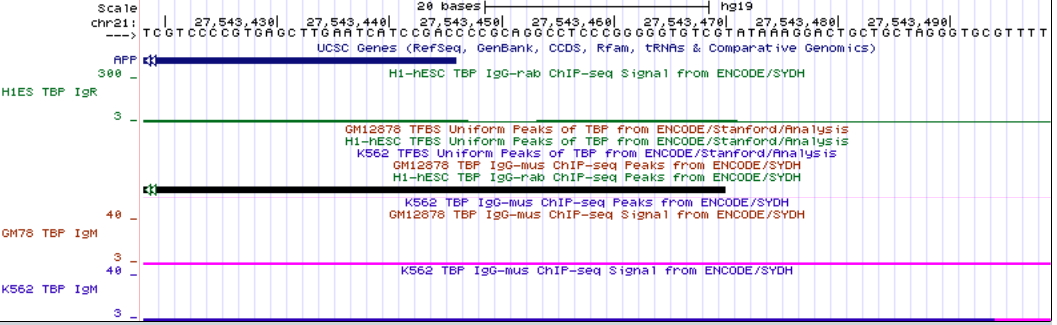
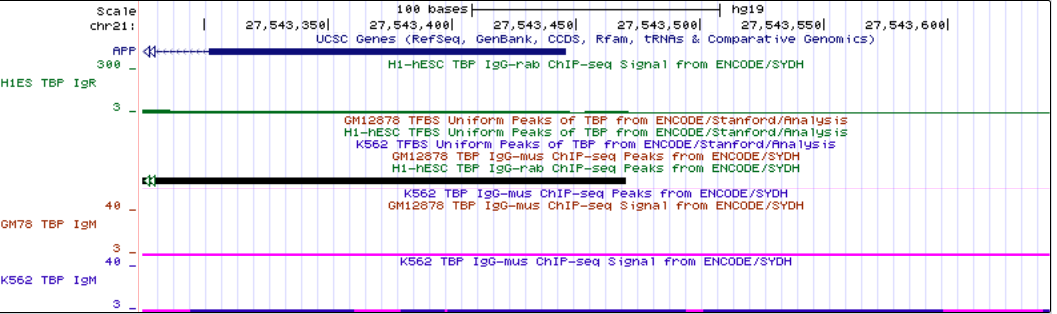
Ген APP
Ген APPБелков без сигнала TATA-бокса в промоторной области: ген SCAF4, hg19 chr21:33,043,313-33,104,431; длина 61,119 п.о.,на - цепи, 20 экзонов.
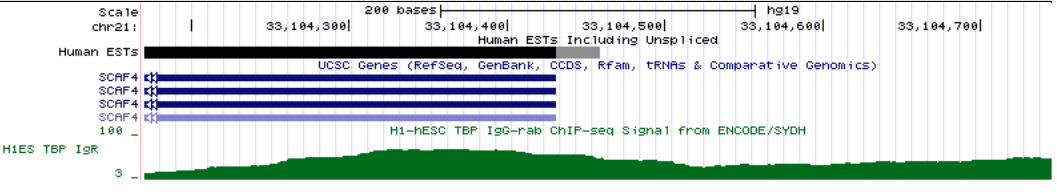
Ген без сигнала TATA-бокса
Ген без сигнала TATA-бокса |