Гомологи белка
В практикуме 7 был взят белок обратная гираза, вызывающий положительное суперскручивание замкнутой кольцевой ДНК.
Для поиска была вставлена последовательность в fasta-формате
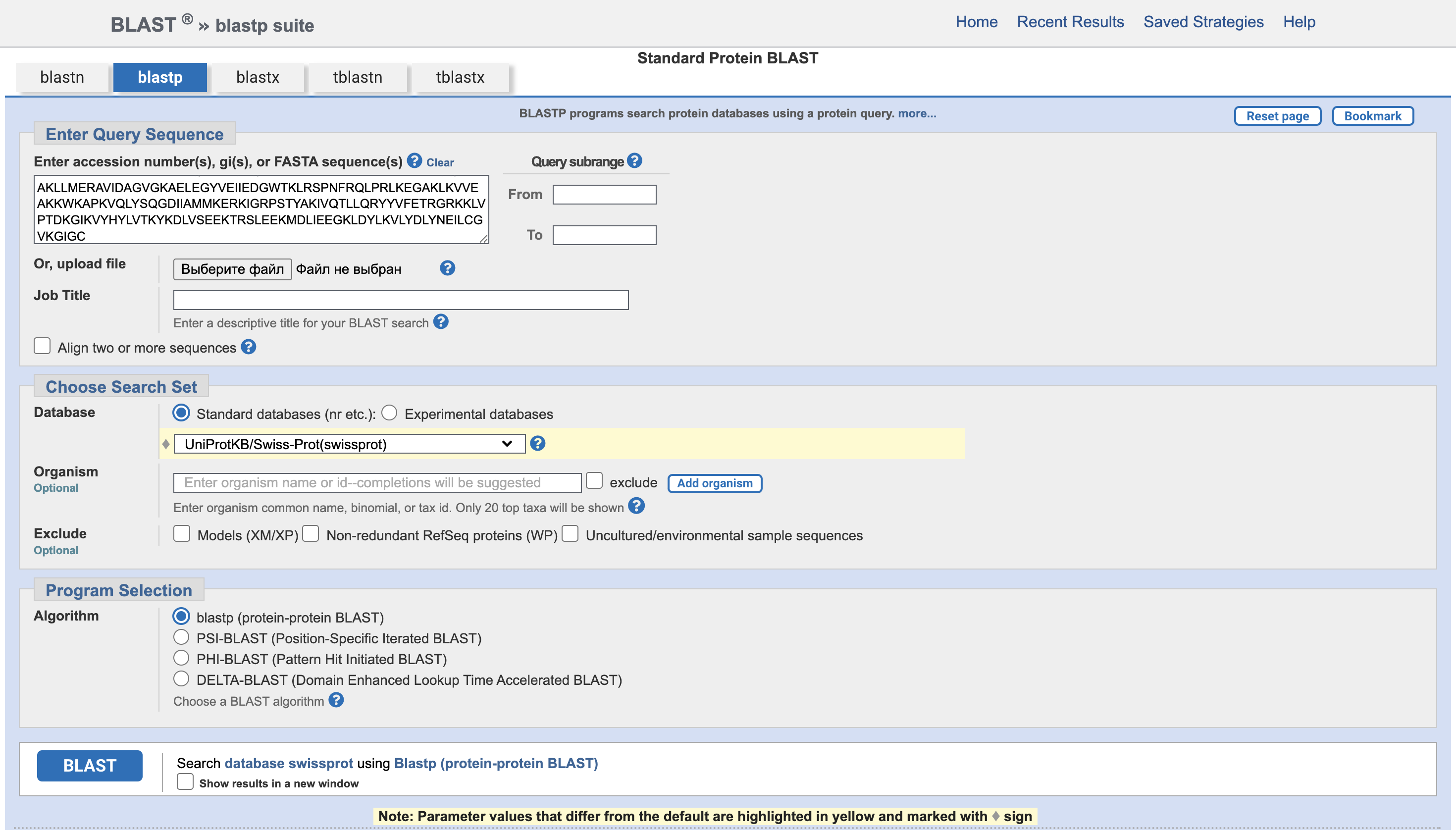
Текстовая выдача Protein BLAST
Для выравнивания были взяты белки:
RecName: Full=Reverse gyrase; Contains: RecName: Full=Pko r-Gyr intein [Thermococcus kodakarensis KOD1]
RecName: Full=Reverse gyrase; Contains: RecName: Full=Pho r-Gyr intein [Pyrococcus horikoshii OT3]
RecName: Full=Reverse gyrase [Pyrococcus furiosus DSM 3638]
RecName: Full=Reverse gyrase [Pyrococcus abyssi GE5]
RecName: Full=Reverse gyrase; Contains: RecName: Full=Mja r-Gyr intein [Methanocaldococcus jannaschii DSM 2661]
RecName: Full=Reverse gyrase 1 [Aeropyrum pernix K1]
RecName: Full=Reverse gyrase [Pyrobaculum aerophilum str. IM2]
Выравнивание было сделано в Jalview с помощью Web Service - Alignment - Muscle with defaults
Проект с выравниваниемНа протяжении всей длины последовательностей есть консервативные участки. Можно сделать вывод, что все выбранные белки гомологичны.
Гомологи зрелого вирусного белка
Выбранный полипротеин:
ID POLA_CHPVE
AC P10941
OS Cryphonectria hypovirus 1 (strain EP713) (CHV-1/EP713) (Chestnut blight fungus hypovirulence-associated virus)
Название зрелого белка: p40 protein
Координаты: 249..622
последовательность зрелого белкаBLAST выдал 2 находки (при изменении значения Word size количество не увеличивалось).
Полученные белки: P10941.2; Q9YTU3.1
Текстовая выдача Проект с выравниваниемМожно однозначно сказать, что белки являются гомологами, поскольку консервативные участки присутствуют на протяжении всей последовательности.
Исследование зависимости E-value от объёма банка
Так как для предыдущего белка было найдено всего 2 находки с E-value равным нулю, было принято решение для этого задания взять другой белок (Coat protein из полипротеина POLG_ASGVP). Для него в BLAST было получено 3 гомолога. Для всех трёх при добавлении поиска по таксону Viruses изменилось значение E-value. Например, для последовательности с AC Q6PLS1.1 в общем поиске E-value = 7e-155, а по вирусам - 3е-156.
Теперь, зная формулу E-value = Kmne-λS и учитывая, что m - длину исходной последовательности мы не меняли, как и S - вес, можно посчитать долю n/n0 (отношения размеров баз данных, в числителе размер базы данных Вирусов в Swiss-Prot, в знаменателе размер всей базы данных Swiss-Prot) как долю E/E0. Получаем приближенную оценку для доли записей по Вирусам в Swiss-Prot, она составляет примерно 4,2%.