На данной странице представлен практикум по поиску ортологов и паралогов белка CLPX_ECOLI в протеомах бактерий, выбранных в предыдущем практикуме, и визуализации полученных данных в Mega.
1) Составление списка гомологичных белков:
Для поиска гомологичных белков в протеомах бактерий, выбранных в предыдущем практикуме, с помощью команд:
makeblastdb -in proteoms.fasta -dbtype prot
были собраны в один файл все протеомы выбранных бактерий, после чего он стал источником для составления базы данных blast. Далее по этой базе с помощью команды:
был произведен поиск гомологов белковой последовательности query.fasta программой blastp с максимальным значением Е-value - 0.001. В результате было найдено 27 последовательностей гомологичных белку CLPX_ECOLI(первая колонка - название, вторая - вес в байтах, третья - значение E-value):
- sp|A1SME0|CLPX_NOCSJ ATP-dependent Clp protease ATP-binding subunit ClpX
- tr|Q1AVT0|Q1AVT0_RUBXD ATP-dependent Clp protease ATP-binding subunit ClpX
- sp|Q9CBY6|CLPX_MYCLE ATP-dependent Clp protease ATP-binding subunit ClpX
- sp|A0LSV2|CLPX_ACIC1 ATP-dependent Clp protease ATP-binding subunit ClpX
- sp|A0JXL2|CLPX_ARTS2 ATP-dependent Clp protease ATP-binding subunit ClpX
- tr|Q47MU4|Q47MU4_THEFY ATP-dependent Clp protease ATP-binding subunit ClpX
- sp|Q6AFZ6|CLPX_LEIXX ATP-dependent Clp protease ATP-binding subunit ClpX
- sp|Q8G5R1|CLPX_BIFLO ATP-dependent Clp protease ATP-binding subunit ClpX
- tr|A0K1M3|A0K1M3_ARTS2 ATPase AAA-2 domain protein
- tr|Q1AU05|Q1AU05_RUBXD ATPase AAA-2
- tr|Q8G871|Q8G871_BIFLO Protease
- tr|A0LW31|A0LW31_ACIC1 AAA ATPase, central domain protein
- tr|Q47MZ2|Q47MZ2_THEFY ATPase
- tr|A0LRB8|A0LRB8_ACIC1 ATP-dependent zinc metalloprotease FtsH
- tr|Q1AY82|Q1AY82_RUBXD ATPase AAA-2
- sp|Q8G6B7|RUVB_BIFLO Holliday junction ATP-dependent DNA helicase RuvB
- sp|A0LR74|FTSH_ACIC1 ATP-dependent zinc metalloprotease FtsH
- tr|Q6ACQ0|Q6ACQ0_LEIXX ATP-dependent zinc metalloprotease FtsH
- tr|Q8G3S2|Q8G3S2_BIFLO ATP-dependent zinc metalloprotease FtsH
- sp|A0JXB1|RUVB_ARTS2 Holliday junction ATP-dependent DNA helicase RuvB
- tr|A1SDV1|A1SDV1_NOCSJ ATP-dependent zinc metalloprotease FtsH
- tr|Q47KU4|Q47KU4_THEFY ATP-dependent zinc metalloprotease FtsH
- tr|A0JR82|A0JR82_ARTS2 ATP-dependent zinc metalloprotease FtsH
- tr|A0K236|A0K236_ARTS2 AAA ATPase, central domain protein
- sp|Q1AV13|FTSH_RUBXD ATP-dependent zinc metalloprotease FtsH
- sp|Q9CD58|FTSH_MYCLE ATP-dependent zinc metalloprotease FtsH
- sp|A1SJA7|RUVB_NOCSJ Holliday junction ATP-dependent DNA helicase RuvB
Оригинальный файл выдачи c выравниваниями можно скачать по ссылке.
2) Реконструкция и визуализация:
Последовательности найденных гомологичных белков были скачаны и выровнены в программе Jalview. После чего для них с помощью алгоритма Maximum likelihood в Mega было построено данное дерево:
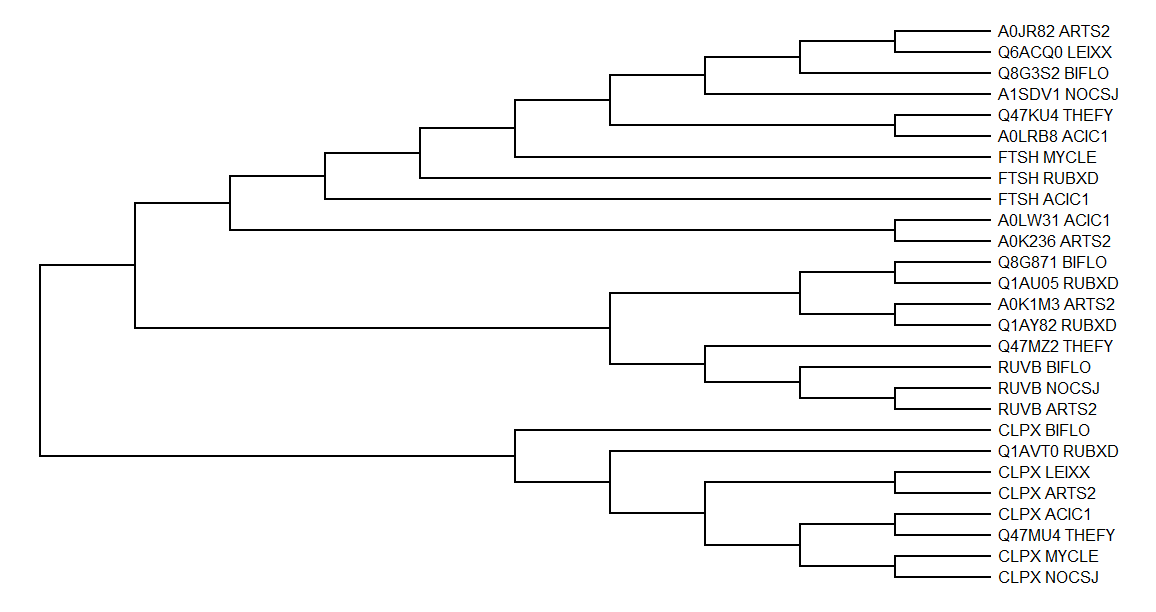
Newick-формула данного дерева.
Для примера на дереве можно выделить:
- 3 пары ортологов: CLPX_NOCSJ и CLPX_MYCLE; CLPX_ACIC1 и CLPX_ARTS2; CLPX_BIFLO и CLPX_MYCLE.
- 3 пары паралогов: CLPX_NOCSJ и RUVB_NOCSJ; FTSH_MYCLE и CLPX_MYCLE; A0K1M3_ARTS2 и AOJR82_ARTS2.
На дереве можно выделить три ортологичные группы:
- ATP-dependent zinc metalloprotease FtsH
- Holliday junction ATP-dependent DNA helicase RuvB
- ATP-dependent Clp protease ATP-binding subunit ClpX
Они представлены на дереве:
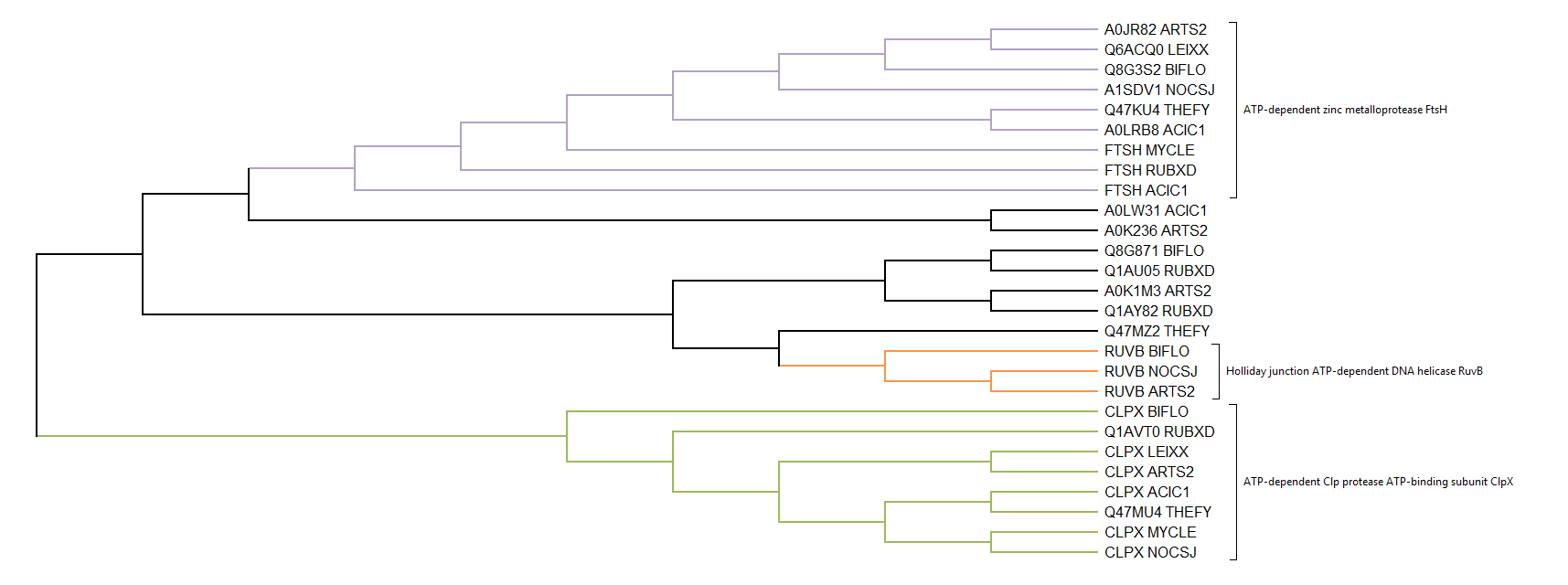
Отнесение к одной группе ортологичных последовательностей
производилось на основании положения на дереве и одинакового названия в базе данных. Поэтому внутри дерева есть несколько последовательностей не относящихся
ни к какой ортологичной группе (они имеют названия ATPase AAA-2/Protease/ATPase AAA-2 domain protein), т.к. эти белки неаннотированы и их функция только предполагается.
В ортологичной группе "ATP-dependent zinc metalloprotease FtsH" встречаются все виды выбранных бактерий, а ACIC1 2 раза, что, вероятно, связано с дупликацией этого гена в
геноме Acidothermus cellulolyticus. Топология дерева ортологов в целом соответствует топологии эталонного дерево - почти все нетривиальные ветви общие.
В группе "Holliday junction ATP-dependent DNA helicase RuvB" всего три последовательности - из NOCSJ, BIFLО и ARTS2, что в целом
соответствует топологии эталонного дерева (все эти последовательности относятся к одной кладе внутри Actinomycetia. Отсутствие в других бактериях вероятно связано
либо с исчезновением данного гена в эволюции, либо неаннотированностью данных последовательностей.
В группе "ATP-dependent Clp protease ATP-binding subunit ClpX"
присутствуют все 8 последовательностей из всех выбранных бактерий и дерево ортологов имеет сходства с эталонным деревом из-за наличия нескольких общих нетривиальных ветвей.
Дерево с схлопнутыми ветвями:
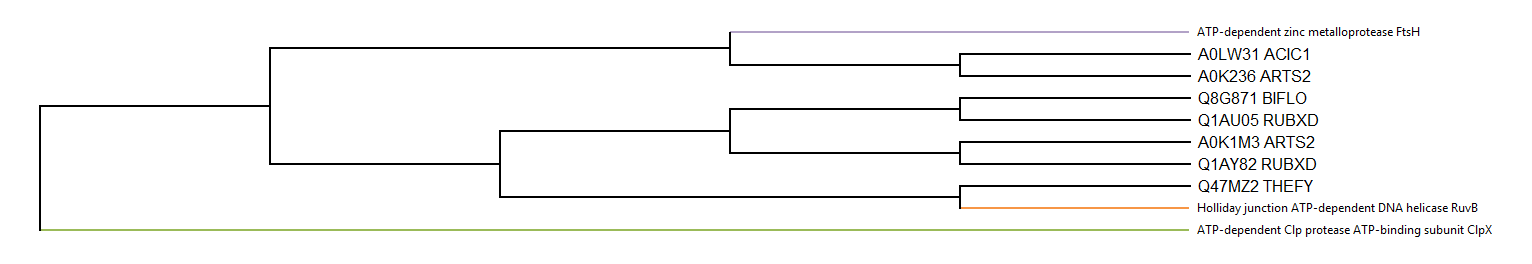
Обсуждение представлено в подписи ко второму дереву.