|
Задание 1. Отобранные бактерии
Скобочная формула дерева ((CLOBA,FINM2),((LACAC,ENTFA),(STAAR,(LISMO,(GEOKA,BACAN))))) |
Нетривиальные ветви 1){CLOBA,FINM2} vs {LACAC,ENTFA,BACAN,GEOKA,LISMO,STAAR} 2){LACAC,ENTFA} vs {CLOBA,FINM2,BACAN,GEOKA,LISMO,STAAR} 3){BACAN,GEOKA} vs {CLOBA,FINM2,LACAC,ENTFA,LISMO,STAAR} 4){BACAN,GEOKA,LISMO} vs {CLOBA,FINM2,LACAC,ENTFA,STAAR} 5){BACAN,GEOKA,LISMO,STAAR} vs {CLOBA,FINM2,LACAC,ENTFA}
|
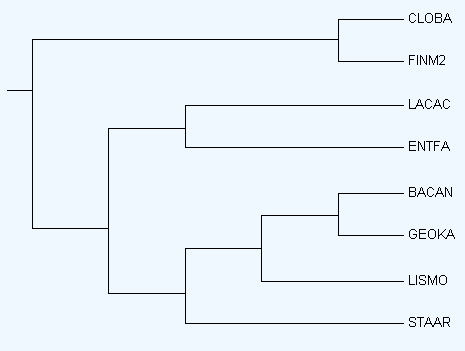
Рис.1. Филогенетическое дерево бактерий |
Таксономия бактерий
| Название | Таксономия |
| Clostridium botulinum | Bacteria; Terrabacteria group; Firmicutes; Clostridia; Clostridiales; Clostridiaceae; Clostridium |
| Finegoldia magna | Bacteria; Terrabacteria group; Firmicutes; Tissierellia; Tissierellales; Peptoniphilaceae; Finegoldia |
| Lactobacillus acidophilus | Bacteria; Terrabacteria group; Firmicutes; Bacilli; Lactobacillales; Lactobacillaceae; Lactobacillus |
| Enterococcus faecalis | Bacteria; Terrabacteria group; Firmicutes; Bacilli; Lactobacillales; Enterococcaceae; Enterococcus |
| Bacillus anthracis | Bacteria; Terrabacteria group; Firmicutes; Bacilli; Bacillales; Bacillaceae; Bacillus; Bacillus cereus group |
| Geobacillus kaustophilus | Bacteria; Terrabacteria group; Firmicutes; Bacilli; Bacillales; Bacillaceae; Geobacillus; Geobacillus thermoleovorans group |
| Listeria monocytogenes serovar 1/2a | Bacteria; Terrabacteria group; Firmicutes; Bacilli; Bacillales; Listeriaceae; Listeria; Listeria monocytogenes |
| Staphylococcus aureus | Bacteria; Terrabacteria group; Firmicutes; Bacilli; Bacillales; Staphylococcaceae; Staphylococcus |
Таким образом, ветвь {LACAC,ENTFA,BACAN,GEOKA,LISMO,STAAR} соответствует таксону Bacilli; ветвь {LACAC,ENTFA} - таксону Lactobacillales, а {BACAN,GEOKA,LISMO,STAAR} - Bacillales; {BACAN,GEOKA} - Bacillaceae.
Реконструкция филогении по белкам семейства RL2
| Выбрав рибосомный белок L2 для реконструкции филогении бактерий, я скачала последовательности белка RL2 для всех моих бактерий и выровнила программой Muscle. Результат - на рис. 2а (внизу). Затем я построила дерево из этого выравнивания методом Neighbor Joining Using % Identity. Результат - на рис. 2б (сбоку). Вот ссылки на файл проекта project.jar, выравнивания muscle.fasta, и дерева tree. |
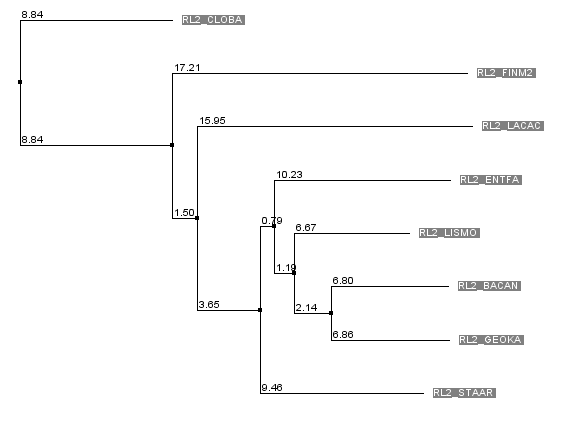
Рис.2б. Филогенетическое дерево бактерий, построенное по белкам семейства RL2
|

Рис.2а. Выравнивание белков
Укоренение в среднюю точку
| Филогенетическое дерево, построенное в предыдущем задании, не укоренено. Чтобы его укоренить, можно использовать несколько методов. Первый - укоренение в среднюю точку. Для его реализации я использовала программу retree пакета PHYLIP. Результат - на рисунке 3. Как мы видим, укоренение произошло в ветвь, отделяющую Clostridium botulinum от остальных бактерий. Ето укоренение вполне можно считать верным, если судить по таксономии, так как CLOBA входит в отдельный класс Clostridia. Показательно и то, что CLOBA отделен от FINM2, хотя на дереве в первом задании они на одной ветви. Но так как FINM2 относится к другому классу - Tissierellia, то верным будет, скорее всего, только что сделанное укоренение в среднюю точку. |
Рис.3. Филогенетическое дерево бактерий, с укоренением в среднюю точку.
|
Укоренение c использованием внешней группы.
| Когда при построении дерева не реконструируются длины ветвей, например при методе максимальной экономии, укоренение в среднюю точку сделать невозможно. В этом случае используются другие методы, например, использование внешней группы. В данном случае в качестве внешней группы была взята RL2_ECOLI. Escherichia coli относится к таксону Proteobacteria, тогда как мои бактерии - к Firmicutes. То есть она заведомо внешняя, но при этом тоже бактерия, то есть не очень сильно отличается от моих. Сначала RL2_ECOLI была выровнена с моими программой Muscle. Затем методом Maximum parsimony было выстроено дерево. Это дерево было вручную укоренено в ветвь RL2_ECOLI. Поддерево с остальными бактериями - конечный результат (рис.4). От предыдущих деревьев оно отличается двумя особенностями. Во первых, ENTFA и LISMO образуют отдельную ветвь. Если верить таксономии, это неправильно. Но так как здесь использовался метод максимальной экономии, подобные ошибки не вызывают удивления. Во вторых, дерево укореняется в FINM2, а не в CLOBA. По таксономии нельзя сказать, верно ли это. |
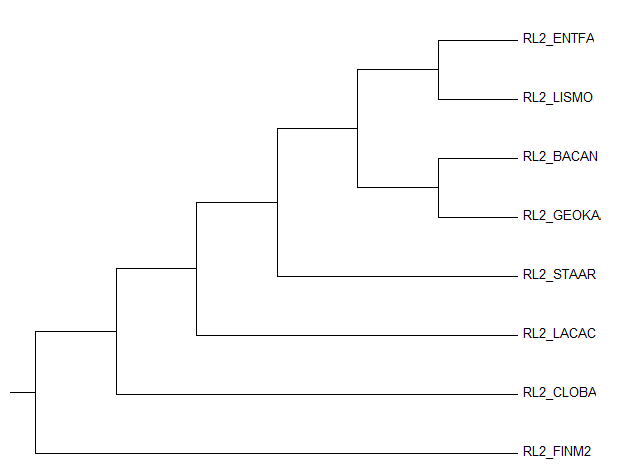
Рис.4. Филогенетическое дерево бактерий, укорененное c использованием внешней группы.
|
Реконструкция филогении по нуклеотидным последовательностям
Чтобы реконструировать филогению, я взяла последовательности РНК малой субъединицы рибосомы (16S rRNA). В базе полных геномов ftp://ftp.ncbi.nlm.nih.gov/genomes/archive/old_refseq/Bacteria/ я нашла геномы указанных выше бактерий. В файлах с расширением .frn лежали последовательности 16S RNA, которые я выровнила программой Muscle. Результат - musclerna.fa. После я построила дерево в MEGA методом Minimum evolution. Результат - на рис. 5.
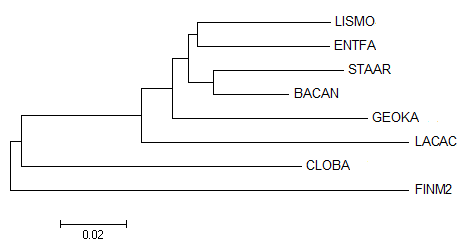
Рис.5. Филогенетическое дерево бактерий, построенное по последовательностям 16S RNA.
Это дерево отличается от предыдущих достаточно сильно: в нем встречены такие ветви как LISMO-ENTFA И STAAR_BACAN, которые не встречены ни в деревьях по белкам, ни в таксономии. Можно сказать, что это дерево недостоверно, хотя в нем есть достоверная ветвь CLOBA-FINM2, которая, правда, обычно расщепляется укоренением.
Поиск паралогов
Для поиска паралогов я взяла белок CLPX_BACSU и с помощью blastp нашла его гомологов в протеомах моих бактерий, взяв evalue ниже 0.001. Ниже использованные мной команды:
wget http://www.uniprot.org/uniprot/P50866.fasta cat /P/y15/term4/Proteomes/CLOBA.fasta >>bacs.fasta cat /P/y15/term4/Proteomes/ENTFA.fasta >>bacs.fasta cat /P/y15/term4/Proteomes/FINM2.fasta >>bacs.fasta cat /P/y15/term4/Proteomes/GEOKA.fasta >>bacs.fasta cat /P/y15/term4/Proteomes/LACAC.fasta >>bacs.fasta cat /P/y15/term4/Proteomes/LISMO.fasta >>bacs.fasta cat /P/y15/term4/Proteomes/STAAR.fasta >>bacs.fasta makeblastdb -in bacs.fasta -dbtype prot blastp -query P50866.fasta -db bacs.fasta -out proteins -evalue 0.001Результат - в файле proteins. Получилось 37 белков - гомологов. Чтобы их скачать, я создала файл text со строками вида uniprot : (имя файла), а после скачала файлы с белками командой:
seqret @text blast.fastaПолученные последовательности я выровнила в Muscle. Результат - muscleblast.fa. Далее я построила дерево методом Maximum likelihood. Оно - на рис. 6a.
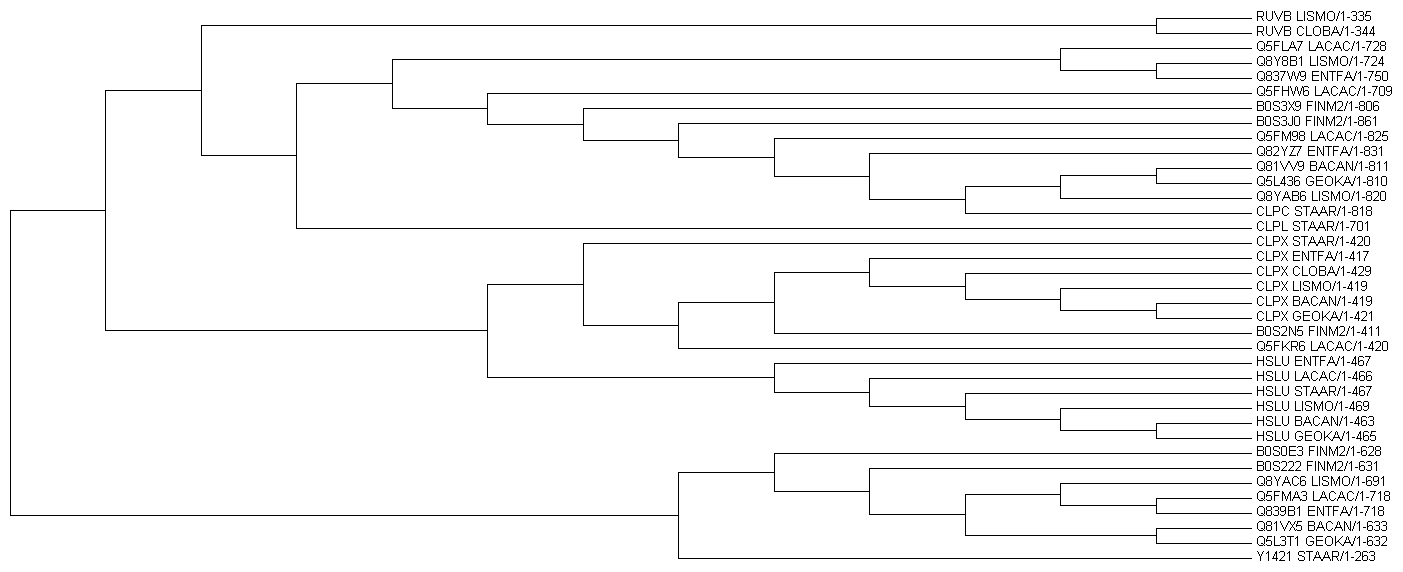
Рис.6a. Дерево белков, гомологичных CLPX_BACSU.
Можно выделить несколько ортологичных групп, например, RUVB, CLPX, HSLU. Видно, что RUVB отделилась в результате дубликации гена раньше, чем произошло разделение CLPX и HSLU. В каждой группе есть паралоги, например RUVB_LISMO, CLPX_LISMO, HSLU_LISMO. Стоит заметить, что в группе RUVB только 2 вида в отличие от остальных, что может свидетельствовать о том, что oбразование видов STAAR, ENTFA, BACAN, GEOKA, FINM2, LACAC произошло уже после отделения RUVB.