Нуклеотидный BLAST
Определение таксономии и функции прочтенной последовательности
На странице Чтение последовательностей по Сэнгеру была проделанна работа с сырыми даннами секвенирования. В результате получилась неплохая нуклеотидная последовательность, однако все равно не понятно к какому организму она принадлежит, и какую функцию она несет (если вообще несет).
Для начала я прогнал по BLASTn (при чем по megablast) первую последовательность (да, я все еще буду называть их 1 и 2. Мне так удобнее). Результат:
Первая последовательность в поиске (нажми на меня)
Glycera capitata isolate A histone H3 (H3) gene, partial cds
Sequence ID: KP113589.1
Length: 314
Score Expect Identities Gaps Strand
575 bits(311) 2e-160 312/313(99%) 0/313(0%) Plus/Plus
Query 18 AGGCCCCCCGAAAGCAGCTCGCCACCAAGGCTGCCCGCAAGAGCGCACCAGCCACCGGCG 77
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Sbjct 2 AGGCCCCCCGAAAGCAGCTCGCCACCAAGGCTGCCCGCAAGAGCGCACCAGCCACCGGCG 61
Query 78 GAGTGAAGAAACCCCATCGTTACAGGCCCGGAACAGTCGCTCTCCGTGAGATCCGTCGTT 137
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Sbjct 62 GAGTGAAGAAACCCCATCGTTACAGGCCCGGAACAGTCGCTCTCCGTGAGATCCGTCGTT 121
Query 138 ACCAGAAGAGCACCGAGCTTCTCATCCGCAAGCTGCCCTTCCAGCGTCTGGTCCGTGAGA 197
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Sbjct 122 ACCAGAAGAGCACCGAGCTTCTCATCCGCAAGCTGCCCTTCCAGCGTCTGGTCCGTGAGA 181
Query 198 TCGCCCAGGACTTCAAGACTGATCTCCGCTTCCAGAGCTCTGCNGTCATGGCCCTTCAGG 257
||||||||||||||||||||||||||||||||||||||||||| ||||||||||||||||
Sbjct 182 TCGCCCAGGACTTCAAGACTGATCTCCGCTTCCAGAGCTCTGCTGTCATGGCCCTTCAGG 241
Query 258 AGGCTAGCGAGGCTTACCTGGTCGGACTCTTCGAGGACACCAACCTGTGCGCCATCCACG 317
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Sbjct 242 AGGCTAGCGAGGCTTACCTGGTCGGACTCTTCGAGGACACCAACCTGTGCGCCATCCACG 301
Query 318 CCAAGCGTGTCAC 330
|||||||||||||
Sbjct 302 CCAAGCGTGTCAC 314
Вторая последовательность в поиске
Glycera capitata isolate B histone H3 (H3) gene, partial cds
Sequence ID: KP113590.1
Length: 311
Score Expect Identities Gaps Strand
571 bits(309) 3e-159 310/311(99%) 0/311(0%) Plus/Plus
Query 20 GCCCCCCGAAAGCAGCTCGCCACCAAGGCTGCCCGCAAGAGCGCACCAGCCACCGGCGGA 79
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Sbjct 1 GCCCCCCGAAAGCAGCTCGCCACCAAGGCTGCCCGCAAGAGCGCACCAGCCACCGGCGGA 60
Query 80 GTGAAGAAACCCCATCGTTACAGGCCCGGAACAGTCGCTCTCCGTGAGATCCGTCGTTAC 139
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Sbjct 61 GTGAAGAAACCCCATCGTTACAGGCCCGGAACAGTCGCTCTCCGTGAGATCCGTCGTTAC 120
Query 140 CAGAAGAGCACCGAGCTTCTCATCCGCAAGCTGCCCTTCCAGCGTCTGGTCCGTGAGATC 199
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Sbjct 121 CAGAAGAGCACCGAGCTTCTCATCCGCAAGCTGCCCTTCCAGCGTCTGGTCCGTGAGATC 180
Query 200 GCCCAGGACTTCAAGACTGATCTCCGCTTCCAGAGCTCTGCNGTCATGGCCCTTCAGGAG 259
||||||||||||||||||||||||||||||||||||||||| ||||||||||||||||||
Sbjct 181 GCCCAGGACTTCAAGACTGATCTCCGCTTCCAGAGCTCTGCTGTCATGGCCCTTCAGGAG 240
Query 260 GCTAGCGAGGCTTACCTGGTCGGACTCTTCGAGGACACCAACCTGTGCGCCATCCACGCC 319
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Sbjct 241 GCTAGCGAGGCTTACCTGGTCGGACTCTTCGAGGACACCAACCTGTGCGCCATCCACGCC 300
Query 320 AAGCGTGTCAC 330
|||||||||||
Sbjct 301 AAGCGTGTCAC 311
Из этого можно сделать вывод, что днк была выделенна из Glycera capitata, изолированный гистон, при чем скорее всего B.
Вообще белки отличаются только на глутамин, но так как в моей последовательности вместо первого триплета cag находится aag, то это либо мутация, либо B. Я склоняюсь ко второму.
При поиске по второй хромотограмме megablast перешол на комплементарную цепь, так что результат остается тем же.
Сравнение трех алгоритмов BLASTn
В BLAST сравнивающем нуклеотиды есть три алгоритма:megablast, discontiguous megablast и blastn. Так чем же они фактически отличаются?
В описании написанно, что они предложенны в порядке убывания скорости и возрастания качества. Чтобы проверить, я запустил три одинаковых запроса, меняя только алгоритм поиска.
Вот параметры поиска
Database Nucleotide collection (nr/nt)
Organism Exclude Annelida (taxid:6340)
Organism Animalia (taxid:33208)
Organism Exclude Bilateria (taxid:33213)
Max target sequences 1000
Expect threshold 0.001
Я довольно долго размышлял, какие параметры лучше выбрать для сравнения, и вот к чему я пришел:
Таблица сравнения:
| Алгоритм | количество найденных последовательностей | Время поиска (секунды) | ID первой последовательности | Процент покрытия первой последовательности | Процент идентичности на месте покрытия | E-value | Количество словДля каждого алгоритма диапазон слов разный, так что этот параметр различается. Однако, я постарался выбрать наиболее близкие по длине. |
|
| megablast | 490 | 13 | 36 | XM_001628717.1 | 97% | 89% | 2e-121 | 16 |
| discontiguous megablast | 529 | 10 | 12 | XM_001628717.1 | 98% | 89% | 4e-128 | 12 |
| blastn | 494 | 10 | 15 | XM_001628717.1 | 98% | 89% | 4e-128 | 15 |
Так же я придумал еще один критерий (по-мне так самый показательный), однако я считаю, что лучше его поместить его в отдельную таблицу. Я скачал полные найденные последовательности, после чего прогнал их через два питоновских скрипта. Если нужно, вот архив.
Результат: у всех общие только 19 находок. Таблица для парных значений:
| blastn | megablast | discontiguous megablast | |
| blastn | - | 19 | 493 |
| megablast | 19 | - | 19 |
| discontiguous megablast | 493 | 19 | - |
Проверка на наличие гомологов
C kodomdo я взял файл X5.fasta. Далее с помощью tblastn (поиск по белку в нуклеотидой базе данных) были найденны предположительные гомологи для трех белков (вот последовательности этих белков)
Heat shock cognate 71 kDa protein (P11142.1)
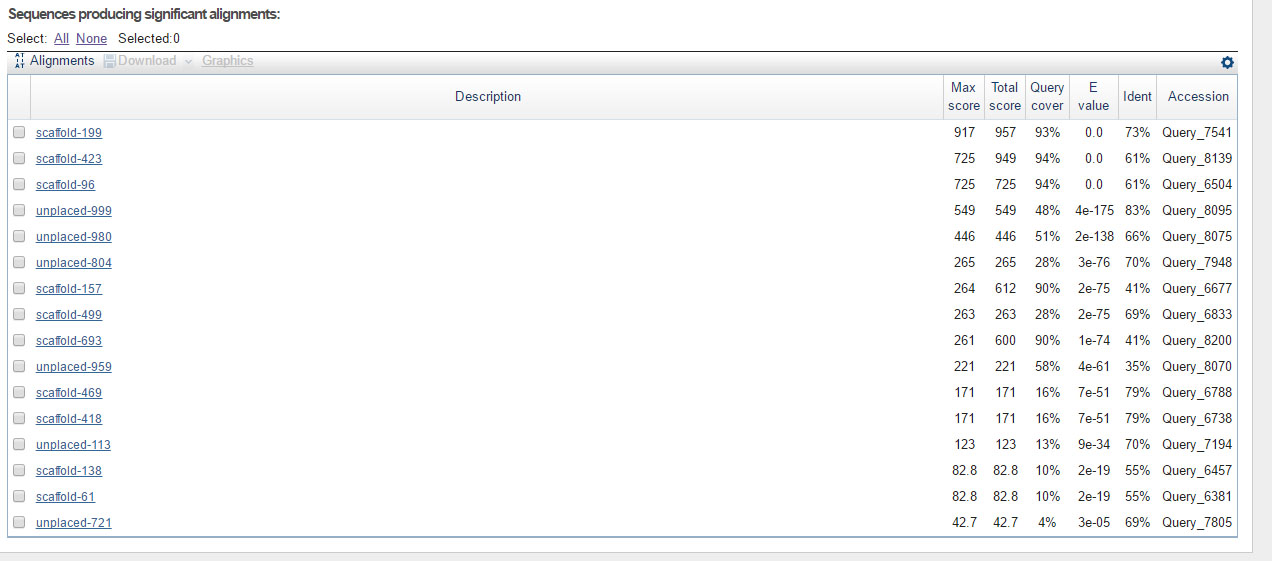
К гомологам я бы отнес первые четыре находки: скэффолды 199,423,96 и unplaced-999.
Citrate synthase (O75390.2)
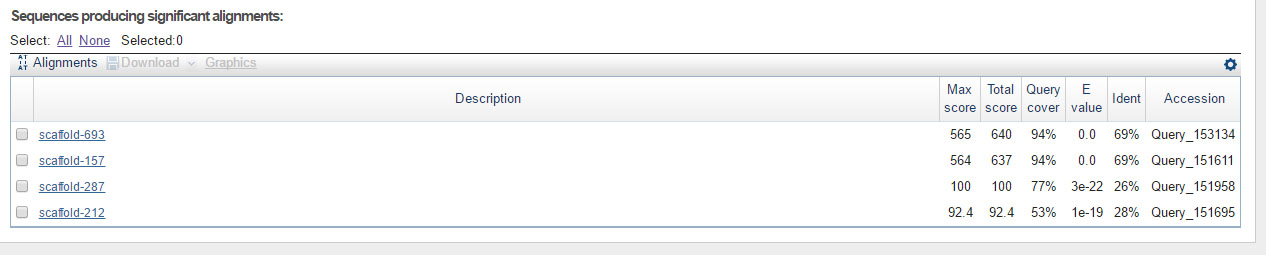
Первые две: скэффолды 693 и 157.
DNA-directed RNA polymerase II subunit RPB1 (P24928.2)
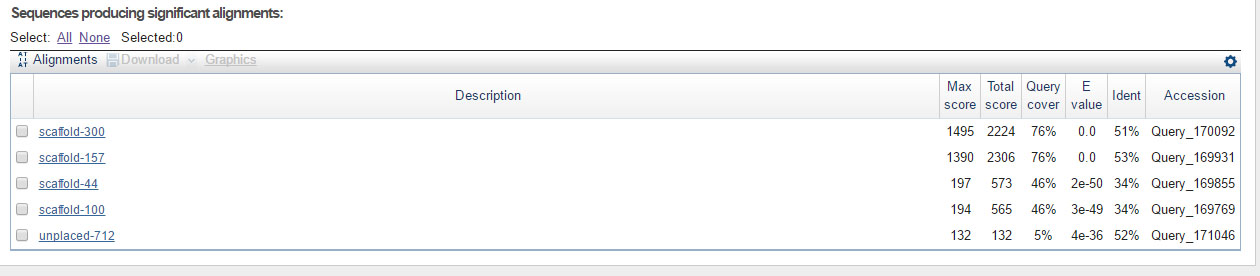
Так же первые две: скэффолды 300 и 157.
Поиск белок-кодирующего гена
Задача: для одного скеффолда X5 найти белок-кодирующий ген, который он несет.
Я взял scaffold-89, и провел его по blastx(нуклеотид-белок). Результат - скорее всего, в нем есть ген, кодирующий белок CLN3.
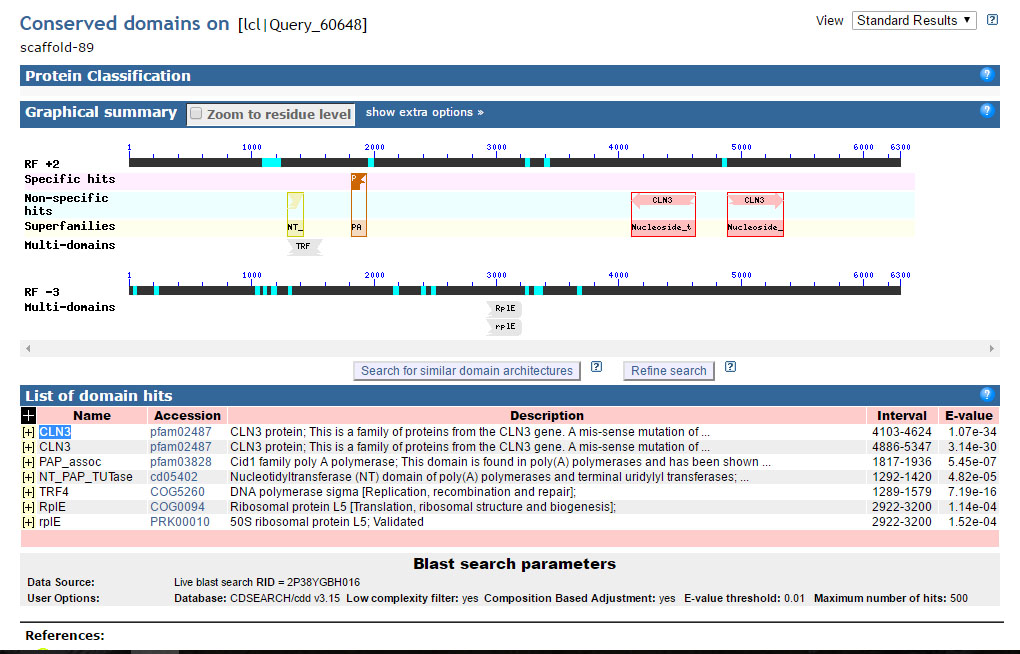
Использовавшиеся команды:
infoseq X5.fasta -only -name -length seqret X5.fasta:scaffold-89 -out scaffold.fasta
| на главную |
© Гавриш Глеб 2016 |