A- и В- формы ДНК. Структура РНК.
Суть работы - сравнительный анализ канонической ДНК и стеблей тРНК.
Задание 1.
C помощью инструментов пакета 3DNA были построены модели структур A-, B- и Z-формы ДНК. Пакет 3DNA - один
из популярных пакетов программ для анализа и простейшего моделирования структур нуклеиновых кислот под операционной системой LINUX.
В программе Рutty, используя протокол ssh, я подсоединилась к серверу kodomo.cmm.msu.ru. В заданной директории term3/block1/pr2 посредством
программы fiber пакета 3DNA
я построила A-, B- и Z-формы дуплекса ДНК, записанные соответственно в файлы gatc-a.pdb, gatc-b.pdb и
gatc-z.pdb. A и B состоят из GATC повторов, Z - только из G:C пар.
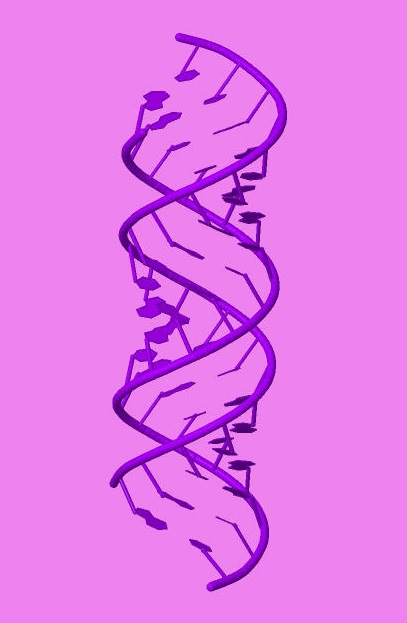
|

|
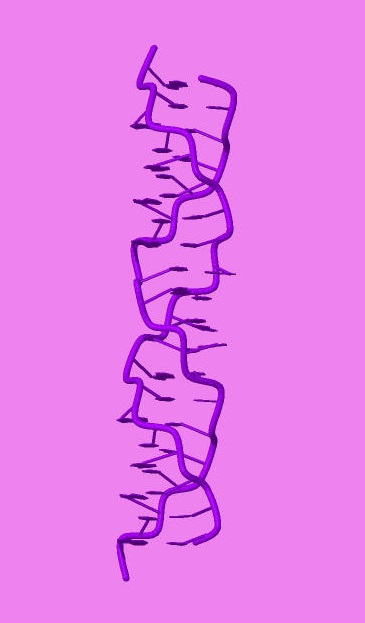
|
A-форма ДНК (Jmol) | B-форма ДНК (Jmol) | Z-форма ДНК (Jmol) |
Задание 2. Упражнение 1.
Анализ структуры дуплексов ДНК в визуализаторе JMol.
Для данного мне основания (цитозина) были определены следующие характеристики:
| Атомы цитозина в трех формах ДНК | |||
|---|---|---|---|
| A-форма | B-форма | Z-форма | |
| В сторону большой бороздки обращены атомы: | C12.N4, C12.C4, C12.C5, C12.C6 | C12.N4, C12.C4, C12.C5, C12.C6 | Не имеет смысла говорить о большой бороздке, так как она слабо выражена в этой форме |
| В сторону малой бороздки обращены атомы: | C12.O2, C12.C2, C12.N1 |
C12.O2, C12.C2, C12.N1 | Малая бороздка узкая и глубокая |
| Остальные атомы основания: | C12.N3 | C12.N3 | - |
В программе MarvinSketch получены изображение основания из 2 ДНК, где красным цветом выделены атомы, смотрящие в сторону большой бороздки, синим - в сторону малой.
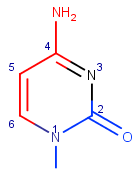
|
|
Азотистое основание цитозин, C12 |
Задание 2. Упражнение 2.
Измерение основных спиральных параметров разных форм ДНК с помощью JMol.
| Таблица спиральных параметров ДНК | ||||
|---|---|---|---|---|
| A-форма | B-форма | Z-форма | ||
| Тип спирали | Правая | Правая | Левая | |
| Шаг спирали (Å) | 28.03 Å | 33.7 Å | 43.5 Å | |
| Число оснований на виток | 11 | 10 | 12 | |
| Ширина большой бороздки | 16.81 Å [DG]33:B.P #657 - [DA]10:A.P #187 |
17.21 Å [DC]4:A.P #64 - [DA]34:B.P #679 | 16.08 Å [DC]4:A.P #64 - [DC]36:B.P #720 |
|
| Ширина малой бороздки | 7.98 Å [DG]1:A.P #1 - [DA]34:B.P #679 |
11.69 Å [DC]36:B.P #720 - [DG]9:A.P #165 | 7.2 Å [DG]29:B.P #575 - [DG]15:A.P #288 |
|
Задание 2. Упражнение 3.
Сравнение торсионных углов в структурах А- и В-форм ДНК.
С помощью команды Settings->Torsion JMol были проведены измерения торсионных углов цитозина.
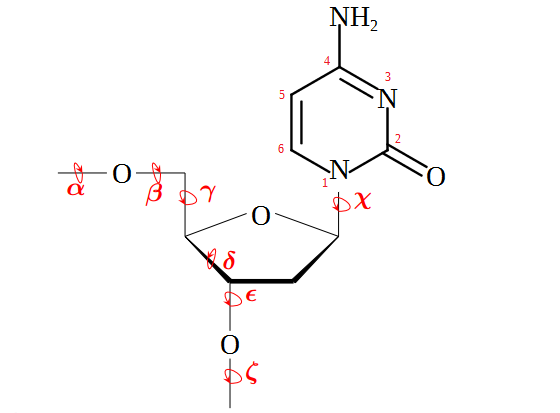
|
Изображение торсионных углов в цитозине |
Полученные результаты сравнивались со значениями, приведенными в презентации.
| Торсионные углы разных форм ДНК в градусах (°) | |||||||
|---|---|---|---|---|---|---|---|
| α | β | γ | δ | ε | ζ | χ | |
| A-форма (в Jmol) | -51.8 | 174.8 | 41.7 | 79.1 | -147.8 | -75.1 | -157.2 |
| A-форма (в презентации) | 62 | 173 | 52 | 88/3 | 178 | -50 | -160 |
| B-форма (в Jmol) | 85.9 | 136.3 | 31.2 | 143.4 | -140.8 | -160.5 | -97.9 |
| B-форма (в презентации) | 63 | 171 | 54 | 123/131 | 155 | -90 | -117 |
Отличия практических (представленных в презентации) и теоретических данных, вероятно, могут объясняться влиянием окружающих ДНК молекул на ее структуру, и, как следствие, на значения торсионных углов.
Задание 3. Упражнение 1.
Определение торсионных углы нуклеотидов c использованием программ пакета 3DNA.
В пакет входят программы для:
- простейшего моделирования структуры нуклеиновых кислот (fiber);
- анализа параметров структуры нуклеиновых кислот (find_pair и analyze);
- получения популярных способов изображения структуры нуклеиновых кислот (pdb2img, stack2img, rotate_mol);
- некоторые вспомогательные программы (например, ex_str, вырезающая фрагмент структуры).
| Средние значения торсионных углов в градусах (°) | |||||||
|---|---|---|---|---|---|---|---|
| α | β | γ | δ | ε | ζ | χ | |
| A-форма | -51.7 | 174.8 | 41.7 | 79.1 | -147.8 | -75.1 | -157.2 |
| B-форма | -29.9 | 136.3 | 31.2 | 143.3 | -140.8 | -160.5 | -98.0 |
| Z-форма (G) | 51.9 | 179.0 | -173.8 | 94.9 | -103.6 | -64.8 | 58.7 |
| Z-форма (C) | -139.5 | -136.8 | 50.9 | 137.6 | -96.5 | 82.0 | -154.3 |
| тРНК (PDB ID 1f7u) | -43.8 | 39.2 | 51.5 | 89.7 | -145.0 | -71.0 | -150.6 |
| "Деформированный" 16G из тРНК | 146.9 | -173.7 | 179.9 | 82.8 | -137.9 | -76.7 | -173.9 |
| ДНК-белковый комлекс (PDB ID 1tro) | -50.5 | 49.2 | 39.7 | 121.4 | -86.2 | -99.0 | -122.8 |
| "Деформированный" 6A из ДНК | -178.9 | -154.4 | 152.6 | 80.1 | -153.3 | -80.1 | -149.6 |
Таким образом, можно сделать вывод, что торсионные углы в А- и В-формах ДНК, несмотря на различия (максимальные из них составили 85.4° и 64.2° для углов ζ и δ соответственно), в целом схожи. Отличия Z-формы от двух других выражены более четко для всех углов: наибольшия расхождения в углах γ и χ (G), β и ζ (C). Заданная структура тРНК наиболее схожа с А-формой, однако мы можем наблюдать явное отклонение угла β. "Странное" значение в 39.2° он принимает потому, что не наблюдается разброса по модулям значений углов разных оснований, и при подсчете среднего арифметического угол фактически обнуляется. Самый "деформированный" нуклеотид в тРНК - 16 гуанин II цепи, в ДНК-белковом комплексе это 6 аденин I цепи.
Задание 3. Упражнение 2.
Определение структуры водородных связей.
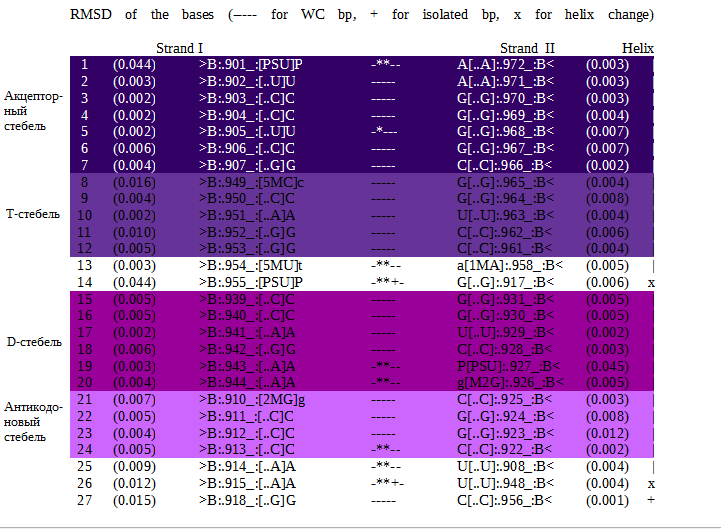
Информацию о нуклеотидах в стемах (стеблях) тРНК можно найти в .out-файле для структуры 1f7u. Stem (стебель) - такой мотив РНК, на котором остатки обеих цепей идут
согласно их нумерации (другие участки не являются стемами). Изображение РНК было получено с помощью Jmol. Разными оттенками сиреневого выделены
соответственно таблице акцепторный стебель, Т-стебель, D-стебель и антикодоновый стебель. Оставшаяся часть молекулы покрашена в белый.
 |
 |
Strand I Strand II Helix
1 (0.044) ....>B:.901_:[PSU]P-**--A[..A]:.972_:B<.... (0.003) |
5 (0.002) ....>B:.905_:[..U]U-*---G[..G]:.968_:B<.... (0.007) |
13 (0.003) ....>B:.954_:[5MU]t-**--a[1MA]:.958_:B<.... (0.005) |
14 (0.044) ....>B:.955_:[PSU]P-**+-G[..G]:.917_:B<.... (0.006) x
19 (0.003) ....>B:.943_:[..A]A-**--P[PSU]:.927_:B<.... (0.045) |
20 (0.004) ....>B:.944_:[..A]A-**--g[M2G]:.926_:B<.... (0.005) |
24 (0.005) ....>B:.913_:[..C]C-**--C[..C]:.922_:B<.... (0.002) |
25 (0.009) ....>B:.914_:[..A]A-**--U[..U]:.908_:B<.... (0.004) |
26 (0.012) ....>B:.915_:[..A]A-**+-U[..U]:.948_:B<.... (0.004) x
Note: This structure contains 9[8] non-Watson-Crick base-pairs.
За счёт водородных связей комплементарных пар оснований, не входящих в стем, происходит стабилизация третичной структуры молекулы.
Strand I Strand II Helix
13 (0.003) ....>B:.954_:[5MU]t-**--a[1MA]:.958_:B<.... (0.005) |
14 (0.044) ....>B:.955_:[PSU]P-**+-G[..G]:.917_:B<.... (0.006) x
25 (0.009) ....>B:.914_:[..A]A-**--U[..U]:.908_:B<.... (0.004) |
26 (0.012) ....>B:.915_:[..A]A-**+-U[..U]:.948_:B<.... (0.004) x
27 (0.015) ....>B:.918_:[..G]G-----C[..C]:.956_:B<.... (0.001) +
Задание 3. Упражнение 3.
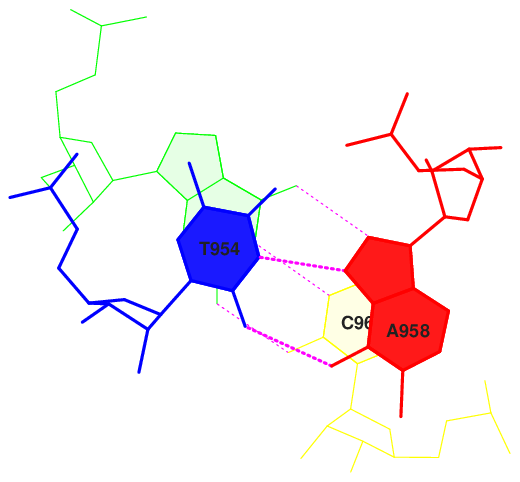
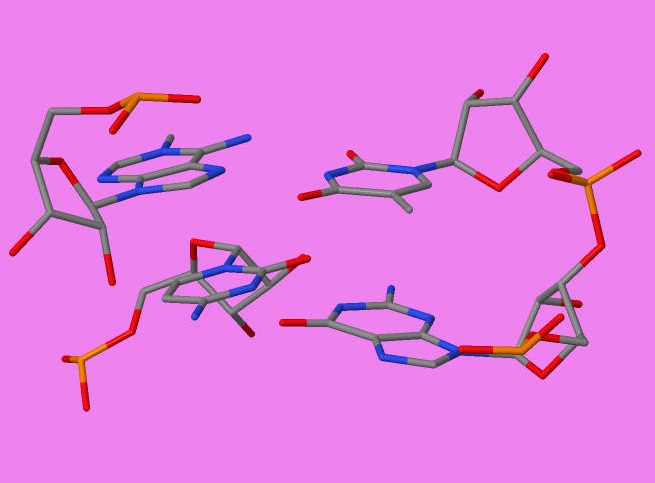
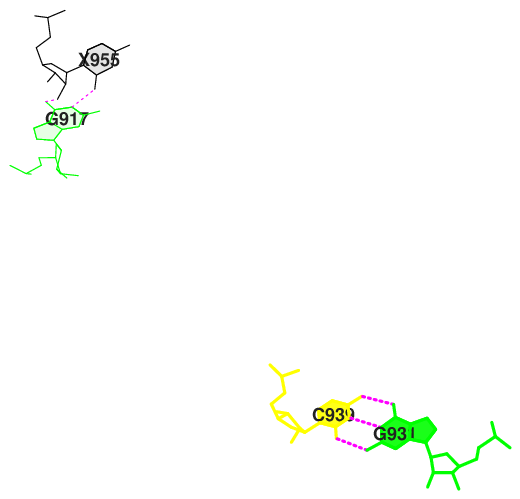
Исследование возможных стекинг-взаимодействий.
Stacking (стэкинг-взаимодействие) происходит между основаниями, уложенными одно над другим. Обусловлено в основном дисперсионными лондоновскими силами и
гидрофобными эффектами. По данным характеристики в файле 1f7u.out было установлено, что наибольшие и наименьшие значения соответствуют приведенным
ниже основаниям.
12 Gt/aC 8.02( 2.73) 0.00( 0.00) 0.00( 0.00) 5.32( 1.87) 13.34( 4.60) 14 PC/GG 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 26 AG/CU 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00)
В файле stacking.pdb хранится информация о структуре всех комплементарных пар РНК, записанных как отдельные модели. Затем, на основе этих моделей программа stack2img (с атрибутом -cdolt) строит изображение стекинг-взаимодействий, получая на вход результат программы ex_str - нужную N-ую модель в формате .pdb. В итоге на выходе мы имеем файл .ps, который можно легко конвертировать в .png. Программа JMol была полезна при проверке взаимной ориентации оснований.
| Стэкинг нуклеотидов тРНК c наибольшим перекрытием | |
|---|---|
| stack2im | JMol |
 |  |
| Стэкинг нуклеотидов тРНК c наименьшим перекрытием | |
| stack2im | JMol |
 |  |