Реконструкция эволюции доменной архитектуры.
Задание 1. Выбор объектов и получение выравнивания.
1. Выбор домена
Для работы был взят центральный домен фактора элонгации транскрипциии TFIIS семейства TFIIS_M (AC: PF07500). Фактор транскрипции S-II (TFIIS) индуцирует расщепление мРНК за счет усиления нуклеазной активности РНК-полимеразы (Pol) II. Он широко распространен среди млекопитающих, дрозофил, дрожжей и архебактерий. Белки S-II имеют относительно консервативную С-концевую область, но вариабельную N-концевую область, а некоторые члены этого семейства тканеспецифично экспрессируются. [1]
2. Выбор доменных архитектур
В выбранное семейство входят 56 различных доменных архитектур, представленных 2341 последовательностью в 804 видах. Среди всех доменных архитектур были выбраны следующие:
| I (Med26, TFIIS_M, TFIIS_C) | II (PHD, TFIIS_M, SPOC) | |
| Схема |  |  |
| Число последовательностей | 887 | 453 |
| Характеристика соседствующих доменов | Med26 (PF08711) - медиатор РНК-полимеразы 2 транскрипционной субъединицы 26,
входит в состав медиаторного комплекса. TFSIIS_C (PF01096) - субъединица С. | PHD (PF00628) - PHD-палец, Cys4-His-Cys3 мотив гомеодоменных
белков HAT3. SPOC (PF07744) - Spen paralogue and orthologue C-terminal, участвует в сигналлинге, связанном с развитием. |
3-6. Выбор таксонов и подтаксонов, получение их совместного выравнивания
Визуализация таксономии последовательностей, входящих в семейство TFIIS_M. [2]
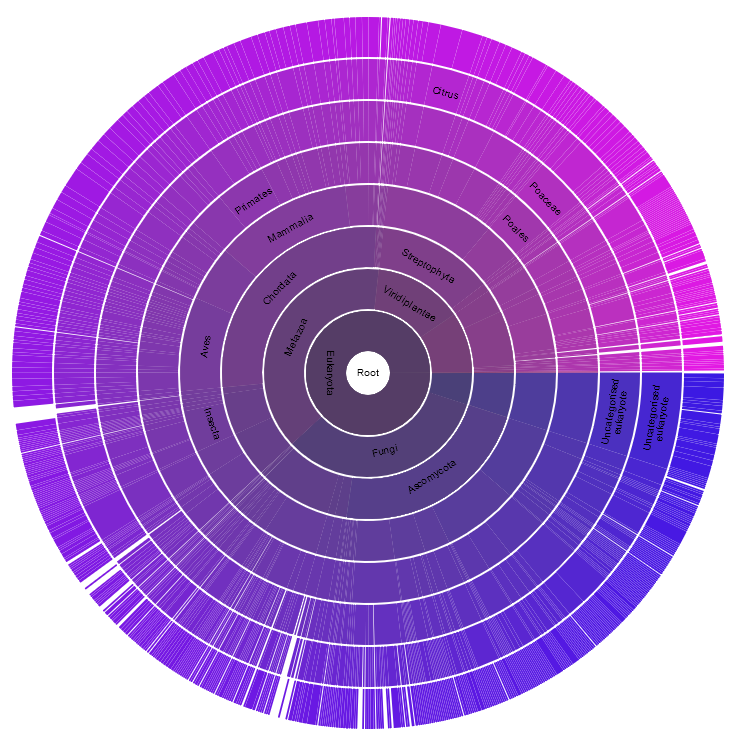
В качестве таксона были выбраны эукариоты, подтаксоны, соответственно, - царства метазои и грибы. Таблица с информацией об архитектуре всех последовательностей, содержащих выбранный домен была получена следующим образом. Файл swisspfam со архитектурами последовательностей Uniprot доступен на сервере kodomo (/srv/databases/pfam/swisspfam.gz). Он был обработан скриптом swisspfam_to_xls.py:
python swisspfam_to_xls.py -z /srv/databases/pfam/swisspfam.gz -p PF07500В Excel файл была создана сводная таблица по последовательностям и соответствующим архитектурам и таксономии. Использовался скрипт uniprot_to_taxonomy.py, которому на вход был подан список с таксономией:
-o PF07500.xls
python uniprot_to_taxonomy.py -i taxa.txt -o taxa.xlsВ таблицу были добавлены таксономия с помощью ВПР (VLOOKUP) и колонка с длиной домена для данной последовательности. Последовательности, удовлетворяющие условиям "Указаний", представлены на отдельном листе.
Для построения выравнивания выбранных мной последовательностей использовался скрипт filter_alignment.py, принимающий на вход fasta-файл со последовательностями, где встречается домен PF07500, и список ID последовательностей, которые надо извлечь (опция -a "/" для отделения имени от координат):
python filter-alignment.py -i all_ali.mfa -m ids -o my_ali.fasta -a "/"При редактировании выравнивания каждой последовательности был присвоен двухсимвольный идентификатор, соотвествующий субтаксону и номеру архитектуры - F1, F2, M1 или M2. Были удалены пустые колонки и плохо выровненные последовательности (в итоге в каждой подгруппе примерно по 20 последовательностей), а также созданы 2 группы, соответсвующие архитектурам 1 и 2 (покраска BLOSUM62 c порогом по консервативности 30%).
Задание 2. Построение филогенетического дерева домена.
На основе выравнивания, содержащего последовательности общего для всех представителей домена, в программе MEGA методом Minimum Evolution с Bootstrap-тестом было построено филогенетическое дерево. Изображение дерева отредактировано в программе iTOL. Величина бутстрэпа отражена в толщине ветвей, цвету фуксии соотвествует подтаксон Fungi, бледно-розовым обозначен подтаксон Metazoa. Названия последовательностей, относящихся к архитектуре 1, покрашены фиолетовым цветом, оставшиеся имеют архитектуру 2.
Филогенетическое дерево эволюции доменов (iTOL)
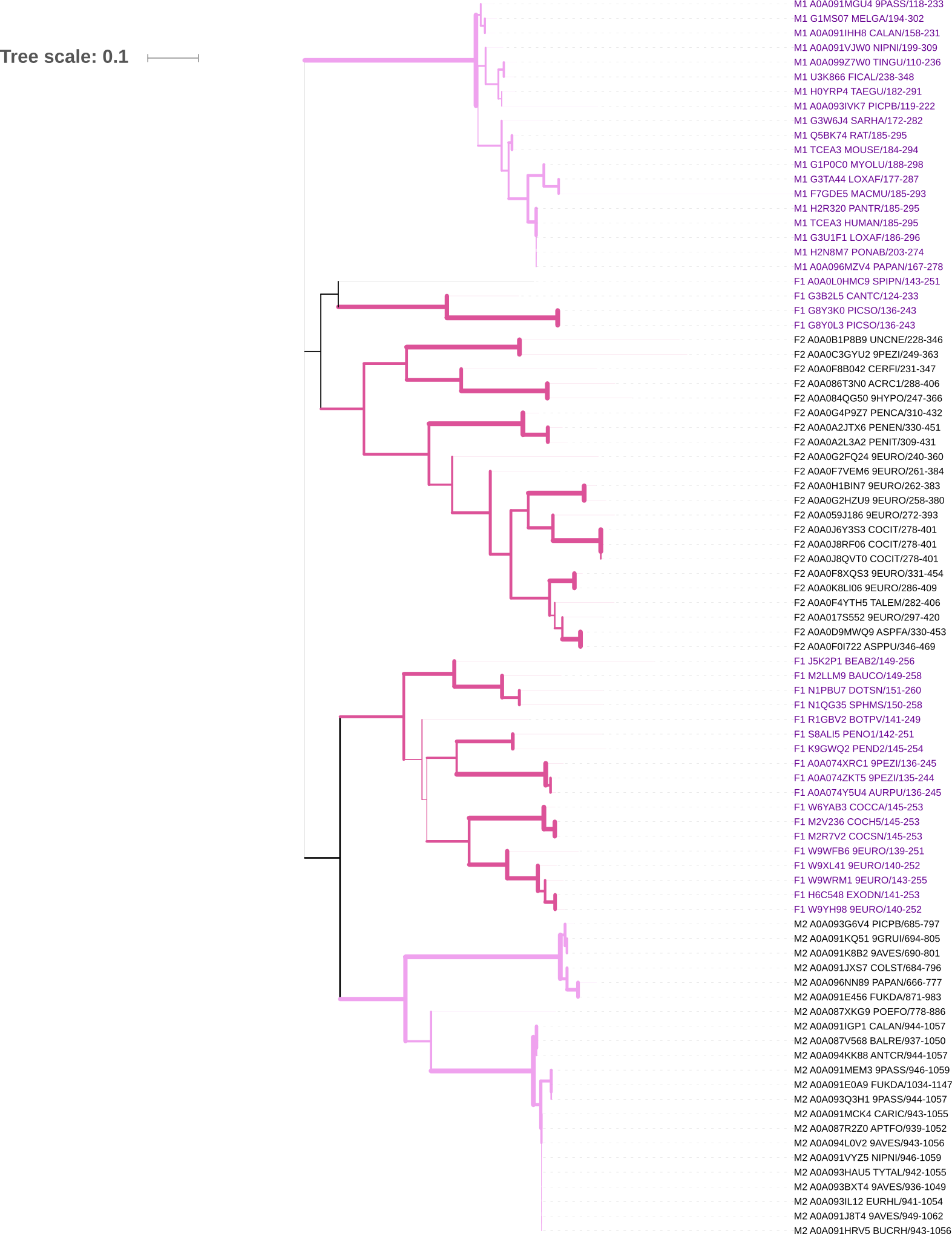
Филогенетическое дерево эволюции доменов (MEGA)

Получилось дерево, где 4 крупные клады соотвествуют каждой из подгрупп F1, F2, M1, M2, однако не наблюдается деления только по архитектурам или только по подтаксонам (т.к. F1 объединена в кладу с М2, а F2 объединена с M1). У M1 и M2 наблюдаются хорошие поддержки (93 и 85). Предположим, что дерево правильно отражает эволюцию домена PF07500 и корень найден верно. Обозначим две верхние клады за Y, а две нижние - за X. Можно видеть, что разделение F1 произошло раньше, чем отделилась F2, значит, 1' (который потом разошелся по таксонам, как, условно, 1Y и 1X) был у общего предка. Независимо от F2, на ветви X также возникает 2 архитектура. Таким образом, LСА имел 1 архитектуру, архитектура 2 возникала несколько раз на разных ветвях.