Реконструкция эволюции доменной архитектуры.
Задание 1. Построение профиля подсемейства, проверка его работы.
Для создания профиля из выравнивания, полученного ранее в практикуме 10 использовался файл
pr11_seqs.fasta, где представлены последовательности "хорошего" подсемейства - M2 (Metazoa с доменной архитектурой 2). Для построения и калибровки профиля применялся
пакет HMMER, установленный на kodomo:
Всего нашлась 3971 последовательность, файл с находками далее анализировался при помощи средств Excel. На листе добавлена колонка "В подсемействе", в ячейках которой стоят 0 или 1 в зависимости от того, входит ли соответствующая последовательность в подсемейство.
По имеющимся данным были построены ROC-кривая и гистограмма, представленные ниже.
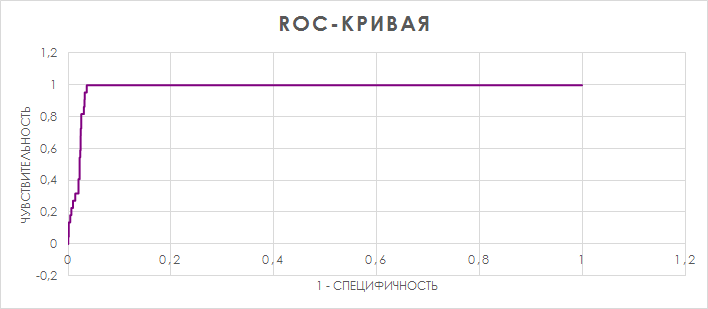
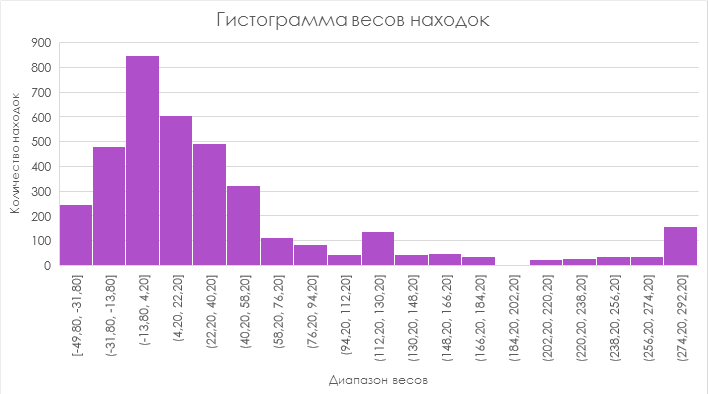
На основании построенной ROC-кривой было выбрано пороговое значение E-value. Критерий выбора - максимум разности [Чувствительность - (1-Специфичность)]. Полученный порог E-value - 2.9e-79. При таком пороге наблюдаются достаточно хорошие чувствительноть (1) и специфичность (0,963297872) профиля, то есть профиль вполне пригоден к использованию для выделения подсемейства.
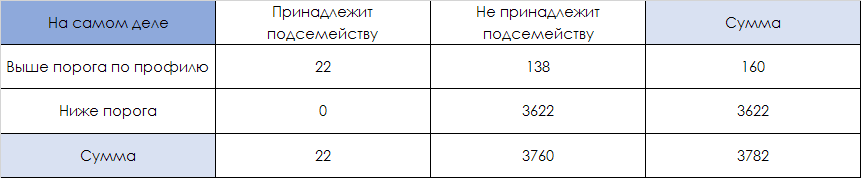
В таблице ниже представлены находки с разделением выше/ниже порогового значения и принадлежностью к семейству.
hmm2build profile pr11_seqs.fastaДалее осуществлялся поиск по всем белкам UniProt, содержащим домен TFIIS_M:
hmm2calibrate profile
hmm2search profile uniprot_pf07500.fasta > pr11_output
Всего нашлась 3971 последовательность, файл с находками далее анализировался при помощи средств Excel. На листе добавлена колонка "В подсемействе", в ячейках которой стоят 0 или 1 в зависимости от того, входит ли соответствующая последовательность в подсемейство.
По имеющимся данным были построены ROC-кривая и гистограмма, представленные ниже.
Изображение ROC-кривой

Гистрограмма весов

На основании построенной ROC-кривой было выбрано пороговое значение E-value. Критерий выбора - максимум разности [Чувствительность - (1-Специфичность)]. Полученный порог E-value - 2.9e-79. При таком пороге наблюдаются достаточно хорошие чувствительноть (1) и специфичность (0,963297872) профиля, то есть профиль вполне пригоден к использованию для выделения подсемейства.
В таблице ниже представлены находки с разделением выше/ниже порогового значения и принадлежностью к семейству.
