Практикум 8
Нуклеотидный BLAST
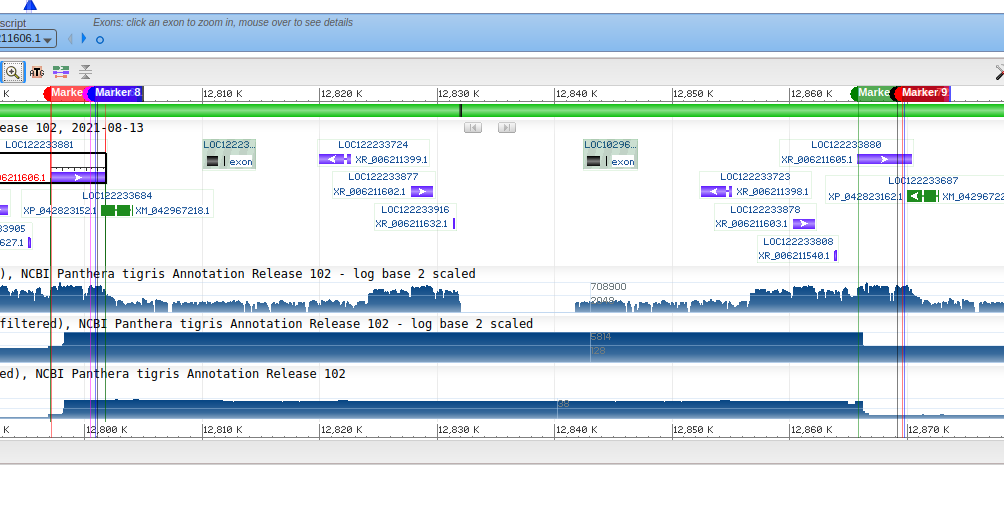
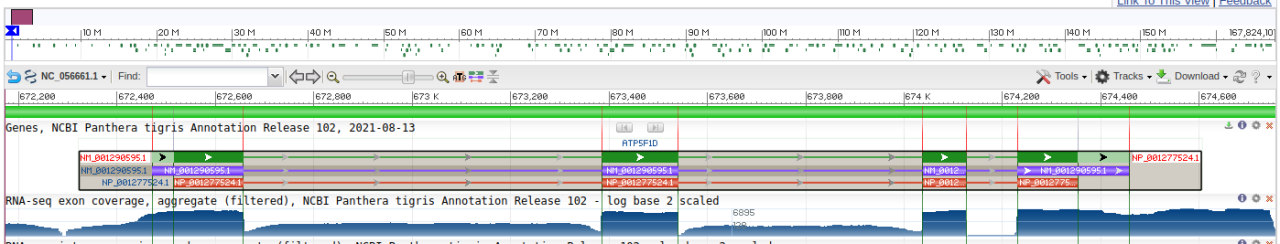
Ген, кодирующий δ-субъединицу АТФ-синтазы
1)NP_001277524.1 - идентефикатор δ-субъединицы АТФ-синтазы, последовательность в формате fasta ссылка
2)NC_056661.1 - идентификатор нуклеотидной записи, к которой относится данный (он находился на хромосоме А2(672472..674457)).
3)Была найдена окрестность (672174..674754) вокруг этого гена, файл с последовательностью ссылка
BLAST
Для данного задания я решил рассмотривать Пауков(Araneae), так как исходный организм был вторичноротым и они достаточно эволюционно удалены. Для изучения была выбрана база данных RefSeq Genome Database (refseq_genomes), общее количество геномных сборок в которой для таксона Araneae было равно 80, из которых 52 помечены как референсные, в базу refseq входят только 4, одну из этих сборок я и взял.
1) Я решил провести поиск с помощью blastn так как он лучше подходит для достаточно удаленных таксонов, в отличии от megablast который ищет практически идентичное совпадение. Я решил сперва искать по гену без окрестности (672472..674457) со стандартными параметрами, только изменил длину слова с 28 до 16, так как при длине слова 28 ожидаемо оказалось 0 находок. При поиске с длиной слова 16, тоже не оказалась находок, я решил в этот раз загрузить последовательность гена с окрестностью, и опять не было найдено никаких находок.
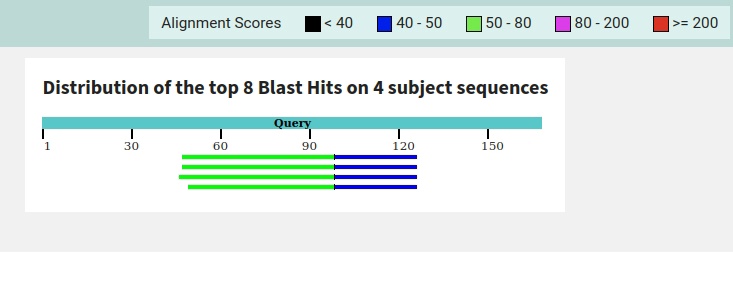
2) Далее я запустил tblastx со стандартными параметрами, было найдено 98 находок. файл с выдачей, несмотря на довольно высокий процент идентичности процент покрытия в локальном выравнивании у всех них был около 10% а так же начиная с 9ой находки E-value переходит порог в 1е-3, что достаточно много.

3) Я решил проверить насколько blastn лучше справится с этой задачей, запустил его с параметрами по умолчанию с длиной слова 5. Было найдено 4 находки. файл с выдачей. Как можно заметить процент покрытия в них при локальном выравнивании был гораздо выше, и учитывая крайне низкое значение E-value и большой процент идентичности можно говорить о том что tblastn позволил найти самые достоверные находки.
Гены рРНК
В этом задании необходимо найти гомологов рРНК нематоды в геноме выбранного эукариота. Сперва я проиндексировал свой файл с геномом с помощью команды:
makeblastdb -in genomic.fna -dbtype nucl
затем я скачал необходимые последовательности рРНК для 16s и 23s субьединиц соотвественно и начал поиск с помощью blastn. Все значения были оставлены по умолчанию, для вывода в табличном виде был дан аргумент -outfmt 7
blastn -task blastn -query 16s.fna -db genomic.fna -out 16srRNA.txt
файл с выдачей
blastn -task blastn -query 16s.fna -db genomic.fna -out 16srRNA -outfmt 7
файл с выдачей
blastn -task blastn -query 23s.fna -db genomic.fna -out 23srRNA.txt
файл с выдачей
blastn -task blastn -query 23s.fna -db genomic.fna -out 16srRNA -outfmt 7
файл с выдачей
Итого было найдено 23 находки для 23s рРНК и 16s для 16s, сразу можно отсечь находки с высоким evalue, тогда остается 4 хита для 16s и 9 для 23s, все 13 находок располагаются на хромосоме Е1(NC_056673.1) что упрощает работу. Теперь рассмотри каждый выравненный участок на этой хромосоме.
1)Для 4ех находоко для 16s хромосомы все чуть проще, все они находились в конце учатсков помеченных как 18s рРНК, следовательно все они достоверно являются гомологами.
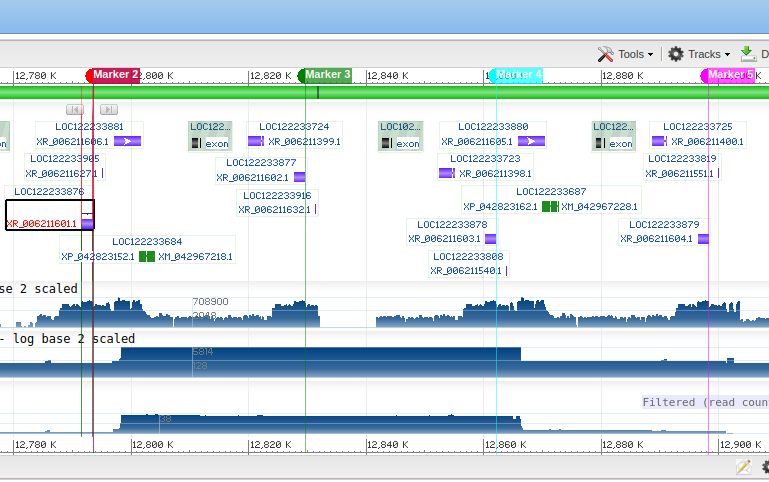
2)Все 9 находок для 23S рРНК лежали на 2ух участках помеченных как 28s рРНК следовательно всего 2 гомолога.