Практикум 9. Домены и профили.
Для выполнения задания 1 были выбраны метилтрансферазные домены YjbR (pf04237) и DNA_binding_1 (pf01035) (архитектура на рис.1). В общем, два домена, достаточно часто составляющих целую рабочую метилтрансферазу.
Таблицу из UNIPROT можно скачать по ссылке.
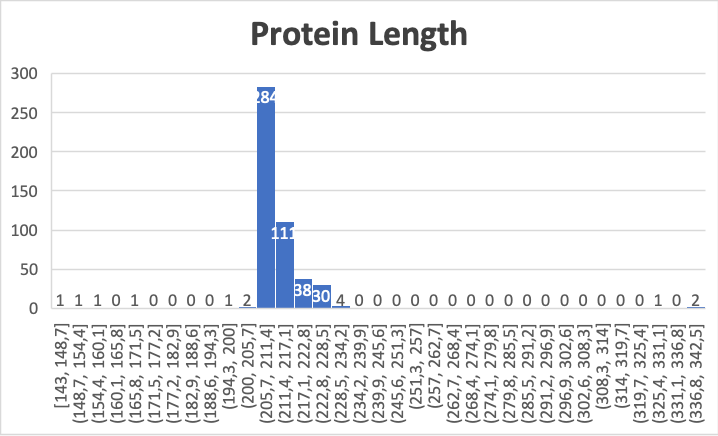
Поскольку при проверке практикума возникли проблемы с отображением гистрограммы из таблицы, она представлена в виде рис.2. Как видно из данной схемы, мода содержится в промежутке (205,7 , 211,4); медиана при этом равна 209 а.к.
Было случайно выбрано 53 белка с длинной, попадающей в этот промежуток.
Эти белки были выделены из организмов, принадлежащих следующим семействам: Clostridiaceae, Eubacteriaceae, Lachnospiraceae, Ruminococcaceae.
Далее было построено множественное выравнивание всех упомянутых выше белков в Jalview с помощью алгоритма Muscle с def параметрами (рис. 3). Выставлена окраска Blossum6 by conservation 30. С левого конца я удалила 3 колонки, с правого - 6. При этом было удалено некоторое количество последовательностей, откровенно портивших выравнивание, и их общее число сократилось до 45.
Для построения HMM профиля были задействованы следующие команды:
Для дальнейшей работы я взяла архитектуру из pf04237 (мтазный домен; 28803 последовательности).
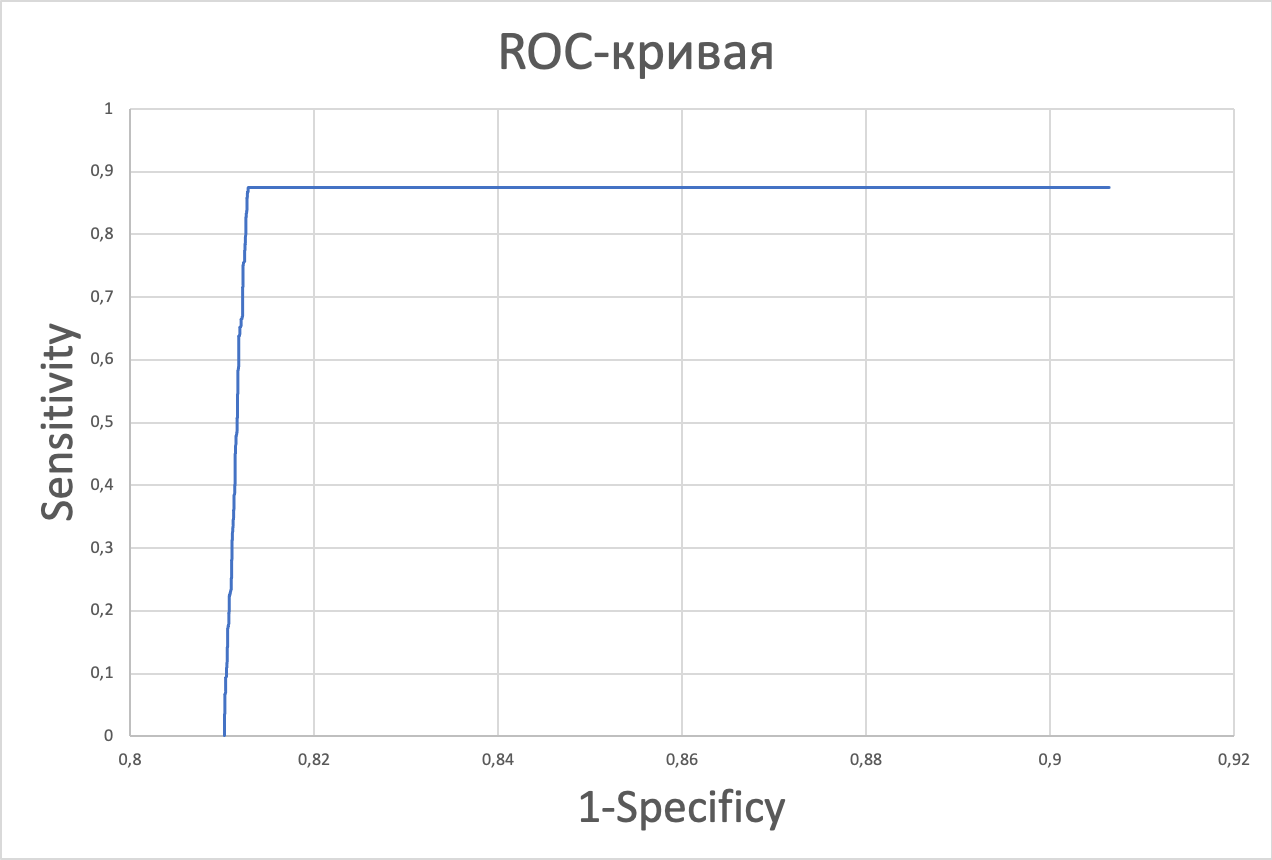
Далее была предпринята попытка построить ROC-кривую. Все результаты можно посмотреть по ссылке.
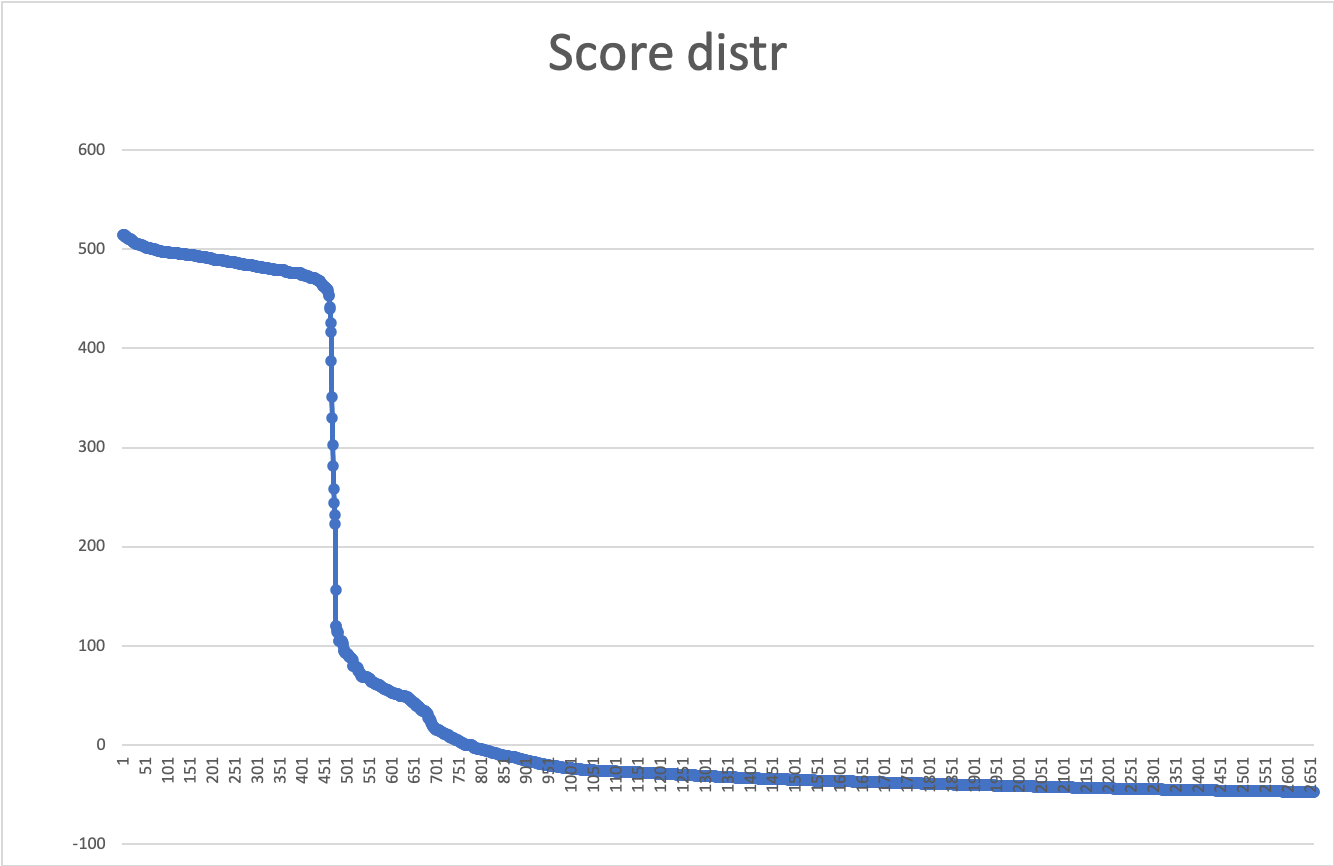
Определенный мной порог для score оказался равен 232,2 (рис.4), что даже похоже на правду (рис. 5). Специфичность и чувствительность: 0,81286976 и 0,86792453, E-value: 3,00E-66. Сравнительная табличка, также входящая в состав excel-файла, представлена на схеме 1.
Мне кажется, что тест оказался слабоват - страдает чувствительность. У нас получилось достаточно много True positive и действительно много False negative (высокйи False Negative Rate). Это в целом наверное может быть оправданно, если мы больше боимся ложно-позитивных результатов <=> ошибки второго рода (False Negative).
ʕ•ᴥ•ʔ



hmm2build out_prof strict_alig.fa
hmm2calibrate out_prof
hmm2search out_prof uniprot-PF04237.fasta > resulting_search
True False Sum Positive 414 4 418 Negative 60 2183 2243 Sum 474 2187 2661