1. Описание выбранного домена
Я выбрал домен,с белками, содержащими который, мы работаем в лаборатории.
АС: PF05199
ID: GMC_oxred_C
Функция:
Данный домен содержат ферметы суперсемейства глюкоза-метанол-холин оксидоредуктаз GMC-superfamily. Белки данного семейства, вообще, содержат как минимум два домена: N-коцевой, связывающий
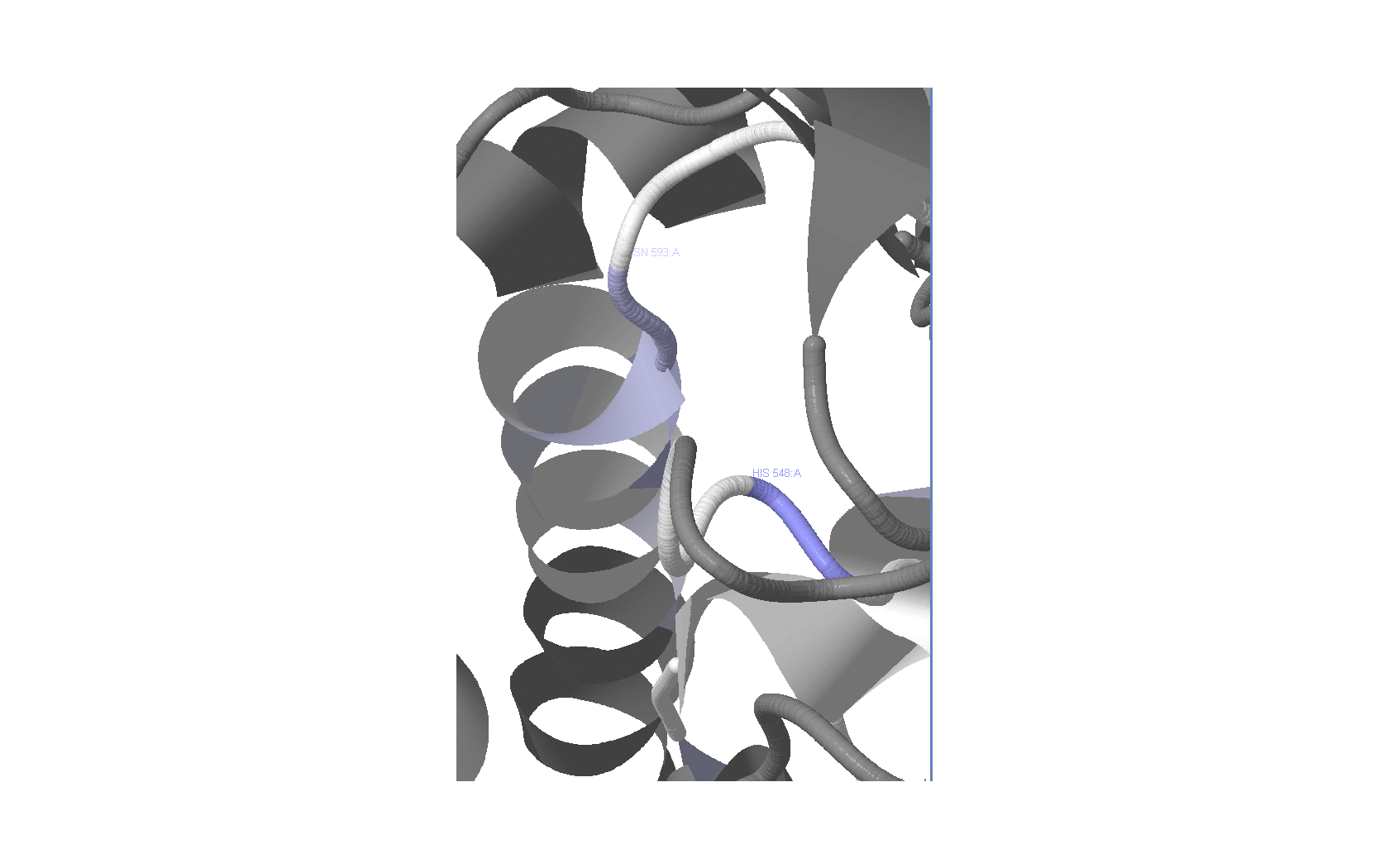
кофактор FAD, и мною выбранный С-концевой, осуществляющий каталитическую активность. Последний содержит два каталитических остатка: гистидин (виден на выравнивании как очень консервативный), депротонирующий гидроксильные группы в каталитическом акте, и аспарагин (либо гистидин),
стабилизирующий анион (гидроксил без протона).
Из изображения Jmol видна консервативность каталитических аминокислот в домене.
Число различных доменных архитектур с данным доменом: 77
Число представителей по таксонам высшего порядка:
| Alveolata
|
Euglenozoa
|
Stramenopiles
|
Metazoa
|
Amoebozoa
|
Fungi
|
Rhizaria
|
Viridiplantae
|
Ichthyosporea |
Choanoflagellida
|
Heterolobosea
|
Bacteria
|
Archaea
|
Viruses
|
| 8
|
8
|
6
|
6
|
3
|
139
|
1
|
28
|
1
|
1
|
1
|
1206
|
30
|
1
|
Ccылка на страницу домена в Pfam
Общее выравнивание из Pfam
2. Выбор доменных архитектур
Первая доменная архитектура наиболее распространена: 4542 представителей ею обладают. Первый домен в этой архитектуре - это N-коцевой, связывающий
кофактор FAD, домен. Вторая архитектура более скромна: имеет лишь 101 представителя. Новый домен DAO - это домен оксидаз D-амино кислот, которые заменяют
аминогруппу на кетогруппу.
К сожалению, скрипт swisspfam_to_xls.py у меня не сработал, поэтому пришлось пойти иным путем, для того чтобы получить информацию об архитектуре всех последовательностей,
содержащих выбранные домены.
На странице с доменными архитектурами Pfam кликнул показать все последовательности с данной архитектурой и копировал содержимое в файл.
Воспользовался командой awk в командной строке, чтобы получить все ID.
Не использовал ID mapping, так как было известно, что сервис Uniprot Retrieve способен осуществлять поиск и по ID.
Скачал полные записи всех последовательностей.
3. Выбор таксонов
Скрипт python uniprot_to_taxonomy.py прекрасно сработал.
Для перовой доменной архитектуры были выбрано несколько царств: животные, сосудистые растения, бактерии, археи, грибы.
Вторая оказалась менее богатой на таксоны: взял два отдела бактерий - актино- и протеобактерий.
В файле Exel находится полная информация о таксономии представителей каждой доменной архитектуры, а также выбранных последовательностей.
4. Выбор представителей архитектур
Чтобы оставить в имеющемся выравнивании лучших представителей архитектур, я попытался воспользоваться скриптом filter_alignment.py. Он,
к несчастью, не сработал. Я решил эту проблему,
использовав свой скрипт fasta2csv.final.py, приготовленные на занятиях по питону,
и написав свой скрипт filter.py, который выделяет из выдачи предыдущего по ID необходимые последовательности.
Мой подход не учитывает длину домена, поэтому изначально я с избытком выбрал представителей, а затем просто удалил из выравнивания в Jalview плохие последовательности.
Итоговое выравнивание представлено здесь.




