База данных ОРМ
Выбранный белок - COLY_YERPE
В данном задании я работал с коагулазой PLA бактерии-возбудителя чумы Yersinia pestis (PDB ID: 2x55; UniProt ID: COLY_YERPE; Plasminogen activator PLA (coagulase/fibrinolysin)). Этот белок является протеазой и катализирует расщепление плазминогена с образованием плазмина, который в свою очередь запускает каскад свертывания крови. Эта активность фермента важна для вирулентности патогена. Также было показано, что PLA имеет высокое сродство к фибронектину и, таким образом, участвует в адгезии бактериальных клеток к клеткам хозяина (Eren et al., 2010).
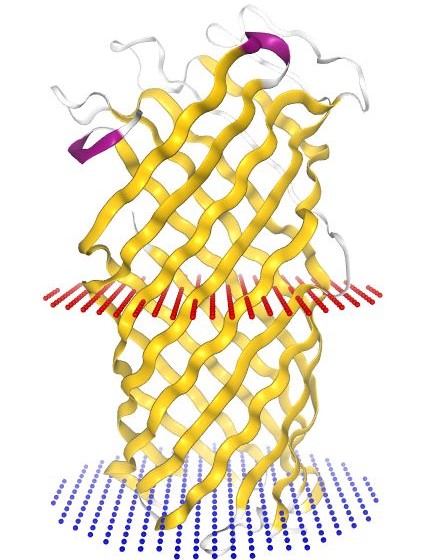
Информация о структуре трансмембранных участков выбранного белка, взятая из базы данных OPM, представлена ниже. С помощью пакета nglview для языка python было получено изображение, отражающее положение белка в мембране (Рис. 1). Трансмембранные участки коагулазы PLA Yersinia pestis.
- Толщина гидрофобной части белка в мембране: 25.6 Å
- Координаты трансмембранных участков: 1(13-22),2(51-60), 3(65-73),4(111-121), 5(126-136),6(174-183), 7(188-197),8(224-235), 9(239-248),10(282-292)
- Среднее число остатков в β-тяже: 10.4 остатка
- Тип мембраны: Внешняя мембрана Грам-отрицательной бактерии
DeepTMHMM: Предсказание трансмембранных элементов по последовательности белка
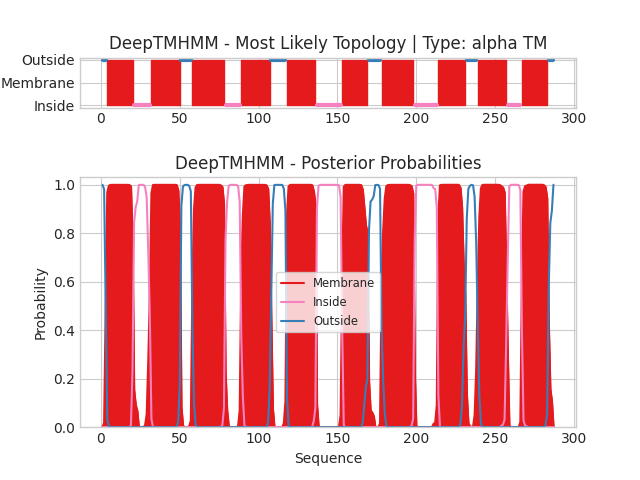
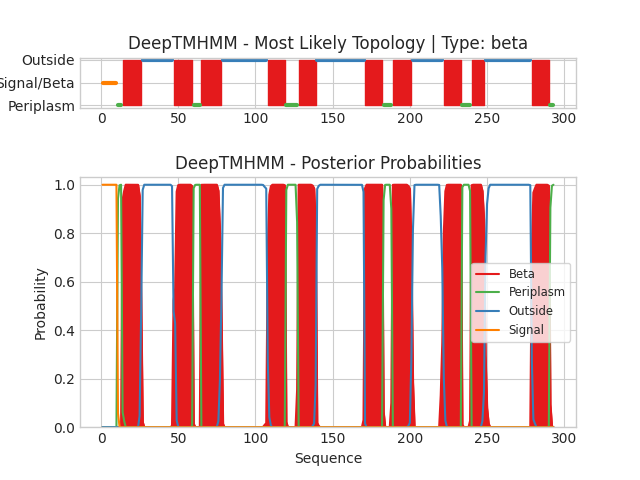
Данное задание состояло в использовании сервера DeepTMHMM для предсказания трансмембранных участков в исследуемых белках Y170_LACJO (альфа-спиральный) и COLY_YERPE (бета-листовой).
Результаты работы сервера в текстовом виде доступны по данным ссылкам:
Графическая выдача представлена на рисунках ниже (Рис. 2, 3).
По оси X в каждом из графиков отложена позиция в первичной последовательности белка (номер остатка начиная с N-конца). По оси Y в верхнем графике (на обеих картинках) отложено наиболее вероятное положение аминокислотного остатка по отношению к мембране, а в нижнем - вероятность каждого из положений. Цвет линий в обоих случаях отражает положение данного участка цепи (красный: в толще мембраны, синий: с наружной стороны, розовый: с внутренней).
PPM: Предсказание положения выданного белка в мембране
Выбранный белок - Y170_LACJO
В данном практикуме я работал с предполагаемым белком для поглощения сахара LJ_0170 бактерии Lactobacillus johnsonii (strain CNCM I-12250 / La1 / NCC 533). Молекулярная функция: активность трансмембранного переносчика углеводов. Участвует в биологическом процессе транспортировки сахара. Расположен в клеточной мембране. Бактерии Lactobacillus johnsonii (strain CNCM I-12250 / La1 / NCC 533) является частью здоровой вагинальной микробиоты и был идентифицирован как обладающий пробиотическими свойствами. Штамм L. johnsonii La1 был одной из первых культур, которые были предложены в качестве пробиотической добавки к молочным продуктам в 1995 году в Исследовательском центре Nestlé в Лозанне.
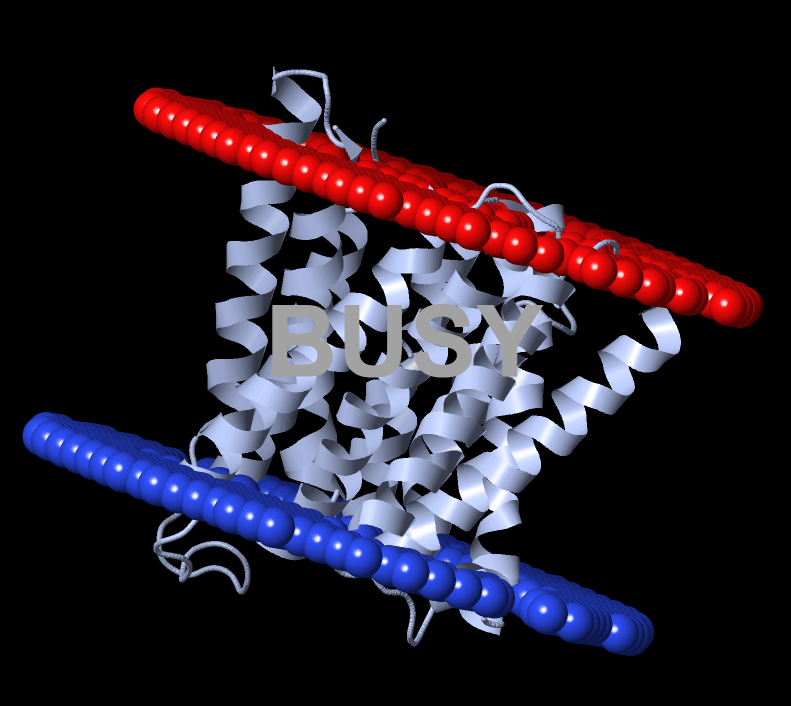
В данном задании положение выданного мне альфа-спирального белка Y170_LACJO в мембране было предсказано с помощью сервера PPM 3.0.
При запуске анализа на сервере тип мембраны был взят за "Gram-positive bacteria inner membrane", так как золотистый стафилококк относится к Грам-положительным бактериям. Был оставлен запрет на изгибы мембраны (белок маленький, ими точно можно принебречь), положение N-конца было взято за "out" в соответствии с предсказанием сервера DeepTMHMM (Рис. 2, началу цепи соответствует синий цвет линии). Параметры трансмембранных участков белка согласно серверу PPM приведены ниже
- Толщина гидрофобной части белка в мембране: 30.7 Å
- Координаты трансмембранных участков: 1( 3- 21), 2( 28- 50), 3( 56- 80), 4( 85- 108), 5( 118- 135), 6( 149- 172), 7( 177- 199), 8( 210- 231), 9( 236- 258),10( 265- 285)
- Среднее число остатков в α-спирали: 25.1 остатка
- Тип мембраны: Мембрана Грам-положительной бактерии
Сравнение алгоритмов предсказания трансмембранных спиралей
Для белка Y170_LACJO сервер DeepTMHMM предсказал 10 трансмембранных альфа-спиралей, что соответствует его структуре из базы данных PDB, полученной экспериментально методом рентгеноструктурного анализа. При этом координаты найденных трансмембранных участков примерно совпадают с указанными в базе данных OPM. Таким образом, в данном случае предсказание DeepTMHMM является достаточно точным.
Для белка COLY_YERPE сервер DeepTMHMM предсказал 10 трансмембранных бета-тяжей, что соответствует его структуре из базы данных PDB, полученной экспериментально методом рентгеноструктурного анализа. При этом координаты найденных трансмембранных участков примерно совпадают с указанными в базе данных OPM. Таким образом, в данном случае предсказание DeepTMHMM является достаточно точным.