Построение и характеристика профиля
Сперва мне нужно было правильно выбрать подсемейство из выравнивания последовательностей домена из предыдущего задания. Я старалась удовлетворить следующим требованиям:
- Подсемейство является кладой на дереве
- По доменной архитектуре, таксономической принадлежности или по диагностическим позициям или признакам в последовательности можно однозначно определить, принадлежит ли какой-либо белок с рассматриваемым доменом Usher выделенному подсемейству или нет
Этим правилам более-менее соответствует подсемейство из последовательностей с кратким обозначением BA. Отмечено серым цветом на изображении:
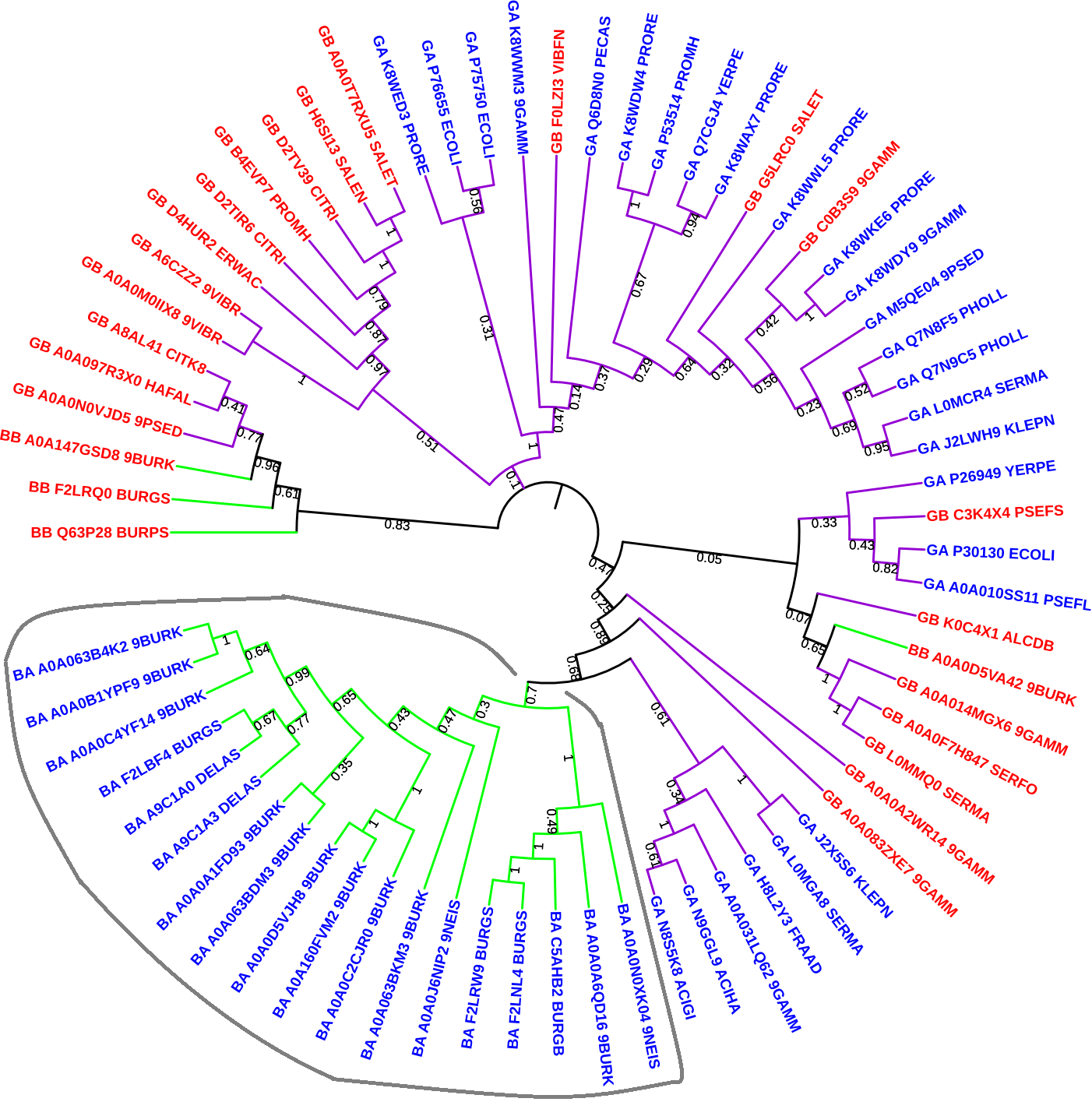
Выбранные последовательности я сохранила отдельно в файле subfamily.fasta. По ссылке доступно изображение выравнивания последовательностей подсемейства с окраской по типу ClustalX с порогом консервации 20%
Для построения профиля я использовала пакет HMMER, который установлен на сервере kodomo. Сначала я построила профиль программой hmm2build:
hmm2build profile.out subfamily.fasta
Затем я откалибровала профиль программой hmm2calibrate:
hmm2calibrate profile.out
Далее я скачала с сайта UniProt последовательности всех белков, включающих домен Usher, в один файл uniprot.fasta. Провела поиск по всем белкам программой hmm2search, используя откалиброванный профиль:
hmm2search profile.out uniprot.fasta > output.txt
В итоге я получила файл output.txt, содержащий все находки.
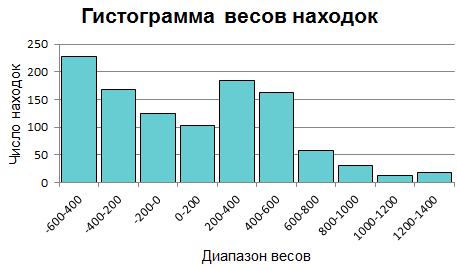
Таблицу с результатами я открыла в программе Excel, после небольших преобразований скриптом. Всего было обнаружено 1088 , среди которых встретились все представители подсемейства (выделены желтым цветом в таблице subfamily.xlsx лист findings). По данным я составила гистограмму весов находок (лист histogram в таблице subfamily.xlsx), ее изображение приведено ниже.
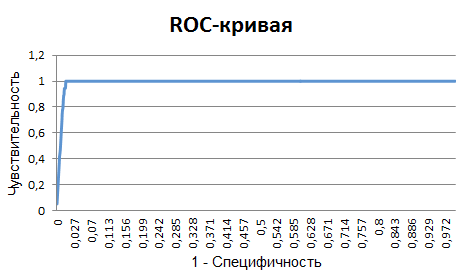
Далее я вычислила показатели чувствительности (SE) и специфичности (SP) и построила ROC-кривую (лист ROC в таблице subfamily.xlsx). Ее изображение приведено ниже.
В качестве порога для нормализованного веса профиля я предлагаю значения:
- E-value = 0
- Score = 1083
На основании графика ROC-кривой, я вычислила максимальную разность (SE + SP - 1) между показателями чувствительности и 1 - специфичности. Она оказалась равной 0,993. При этом значении:
- SE = 1
- SP = 0,007
График ROC-кривой выглядит практически идеально. Мне кажется, что построенный профиль будет эффективен для выделения подсемейства. При выбранном пороге результаты поиска будут как в таблице 1.
| Таблица 1. Результаты поиска при выбранном пороге | |||
| На самом деле | Принадлежит подсемейству | Не принадлежит подсемейству | Сумма |
| Выше порога по профилю |
18 | 7 | 25 |
| Ниже порога по профилю |
0 | 1063 | 1063 |
| Сумма | 18 | 1070 | 1088 |