BLAST
Знакомство с BLAST
В данном практикуме требуется найти гомологов своего белка в базе данных Swissprot при помощи сервиса BLASTp.
Ход работы
При помощи BLAST были найдены сходные с моим белком (Lactate racemase) последовательности в базе данных SwissProt.
Заданные при запуске BLAST параметры:
Параметр |
Значение |
Смысл |
|---|---|---|
| Accession number | ABC45135.1 | Accession number - код доступа данной последовательности в NCBI. |
| Database | UniProtKB/Swiss-Prot(swissprot) | База данных, для поиска последовательностей |
| Organism | - | С помощью этого параметра можно ограничивать находки последовательностей по их принадлежности к определённым таксонам организмов |
| Max target sequences | 2000 | Максимальное количество находок сходных последовательностей |
| Short queries | ✓ | Автоматическая настройка параметров для коротких входных последовательностей |
| Expect threshold | 100 | Ограничение сверху на значение E-value |
| Word size | 2 | Длина слова из исходной последовательности, которое ищется в других последовательностях банка и которое инициирует выравнивание с этими последовательностями |
| Max matches in a query range | 0 | Ограничивает количество находок, убирая середине значеия, что даёт возможность увидеть более слабые совпадения |
| Matrix | BLOSUM62 | Выбор матрицы весов замен |
| Gap Costs | Existence: 11 Extension: 1 | Штраф за наличие и продолжение инделя (разрыва) |
| Compositional adjustments | Conditional composition score matrix adjustment | Метод корректировки матрицы, подстраивающий её под определённую аминокислотную последовательность |
| Filter low complexity regions | - | Маскировка участков низкой сложности |
| Mask for lookup table only | - | Применение маски только на начальном этапе поиска совпадений, но не при расширении выравнивания |
| Mask lower case letters | - | Маскирует строчные буквы у входной последовательности |
Результаты осуществления поиска совпадающих последовательностей с помощью BLASTp приведены в таблице Exel
Далее было построено множественное выравнивание с выбранными из этого списка последовательностями, которые приведены на листе "Selected". Отбирались белки, имеющие большее покрытие, но с сильно различающимися значениями E-value, из начала, середины и конца списка. Даже не смотря на то, что эти последовательности имеют нехудшее покрытие, а их E-value отличается всего на несколько единиц (в отличие от E-value первых двух последовательностей, которое отличается на 162 порядка), по результатом выравнивания можно предположить, что они не гомологичны исходной последовательности. С помощью программы jalview выравнивание было визуализированно. Из выравнивания пришлось убрать две последние последовательности, так как они чаще всего выпадали из общего сходства. Скачать отредактированное выравнивание.
Карта локального сходства двух белков
Чтобы составить карту локального сходства в BLAST необходимо активировать опцию "Align two or more sequences". Для этого задания были взяты два белка: B8P6F4 и F4RBD6. Параметру "Word size" было задано значение 3.
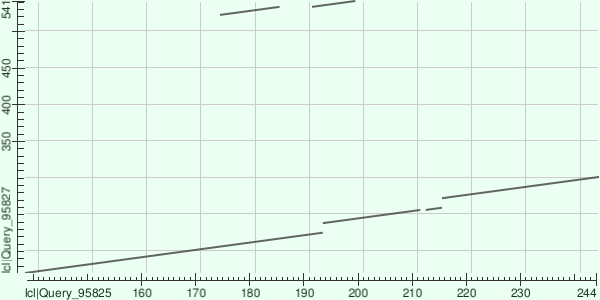
Карта показала впечатляющее сходство. На ней изображена практически непрерывная линия, что говорит о близком родстве данных белков. Об этом свидетельствуют и другие характеристики: вес выравнивания превышает максимальный, а E-value составляет 4e-08. Однако покрытие составляет всего 40%.
На карте также можно увидеть дублирующийся участок в вертикальной последовательности (B8P6F4), что говорит о произошедшей дупликации.
Игры с BLAST
В данном задании проводился поиск по случайной небелковой последовательности. Мной была введена следующая последовательность: "GHHJKXKGZVOPAKGJFHGJKKXHVKJVNRNICMXHJDKDNFFFKCNYNCSKALNAU".
Изменённые параметры поиска:
Параметр |
Значение |
|---|---|
| Database | UniProtKB/Swiss-Prot(swissprot) |
| Max target sequences | 2000 |
| Expect threshold | 50 |
| Word size | 6 |
По-моему, моя последовательность оказалась не такой уж и откланяющейся:
① Минимальное E-value составило 0.077
② Процент идентичности достиг 55.56%
③ Максимальный вес составил всего 32.3
④ А максимальное покрытие 85%
⑤ Всего 20 находок
В поиске небелковой последовательности большое значение имеет параметр длины слова. Было предположение, что, так как "небелковой" последовательность делает именно сочетание букв, и, следовательно, чем оно длиннее, тем менее вероятно, что оно встретится в белковой последовательности, но почему-то на практике это не полностью подтвердилось: при изменении длины слова с 2 на 3 количество находок увеличелось на семь последовательностей, при изменении на 6 оно составило 5 последовательностей.
При изменении в первичных параметрах только матрицы весов замен на PAM250 количество находок сократилось до 17, минимальный E-value поднялся до 3.4, вес упал до 27.2, выровнялись новые последовательности.
При поиске последовательности в другом банке модельных организмов landmark, были найдены 18 последовательностей, все граничные значения находок остались теми же, что и в банке , кроме минимального E-value, он составил 0.091 и максимального процента идентичности, он понизился на 12.08%.
Поиск последовательности своего белка в Swiss-Prot
Здесь необходимо изменять параметры BLAST и сделать выводы об этих параметрах на основании полученных результатов.
В поиске небелковой последовательности большое значение имеет параметр длины слова.
Будем изменять параметры по очереди и смотреть как изменяются число находок и E-value.
Database |
Число находок |
Минимальное E-value |
|---|---|---|
| UniProtKB/Swiss-Prot(swissprot) | 67 | 0.0 |
| Model Organizms (landmark) | 105 ссылка на таблицу находок | 0.0, скачёк после второй послеовательности также большой, но не на 162 порядка, а на 11 |
Organism |
||
| - | 67 | 0.0 |
| Homo sapiens neanderthalensis (taxid:63221) | 1 ссылка на таблицу находок | 0.63 (Osteocalcin) |
Expect threshold |
||
| 100 | 67 | 0.0 |
| 50 | 34, изменилось только количество находок, ссылка на таблицу находок | 0.0, скачёк такой же |
Word size |
||
| 2 | 67 | 0.0 |
| 6 | 10 ссылка на таблицу находок | 0.0, скачёк тот же |
Max matches in a query range |
||
| 0 | 67 | 0.0 |
| 5 | 11 ссылка на таблицу находок | 0.0, скачёк тот же, уменьшилось количество последовательностей со средним значением |
Matrix |
||
| BLOSUM62 | 67 | 0.0 |
| PAM250 | 94 ссылка на таблицу находок | 0.0, скачёк на 156 порядка |
Мы видим, что параметр Word size снова сильно ограничил количество находок, E-value изменилось незначительно. Как и предпологалось, параметр "Max matches in a query range" ограничил количество находок, но не их последовательность. База данных, в которой искались последовательности, тоже играет важную роль при получении результатов поиска: сильно изменилисть и E-value, и количество находок. Ограничение на отношение к разным таксонам организмов, сократило количество сходных последовательностей до 1. Матрица весов замен изменила несущественно оба значения и отобрала новые последовательности.