Команды, используемые в практикуме:
| Команда | Результат |
| fastqc SRR4240388.fastq, fastqc noad_SRR4240388.fastq, fastqc trnoad_SRR4240388.fastq | Zip-файлы и файлы html - 1,2,3 с анализом чтений |
| java -jar /nfs/srv/databases/ngs/suvorova/trimmomatic/trimmomatic-0.30.jar SE -phred33 SRR4240388.fastq noad_SRR4240388.fastq ILLUMINACLIP:adapters.fasta:2:7:7 | Чтения с удаленными остатками адаптеров, последовательности которых лежат в файле adapters.fasta |
| java -jar /nfs/srv/databases/ngs/suvorova/trimmomatic/trimmomatic-0.30.jar SE -phred33 noad_SRR4240388.fastq trnoad_SRR4240388.fastq TRAILING:20 MINLEN:32 | Чтения с обрезанными с концов нуклеотидами, у которых качество больше 20 и длина более 32 нуклеотидов в файле short2_chr10.fastq |
| velveth hashread 31 -fastq -short trnoad_SRR4240388.fastq | Создание k-меров длиной 31 нуклеотид |
| velvetg hashread | Сборка k-меров |
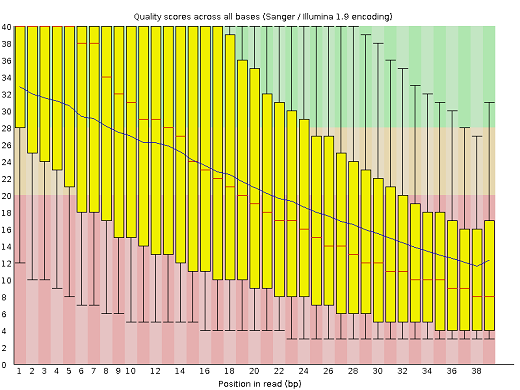
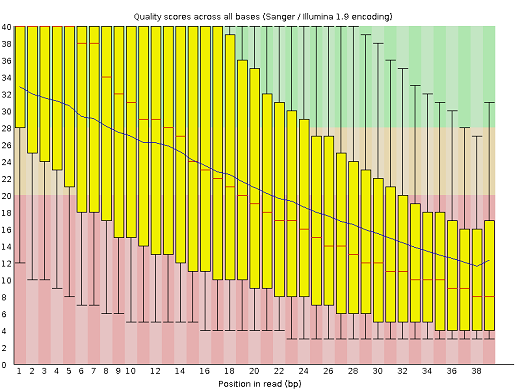
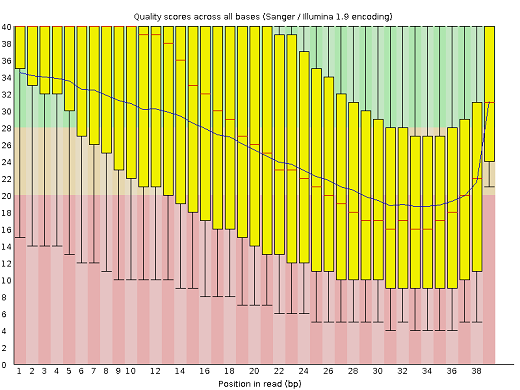
С помощью программы trimmomatic были удалены остатки адаптеров и оставлены риды с качеством более 20 и длиной более 32 нуклеотидов. После первой команды из 10833163 2998 (0.03%) было удалено. После второй команды 4154245 (38,36%) из оставшихся было удалено. В итоге осталось 6675920 ридов. Размеры файлов соответсвенно составили 1157 M, 1156 M и 685 M. Результаты fastqc до использования trimmomatic и после использования 2 команд:



C помощью команд velveth и velvetg были получены контиги. Информация о 3 самых длинных контигах и 2 нетипичных представлинна в таблице 2:
| ID | 9 | 25 | 4 | 805 | 522 |
| Длина | 13950 | 10101 | 9640 | 1 | 144 |
| Покрытие | 55,77 | 38,42 | 58,13 | 481550 | 3,875 |
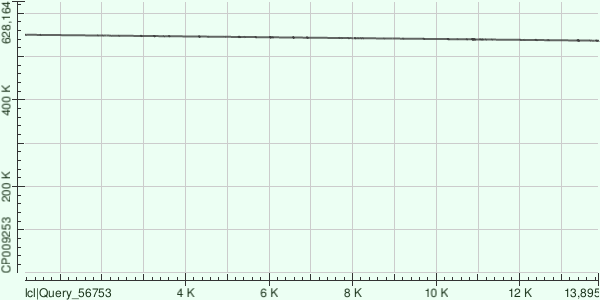
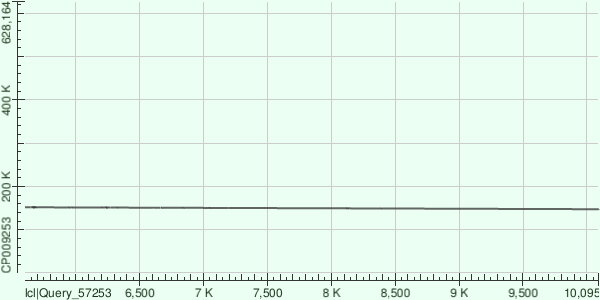
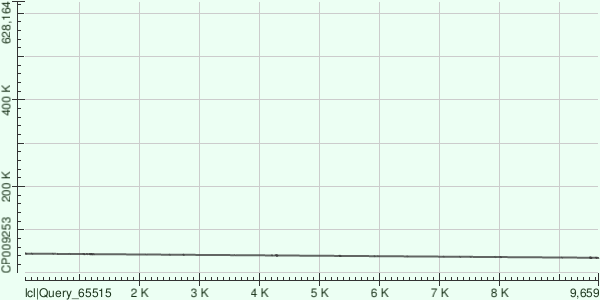
3 самых длинных контига были сравнены программой megablast с хромосомой Buchnera aphidicola. В резльтате для каждого контига было выдано 1 выравнивание. Особенности выравниваний можно увидеть в таблице 3:
| ID | Query length, bp | Query cover, % | Координаты выравненной части контига | Score, bits | Identities, % | Gaps | Strand |
| 9 | 13950 | 98 | 536475 - 550219 | 10654 | 81 | 260 (1%) | +/- |
| 25 | 10101 | 44 | 147305 - 151796 | 3554 | 81 | 63 (1%) | +/- |
| 4 | 9640 | 98 | 35124 - 44693 | 8521 | 83 | 125 (1%) | +/- |
Как видно из таблицы, процент identity достаточно высок, а количество гэпов не высоко. Однако у 2 выравнивания небольшое покрытие (всего 44% от контига выравнилось). Ниже представленны карты локального выравнивания для этих контигов.