Практикум 12. Предсказание генов эукариот

Квест 1. Предсказание генов некоего организма (кодовое название - X5) с помощью сервиса AUGUSTUS
Контиги сборки генома X5 лежали в ожидании своего часа на сервере kodomo. Правила игры требовали взять лишь один контиг длиной от 20 до 100 kb. С помощью программы infoseq (пакет EMBOSS) был получен список длин контигов в сборке. Следуя интуитивному предчувствию, я быстро выбрала один подходящий по длине, unplaced-307 (84991 bp). Долго задерживаться на kodomo не хотелось. Это могло быть опасно.
Итак, у меня был материал для работы. Несколько метров сплошного текста из четырех символов. И никаких зацепок. Ничего не поделаешь, придется полностью положиться на AUGUSTUS.
Как любому экстрасенсу требуется личная вещь пропавшего, чтобы настроиться на его кармический отпечаток, как они это называют, так и AUGUSTUSу необходима процедура обучения на уже известных генах данного организма. У меня не было ни известных генов, ни даже названия организма, только две жалкие буквы - x5 и фрагмент его генома. Существовал обходной путь - AUGUSTUS может использовать результаты обучения на родственном организме. Выбора не было. Пришлось устанавливать примерное таксономическое положение x5. Я обратилось за помощью к старому знакомому - blast.
Поскольку AUGUSTUSу предстояло работать с генами, большая часть которых должна была кодировать белки, имело смысл искать родственные организмы с наиболее похожими последовательностями белков и, соответственно, использовать алгоритм blastx, который ищет формальные трансляции нашей нуклеотидной последовательности в 6 рамках против базы белков. Учитывая сложную экзонно-интронную структуру генов эукариот, поиск среди готовых белков мог дать наилучшие результаты.
Итак, blastx. Сразу было понятно, что искать весь контиг unplaced-307 длиной порядка 58 kb - дело гиблое. Пришлось искать отдельные фрагменты длиной несколько тысяч п.н.
На первом же фрагменте меня ждала удача - в последовательности была найдена часть некого гена.
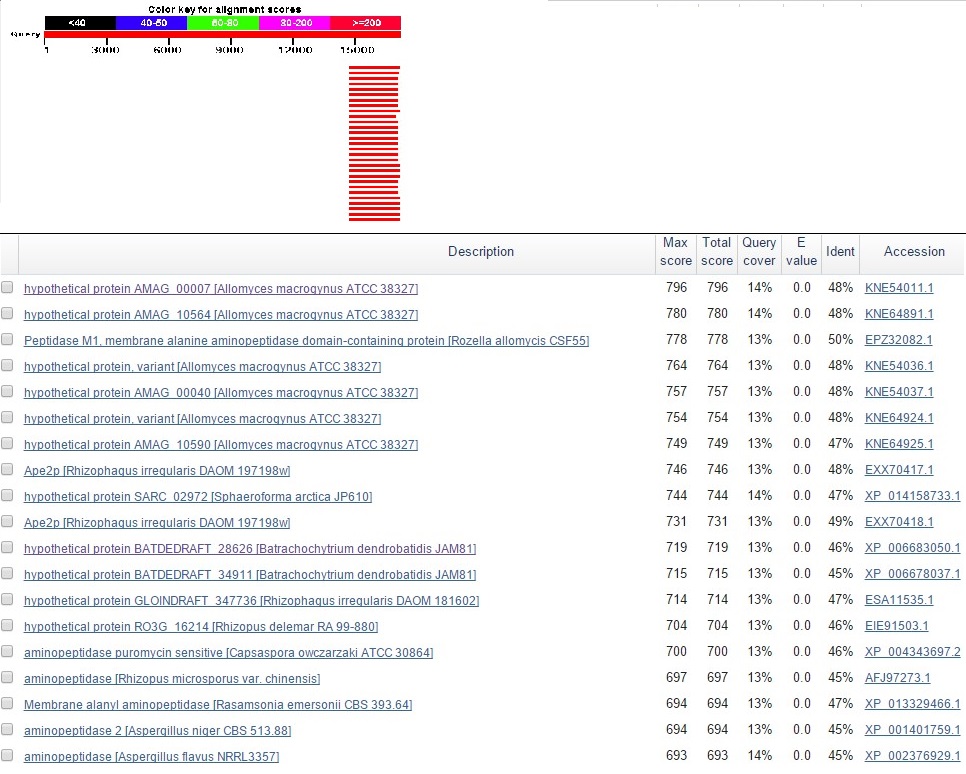
Рис.1 Результаты blastx по запросу части контига unplaced-307
Все указывало на грибы. X5 - гриб. Воистину злая шутка судьбы. Неустоявшаяся систематика, большое морфологическое и генетическое разнообразие, множество видов с неопределенным таксономическим положением - навряд ли можно найти объект для выяснения таксономии хуже, чем гриб.
Среди лучших находок по запросу первого фрагмента присутствовали:
Allomyces macrogynus: Eukaryota; Fungi; Blastocladiomycota; Blastocladiomycetes; Blastocladiales; Blastocladiaceae; Allomyces
Rozella allomycis: Eukaryota; Fungi; Cryptomycota; Rozella
Rhizophagus irregularis: Eukaryota; Fungi; Glomeromycota; Glomeromycetes; Glomerales; Glomeraceae; Rhizophagus
Sphaeroforma arctica: Eukaryota; Ichthyosporea; Ichthyophonida; Sphaeroforma
Batrachochytrium dendrobatidis: Eukaryota; Fungi; Chytridiomycota; Chytridiomycetes; Rhizophydiales; Rhizophydiales incertae sedis; Batrachochytrium.
Rhizopus delemar: Eukaryota; Fungi; Fungi incertae sedis; Mucoromycotina; Mucorales; Mucorineae; Rhizopodaceae; Rhizopus
Aspergillus niger: Eukaryota; Fungi; Dikarya; Ascomycota; Pezizomycotina; Eurotiomycetes; Eurotiomycetidae; Eurotiales; Aspergillaceae; Aspergillus
Очевидно, грибы своих не выдают. Уже после царства Fungi начинаются расхождения.
Результаты поиска еще одного фрагмента unplaced-307 были в целом похожи, только в список подозреваемых добавились еще несколько грибов из Mucoromycotina.
Среди организмов, на которых натренирован AUGUSTUS, присутствует порядка трех десятков грибов. Наиболее подходящим мне показался Rhizopus oryzae, поскольку виды рода Rhizopus неоднократно всплывали среди хороших находок по unplaced-307.
С названием Rhizopus oryzae в голове и тяжелым сердцем я отправилась к AUGUSTUS. Файл 307.fasta в качестве фрагмента генома, Rhizopus oryzae в качестве тренировочного организма, остальные параметры по умолчанию. Несколько десятков минут томительного ожидания - и я получила архив с выдачей - tar.gz.
Архив содержал следующие файлы:
augustus.gff - файл с основной информацией, а именно предсказаниями генов в формате gff, а также их предсказанными кодирующими и нуклеотидными последовательностями;
augustus.aa - предсказания генов в виде аминокислотных последовательностей в формате fasta;;
augustus.cdsexons - предсказанные экзоны в ДНК;
augustus.codingseq - предсказания генов в виде CDS (кодирующие нуклеотидные последовательности) в формате fasta;
augustus.gbrowse - gene prediction track for the GBrowse genome browser;
augustus.gtf - предсказания генов в формате gtf, менее удобном, чем gff, на мой вкус.
Далее нужно было проанализировать выдачу AUGUSTUS и проверить некоторые предсказания с помощью blast. Никому нынче нельзя доверять безоговорочно, особенно подобным программам.
Предсказания генов в формате gff содержатся в файле augustus.gff . Я не стала переделывать его в таблицу Excel, поскольку предсказанных генов оказалось довольно много, а формат кажется мне вполне удобным. Там приведены предсказанные кодирующие нуклеотидные и аминокислотные последовательности, а также экзонно-интеронная структура предсказанных генов.
Предсказания нескольких генов я проверила с помощью blastp.
Информация о выбранных генах отдельно помещена в данную таблицу . Нумерация генов совпадает с таковой из augustus.gff. Здесь я привожу только те гены, для которых были найдены достоверные гомологи.
1. Предсказанный ген 1.
Он содержит 2 экзона.
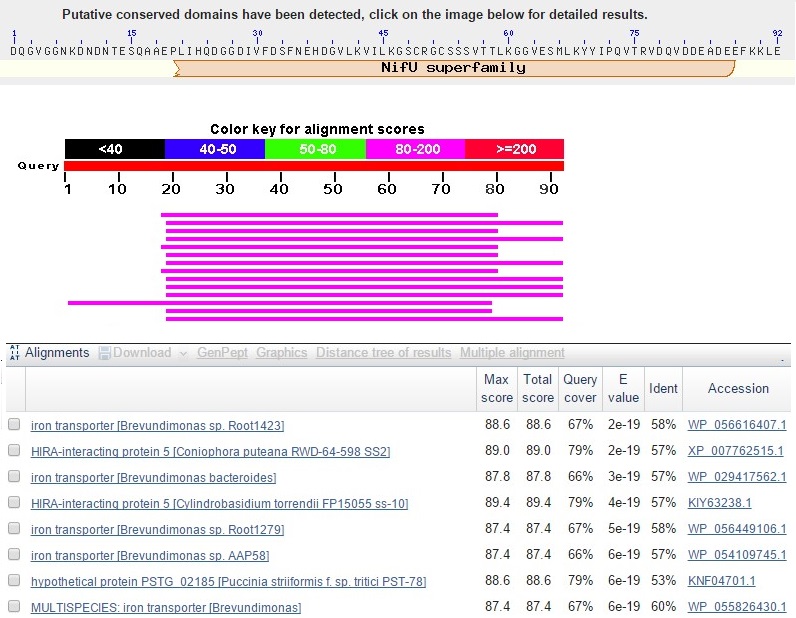
Рис.2 Результаты blastp по запросу предсказанного гена 1
Среди находок были и такие, которые соответствовали почти полноразмерной последовательности. Но - среди насекомых.
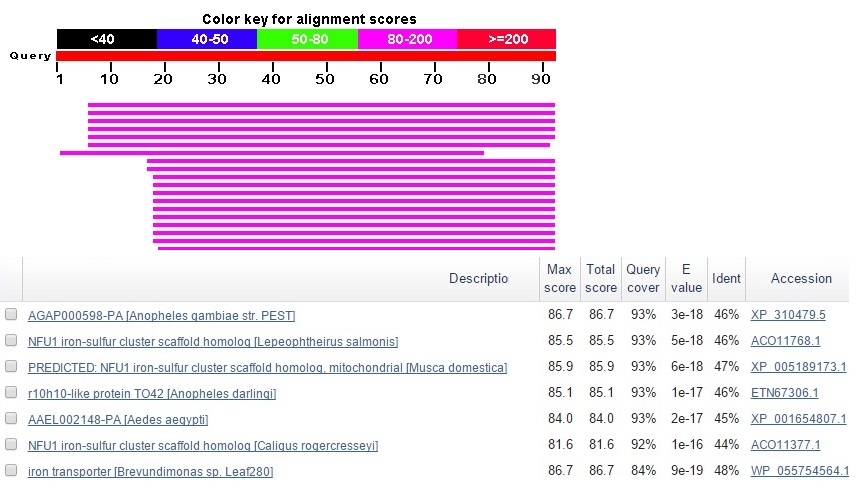
Рис.3 Результаты blastp по запросу предсказанного гена 1. Отсортированы по query cover.
Что касается начала и конца гена, находки blastp в основном начинаются позже, а заканчиваются раньше или так же. я попробовала поискать по Swiss-Prot:
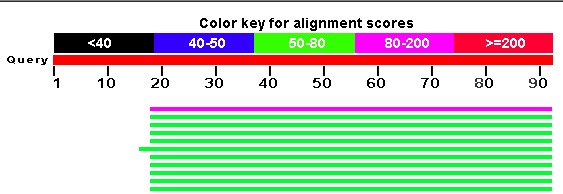
Рис.4 Результаты blastp против Swiss-Prot по запросу предсказанного гена 1. Несколько лучших находок - предшественники гомологов митохондриальных железосерных кластеров из насекомых.
Пожалуй, можно заключить, что конец гена был определен AUGUSTUS правильно, а насчет начала гена возможны всяческие спекуляции. И функции находок другие, нежели чем у лучших находок против non-redundant базы, и организмы другие - где грибы, а где насекомые.
Судя по находкам, экзонно-интронная структура была определена правильно. Также, как видно из рис.2, в структуре последовательности был найден 1 консервативный домен. Результаты blast свидетельствуют о том, что функция белкового продукта связана с железом - лучшие находки являются транспортерами железа.
2. Предсказанный ген 9.
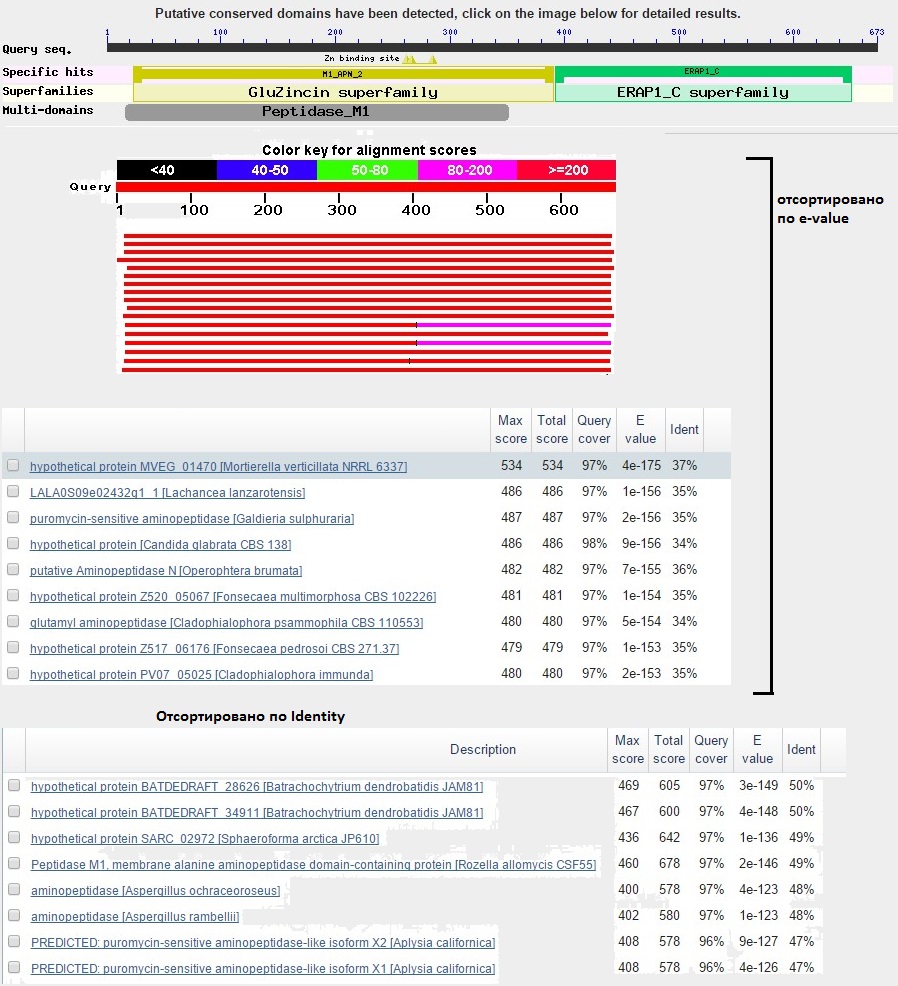
Рис.5 Результаты blastp по запросу предсказанного гена 9.
По запросу аминокислотной последовательности было найдено множество хороших гомологов, в том числе в Swiss-Prot (именно находки оттуда помогли с высокой вероятностью установить функцию белкового продукта - это, по всей видимости, аминопептидаза). Находки из SwP не содержат достаточно близких организмов - лучшая находка (и в плане предполагаемого родства организма, и по e-value) - это аминопептидаза из митохондрий S. cerevisiae. Она покрывает практически всю искомую последовательность предсказанного белка, но в выравнивании присутствует несколько крупных гэпов, то есть экзонно-интронная структура нашего предсказанного гена и данного гена дрожжей отличается. Однако не думаю, что можно безоговорочно судить о качестве предсказания нашего гена по данной находке - ее идентичность составляет 37% против 50% у некоторых других находок из nr-базы (см. ниже в тексте, а также рис. 5, сортировка по identity).
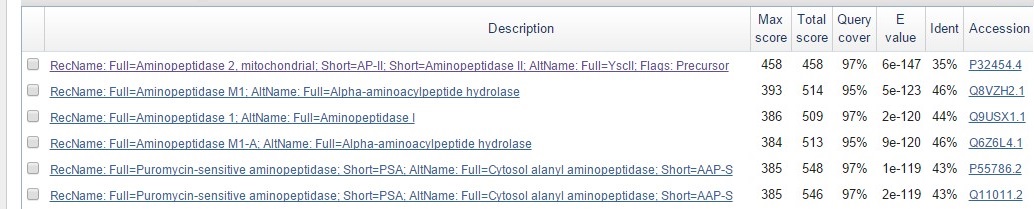
Рис.6 Результаты blastp против Swiss-Prot по запросу предсказанного гена 9.
В находках blastp против non-redundant базы присутствует множество белковых последовательностей из предположительно более родственных организмов и с лучшим значением identity (рис.5). Выравнивания первых нескольких десятков лучших находок с query выглядят примерно одинаково. Во-первых, присутствует два больших выровненных куска (соответствующих двум большим доменам, рис.5), между которыми мало похожие участки или даже гэпы в одной из последовательностей. Во-вторых, практически во всех выравниваниях в query присутствует несколько гэпов в одних и тех же местах.
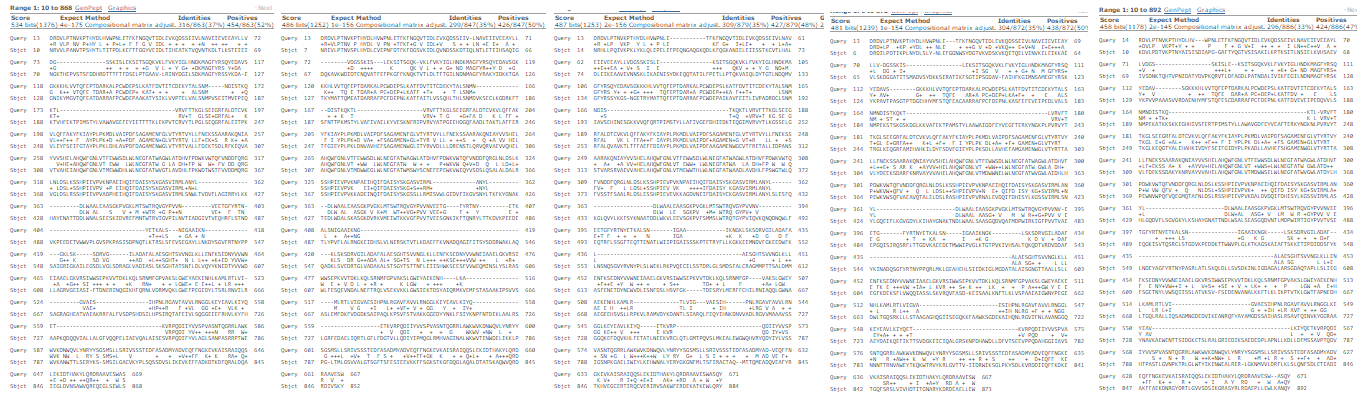
Рис.7 Выравнивания нескольких лучших находок blastp против nr по запросу предсказанного гена 9 с самим запросом. Видна похожая структура выравниваний, а именно рисунок гэпов в query.
Это может означать, что AUGUSTUS неправильно предсказал экзонно-интронную структуру данного гена. Установить точно, так ли это, из имеющихся данных я не могу.
3. Предсказанный ген 23.
Находок было много, поэтому я запускала blast только среди грибов. Что странно, при поиске по всем организмам 50 лучших находок принадлежали бактериям. Для этого гена легко удалось определить функцию белкового продукта - это 50S ribosomal protein L2. Первые несколько десятков находок blast против nr-базы и против Swiss-Prot единодушны в этом плане.
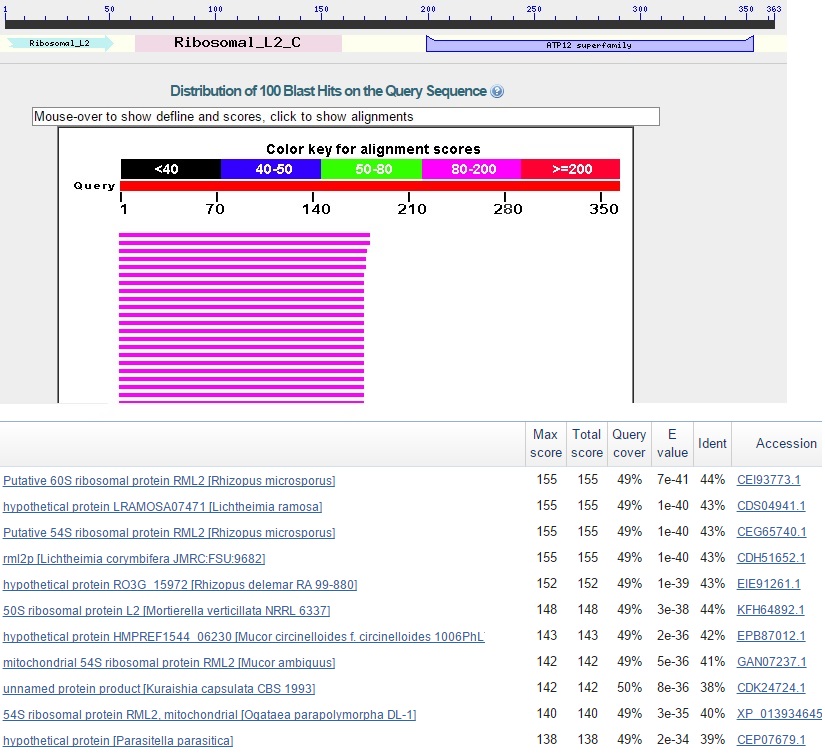
Рис.8 Результаты blastp против nr-базы по запросу предсказанного гена 23.
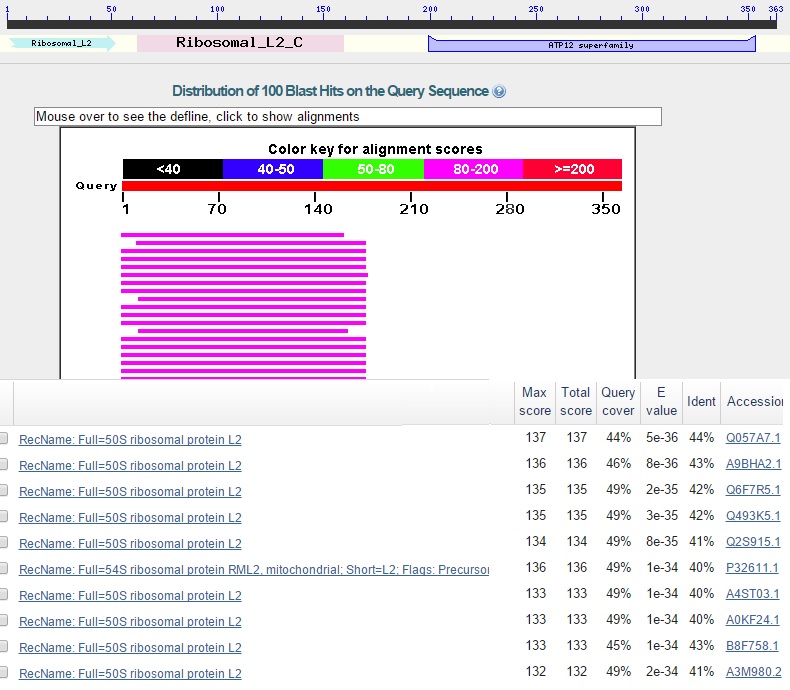
Рис.9 Результаты blastp против Swiss-Prot по запросу предсказанного гена 23.
Во второй половине белка был предположительно найден некий домен из ATP12 superfamily, однако в результатах поиска (и против nr, и против Swiss-Prot) не было ни находок с query cover > 50%, ни находок, которые бы выравнивались со второй половиной белка. Из этого можно заключить, что AUGUSTUS ошибся и белок заканчивается раньше, чем в предсказании.
Также по выравниваниям хороших находок с запросом можно судить, что AUGUSTUS не совсем точно предсказал экзонно-интронную структуру данного гена. Во всех выравниваниях в query присутствуют два гэпа в одних и тех же местах (рис.10). Вероятно, AUGUSTUS не нашел два экзона.
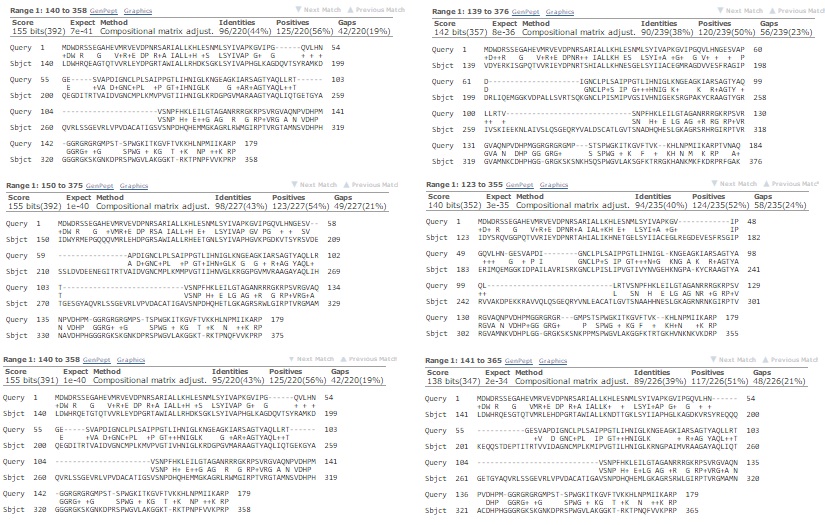
Рис.10 Выравнивания нескольких лучших находок blastp против nr по запросу предсказанного гена 23 с самим запросом. Видна похожая структура выравниваний, а именно рисунок гэпов в query.
Также я проверяла с помощью blastp предсказанные гены 2, 10 и 13. Для них достоверных находок найдено не было, из чего можно заключить, что предсказанные последовательности генами белков не являются.
Я попробовала поискать их предсказанные кодирующие ДНК-последовательности против базы Refseq РНК среди грибов - вдруг продукты функционируют в форме РНК.
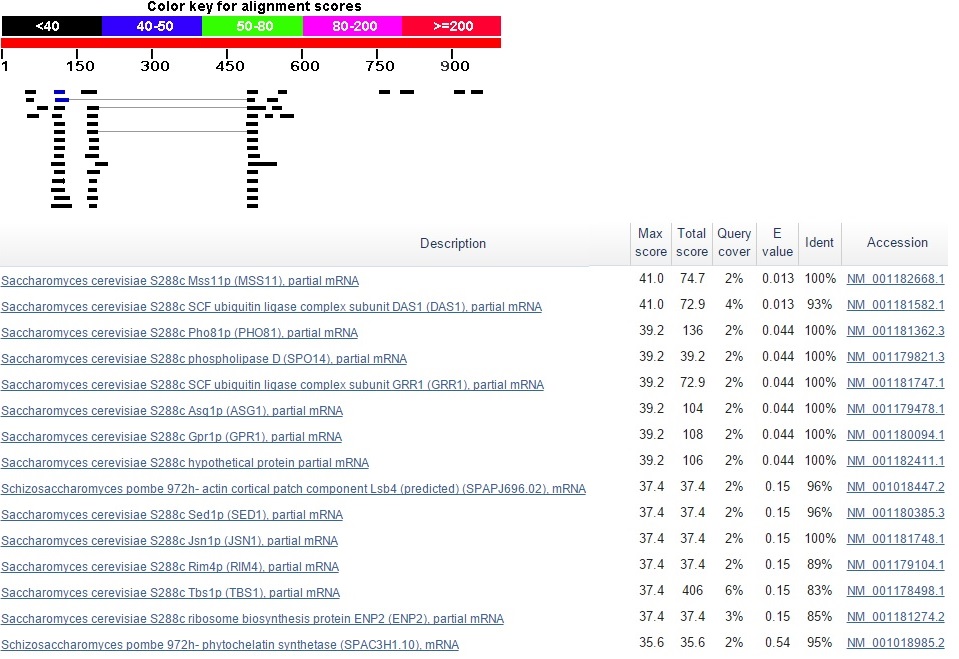
Рис.11 Результаты blastn против Refseq РНК среди грибов по запросу предсказанного гена 13.
На рис. 11 показаны результаты поиска по запросу кодирующей последовательности предсказнанного гена 13. Были найдены очень короткие фрагменты с очень высоким identity из мРНК различных белков. Такая ситуация оказалась типичной: для генов 2 и 10 результаты выглядели так же. Полагаю, найденные фрагменты, встречающиеся во многих разных РНК, - это регуляторные элементы. То есть можно заключить, что нет никаких свидетельств в пользу того, что гены 2, 10 и 13 кодируют какие-то функциональные РНК или белки. Значит, эти предсказанные последовательности, вероятно, не соответствуют никаким реально существующим генам, и AUGUSTUS ошибся с предсказанием.
Квест 2. Сравнение аннотации Refseq и AUGUSTUS одного гена человека
Я выбрала ген карнитин-пальмитоилтрансферазы 2 (carnitine palmitoyltransferase 2 [Homo sapiens]) путем случайного тыка в NCBI Genes, организм H.sapiens. Затем я нашла этот ген в UCSC Genome Browser, используя последнюю сборку генома hg38.
Координаты гена: хромосома 1: 53,196,859 - 53,202,736
Расположен на прямой цепи
Далее требовалось сравнить аннотации Refseq и AUGUSTUS. Для этого я оставила только три трэка: base position, Refseq и AUGUSTUS. С помощью меню Tools => Table Browser получила интрон-экзонную структуру в текстовом виде.
Результаты:

Рис.12 Изображение интрон-экзонной структуры гена carnitine palmitoyltransferase 2 согласно предсказаниям Refseq и AUGUSTUS из UCSC Genome Browser
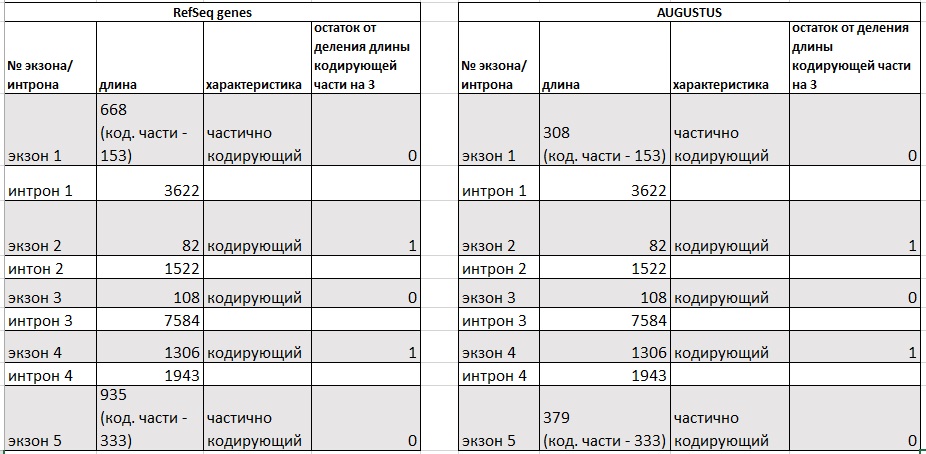
Рис.13 Сводные таблицы интрон-экзонной структуры гена carnitine palmitoyltransferase 2 согласно предсказаниям Refseq и AUGUSTUS
Данные, представленные на рис. 13, продублированы в таблице, приведенной выше в задании 1.
Как видно из рисунков 12 и 13, аннотации Refseq и AUGUSTUS расходятся только в описании некодирующих регионов первого и последнего экзонов. Остальные данные идентичны. Что странно, суммарная длина кодирующих частей экзонов не делится на 3 нацело. Я несколько раз перепроверила числа, однако проблема сохранилась. Не знаю, чем это можно объяснить. Разве что неточностью обеих аннотаций.