Практикум 15.
Задание 1. Пакетом velvet cобрать из всех чтений своего набора (прошедших очистку) контиги без использования референса.
Пакет velvet стоит на kodomo и состоит из двух программ: velveth для выделения k-меров из чтений ("hashing") и velvetg для сборки контигов на основе графа k-меров.
Использовавшиеся команды для velvet:
velveth vel 31 -fastq 9_2_imp.fastq
velvetg vel
Соответственно, была взята длина k-мера, равная 31. Я пробовала ее изменять, результаты чего приведены ниже, в конце отчета.
Промежуточные результаты:
в финальном графике 112 узлов (т.е. собрано 112 контигов)
N50 = 270
максимальная длина контига = 1018
Задание 2. Программой blastn (алгоритм megablast) сравнить получившиеся контиги с последовательностью хромосомы.
Использовавшиеся команды:
makeblastdb -in /nfs/srv/databases/ngs/Human/chr9.fasta -dbtype nucl
blastn -db chr9 -query vel/contigs.fa -outfmt 6 -out 92bl
Результаты сборки и картирования контигов
Файл с контигами, полученный в результате работы velvet
Результаты поиска blastn представлены в таблице.
В этом файле представлено несколько таблиц: исходная со всеми картировавшимися контигами, только с контигами, картировавшимися на одно место (это и есть результаты для проверки), таблица, иллюстрирующая странную ситуацию с "контигами длины 31" (см. ниже в отчете).
Анализ результатов
В исходной таблице присутствуют контиги, картировавшиеся на одно место и на несколько мест, а также более сложные случаи. Контиги, не единственным образом картированные на хромосому, по-видимому, представляют из себя повторы.
NB! Названия контигов из файла contigs.fa, полученного в результате работы velvet, имеют в составе длину (length_*). Однако это указанное значение длины на 30 меньше реальной длины последовательности, так что, видимо, это количество k-меров.
Более сложные случаи:
После сборки программой velvet оказалось, что в файле contigs.fa присутствуют контиги с разными названиями, последовательности которых отличаются очень незначительно. Например, контиги node_8 и node_10
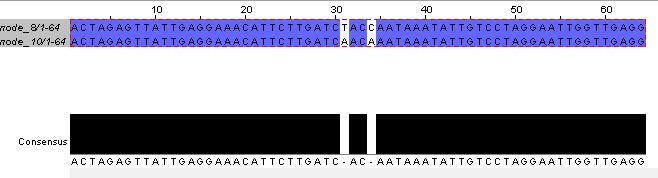
Рис. № Выравнивание контигов node_8 и node_10 из contigs.fa
После работы blastn node_8 и node_10 картировались на одно и то же место. При этом последовательность контига 8 полностью совпала с последовательностью хромосомы в этом месте, а у контига 10 обнаружилось 2 SNP. Возможно, в этом случае мы имеем дело с гетерозиготными SNP. То же самое и с контигами node_2 и node_4. Для подобных случаев перекрытие последовательностей в таблице я не указывала.
На самом деле ситуация еще более сложная. В файле с контигами, полученном после работы velvet, более половины контигов в названии имеют словосочетание length_31 (или 30, 34, 32, но таких меньше). Я заметила, что именно эти контиги имеют перекрывания друг с другом (см. соответствующий лист из прикрепленной таблицы), причем длина перекрывания чаще всего составляет 30 п.н. Я попыталась понять, откуда берутся эти перекрывания и почему velvet не смогла объединить перекрывающиеся контиги в более крупный контиг.
В случае контигов NODE_18 и NODE_20 наблюдается перекрывание в 38 п.н.
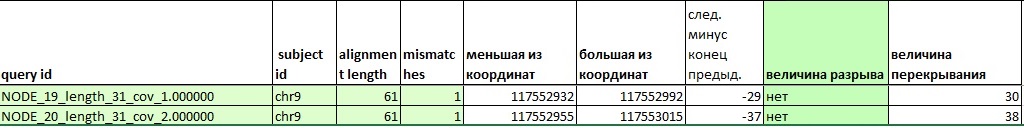
Я проверила, что представляют из себя их последовательности, и построила выравнивание с помощью ресурсов JalView. Оказалось, что в перекрывающемся участке есть 2 SNP.

Рис. № Выравнивание контигов node_ 19 и node_20
Для другой пары контигов, node_9 и node_14, с перекрыванием 30, оказалось, что перекрывающиеся участки полностью идентичны:

Рис. № Выравнивание контигов node_ 9 и node_14
Однако программа не смогла объединить их. Я проверяла также пары 90 и 91, не объединенные программой, и все они имеют участки перекрывания длиной около 30 п.н.
Более того, есть даже такие контиги, не объединенные программой, где участки перекрывания составляют больше 50 п.н. (например, 57 для контигов 104 и 108).

Рис. № Выравнивание контигов node_ 9 и node_14
Судя по всему, дело в несовершенстве программы velvet.
При работе с velvet я пробовала менять длину k-меров и минимальную длину контигов. Результаты:
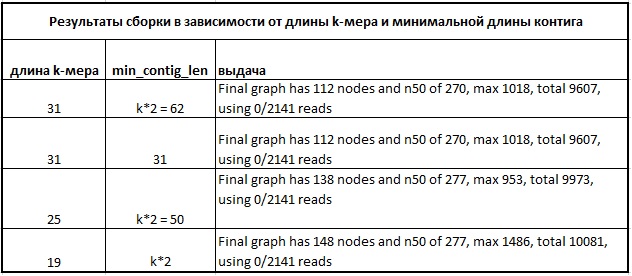
Рис. № Изменение результатов работы velvet в зависимости от параметров
Видно, что при уменьшении минимальной длины контига ничего не изменилось. При уменьшении длины k-мера количество контигов увеличилось, то есть результат ухудшился. Для длины k-мера = 19 я запускала blastn по хромосоме 19, при этом находки были довольно плохие и содержали в среднем по 5-10 несовпадений против 0-1 для правильно собранных контигов (при длине k-мера = 31). Очевидно, что такая сборка может считаться очень плохой.