Практикум 8. Нуклеотидный blast
Задание 2. Сравнение списков находок, полученных 3-я разными алгоритмами blast: blastn, megablast и discontiguous megablast.
Три алгоритма blast - blastn, megablast и discontiguous megablast - оптимизированы для поиска в разных условиях. Blastn ищет все последовательности, хоть в чем-то схожие с данной, megablast - очень похожие последовательности, discontiguous megablast - похожие, но не очень сильно.
Последовательность нужно было взять из практикума 7. Скачать данную последовательность в формате fasta
Требовалось в зависимости от находок blastn (4-е задание практ.7) ограничить область поиска подходящим таксоном.
Вначале я попробовала запустить blastn с ограничением области поиска надотрядом Eumalacostraca - самым низким таксоном, который мне удалось установить для данной последовательности в предыдущем практикуме.
Было выдано 2472 находки. В задании требовалось подобрать условия таким образом, чтобы находок было 10-1000, поэтому я изменила условия.
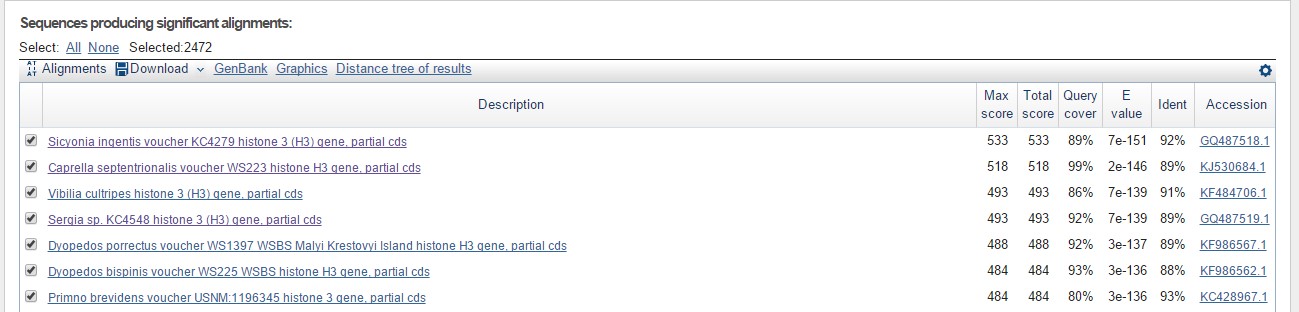
Рис. 1 Результаты работы blastn по поиску данной последовательности с ограничением области поиска надотрядом Eumalacostraca
Далее я пробовала искать внутри таксонов Eucarida (2088 находок), Decapoda (2084 находок), Dendrobranchiata (46 находок). Я решила остановиться на поиске внутри Dendrobranchiata.
Я запустила поиск по данной последовательности и с данным ограничением в blastn, megablast и discontiguous megablast.
Результаты:
Все достоверные находки - это части гена гистона 3.
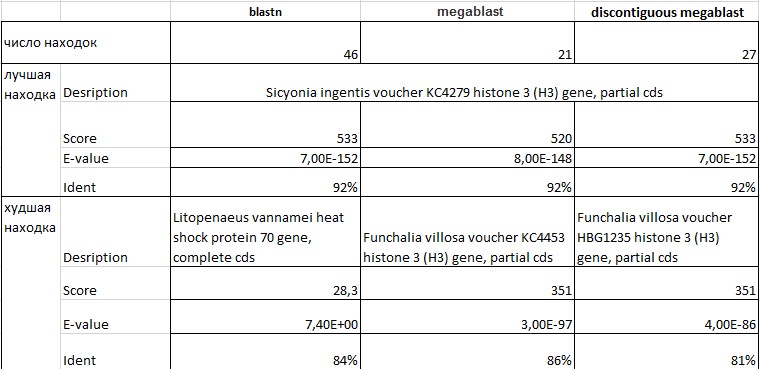
Рис. 2 Результаты работы blastn, megablast и discontiguous megablast по поиску данной последовательности с ограничением области поиска надотрядом Dendrobranchiata
Blastn нашел 27 последовательностей с E-value < e-4 и query cover > 75%. Оставшиеся находки явно не представляют интереса, поскольку имеют query cover <= 5%.
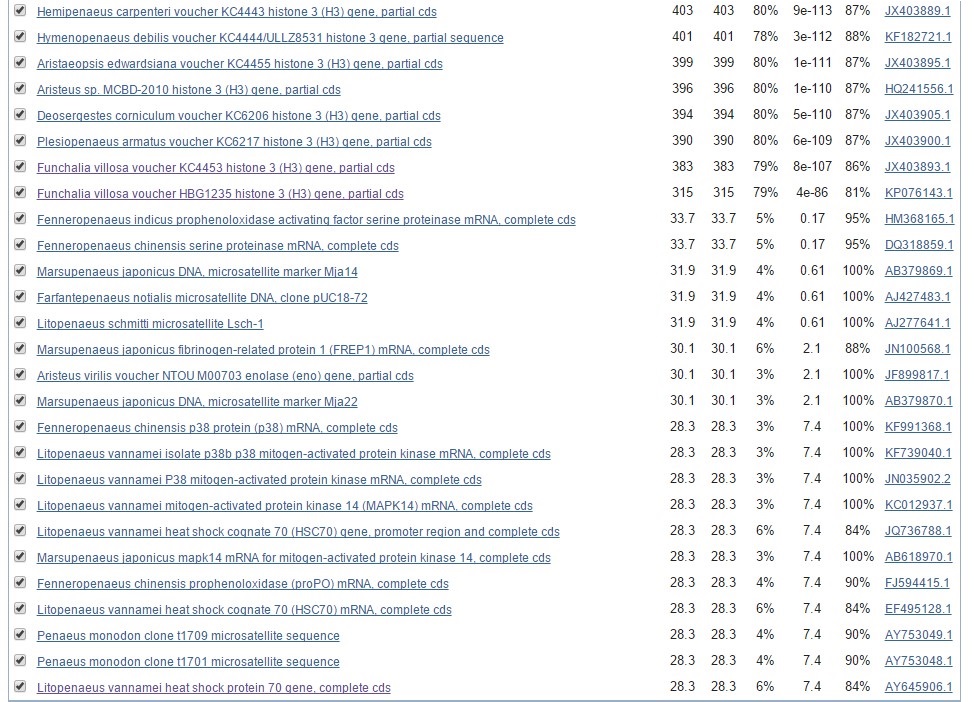
Рис. 3 Худшие находки blastn. Поиск данной последовательности с ограничением области поиска надотрядом Dendrobranchiata.
Список достоверных находок blastn совпадает с выдачей discontiguous megablast, который содержит только достоверные находки.
Выдача megablast содержт 21 результат, все находки достоверные. При этом этих достоверных находок на 6 меньше, чем в выдаче discontiguous megablast.
Отсутствует, например, находка Hymenopenaeus debilis voucher KC4444/ULLZ8531 histone 3 gene, partial sequence; Score = 401, E-value = 3e-112, Identity = 88%.
Алгоритм megablast не нашел несколько хороших последовательностей из-за того, что оптимизирован для поиска очень похожих последовательностей, поэтому может упускать не очень похожие.
Blastn, наоборот, нашел много лишнего. Можно заключить, что в данных условиях поиска оптимальным алгоритмом является discontiguous megablast. Это и понятно, ведь мы искали один и тот же ген у организмов внутри одного подотряда.
Задание 3. Проверить наличие гомологов пяти белков в геноме одного из организмов
Задание 3.1
Требовалось взять организм из задания 1 практикума 7 - это Apis mellifera - и проверить наличие у него гомологов следующих пяти белков: HSP7C_HUMAN, TERT_HUMAN, CISY_HUMAN, RPB1_HUMAN, PABP2_HUMAN.
Для выполнения задачи использовался tblastn, то есть поиск белка в базе транслированных в 6-ти рамках нуклеотидных последовательностей. Область посика была ограничена организмом Apis mellifera.
Вначале для каждого белка была найдена запись в Uniprot, сохранены их AC и fasta-последовательности. Затем проведен поиск с помощью tblastn. На вход blast подавался AC белка.
Результаты:
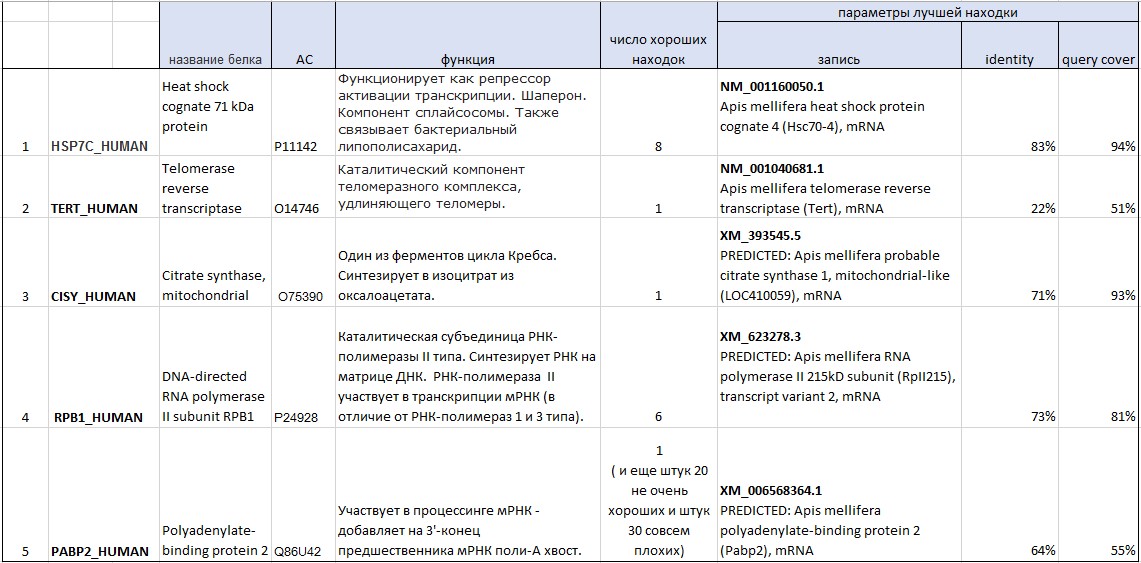
Рис.4 Результаты поиска гомологов пяти белков.
-
1) HSP7C_HUMAN - Heat shock cognate 71 kDa protein
По этому запросу было выдано 8 находок, все достоверные (с хорошим E-value)
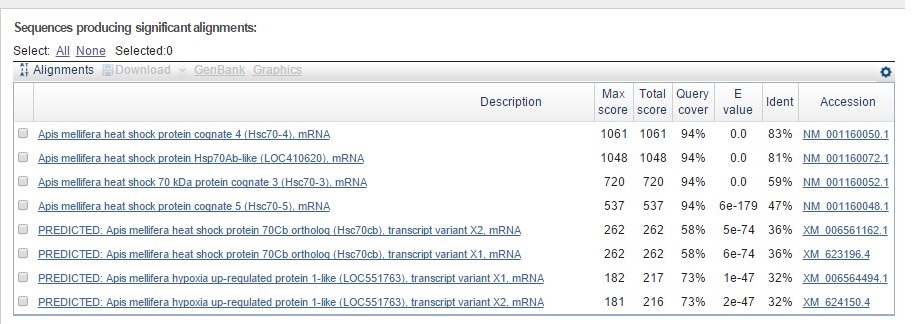
Находки из Apis mellifera по запросу P11142
Судя по названиям,первые 6 находок являются гомологами нашего белка. Две последние имеют уровень идентичности (32%), сравнимый тем, что имеют 5-я и 6-я. Поэтому, думаю, можно предположить, что 7-я и 8-я находки также гомологичны нашему белку.
В выдаче присутствуют 4 гомолога нашего белка, в том числе Heat shock protein (и три его гомолога). Также есть 2 варианта транскрипта предсказанного ортолога искомого белка. Две последних находки, варианты транскрипции некоего предсказанного гена регулятора гипоксии, выглядят достаточно хорошо, чтобы предполагать их гомологию с нашим белком.
-
2) TERT_HUMAN - Telomerase reverse transcriptase
Было найдено 11 последовательностей. Одна и есть теломераза пчелы, остальные - недостоверны.
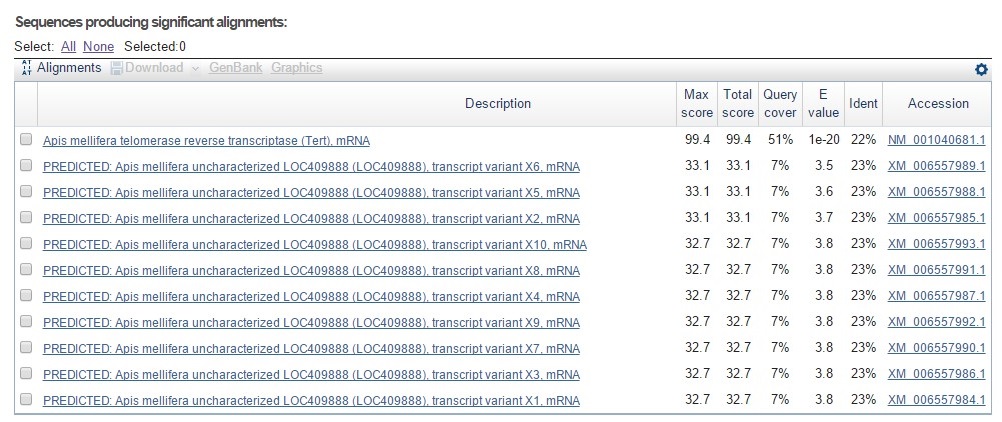
Находки из Apis mellifera по запросу O14746
-
3) CISY_HUMAN - Citrate synthase, mitochondrial
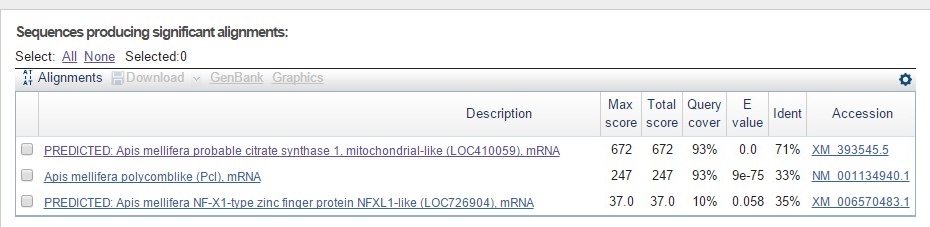
Найдено 3 последовательности, 2 из них имеют низкий E-value (достоверны). Лучшая находка - это мРНК предсказанного искомого белка пчелы (PREDICTED: Apis mellifera probable citrate synthase 1, mitochondrial-like (LOC410059), mRNA). Есть еще одна достоверная находка - мРНК белка policomblike, то есть, судя по всему, похожего на белки семейства polycomb. Они способны ремоделировать хроматин, в частности, у дрозофил они участвуют в регуляции гомеозисных генов. Перекрывание последовательностей искомого и найденного белка высокое (93%), Identity последовательностей - 33%. Вроде бы как эти данные свидетельствуют о гомологии, но функции белков разные, и идентичность низкая.
-
4) RPB1_HUMAN - DNA-directed RNA polymerase II subunit RPB1
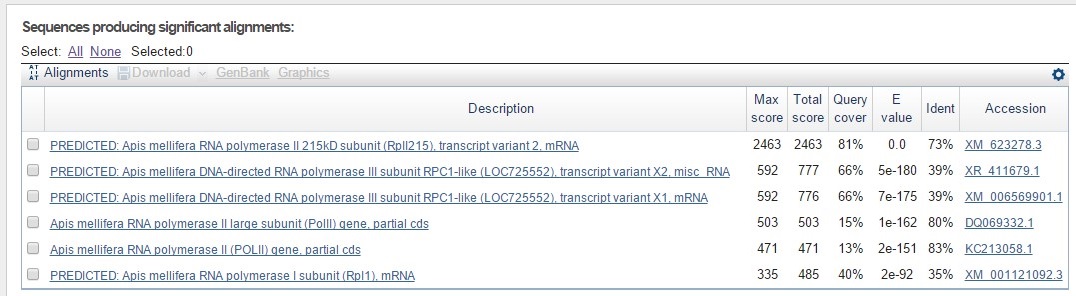
Найдено 6 последовательностей, все они кодируют субъединицы РНК-полимераз I, II и III. РНК-полимеразы всех трех типов похожи, имеют приблизительно одинаковые задачи и механизмы работы и явно гомологичны, поэтому неудивительно, что эти последовательности были найдены по данному запросу. Лучшая находка - предсказанный вариант транскрипта Apis mellifera RNA polymerase II 215kD subunit (RpII215).
-
5) PABP2_HUMAN - Polyadenylate-binding protein 2
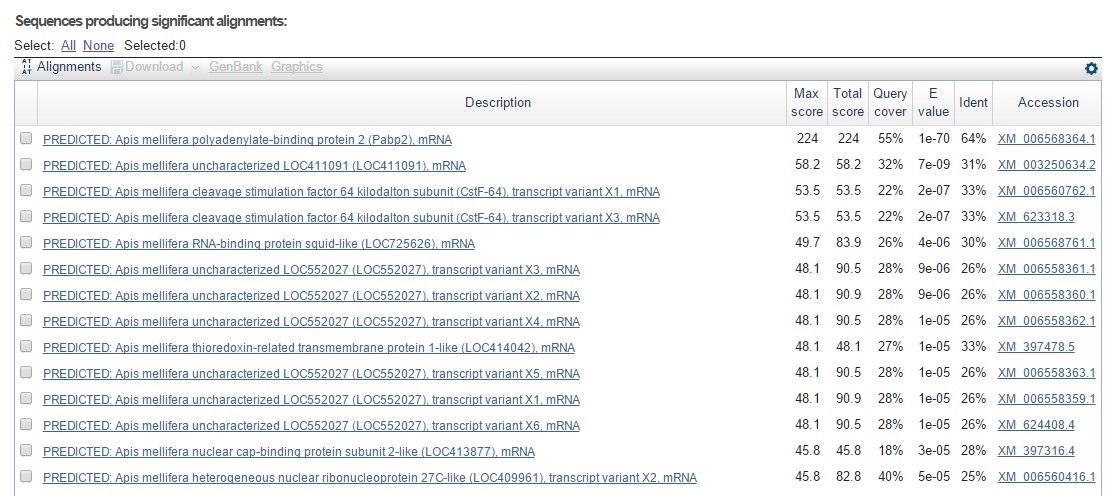
Было найдено 53 последовательности. Лучшая находка - мРНК пчелиного ортолога искомого белка. Остальные последовательности имеют в разной степени меньшее покрытие и идентичность и соответствуют разным белкам, среди которых много РНК-связывающих. Эти находки можно объяснить тем, что во всех этих белках присутствуют домены, имеющиеся в искомом белке ( и выполняющие те же функции).
Задание 4. Классифицировать геномы родственных вирусов по сходству последовательностей
Для вируса Pea enation mosaic virus-2 были найдены родственники. Это было сделано с помощью сайта http://www.ictvonline.org/, посвященного таксономии вирусов.
Страница с таксономией выбранной группы вирусов
Были выбраны следующие вирусы:
Pea enation mosaic virus-2, Carrot mottle virus, Tobacco bushy top virus , Velvet tobacco mottle virus, Groundnut rosette virus, Carrot mottle mimic virus.
Их полные геномы были скачаны и сохранены в одном файле viruses.fasta. Далее я работала в локальном blast.
1) Создала базу данных: makeblastdb -in viruses.fasta -dbtype nucl
2) Запустила tblastn.
3) Несколько раз запустила скрипт с разными параметрами, получила таблицы.
4) Обработала эти таблицы в Excel.
Финальный файл с обработанной выдачей tblastx всех против всех + матрицы сравнений геномов
Я решила анализировать результат работы скрипта с такими параметрами: min aln_len =100, min identity =25, max e-value =0.0001. Так отсеялись недостоверные находки, находки "сам-против-себя" и слишком короткие последовательности.
Таблицы я выровняла по query_id, затем по subject_id, получила блоки находок для двух геномов. Для этих блоков я создала матрицы значений некоторых параметров:
суммы Identity*aling_len/1000 (как характеристики числа совпадающих позиций)
среднего процента сходства всех трансляций (средней identity)
максимальной длины сходных белковых последовательностей (суммарной aln_len)
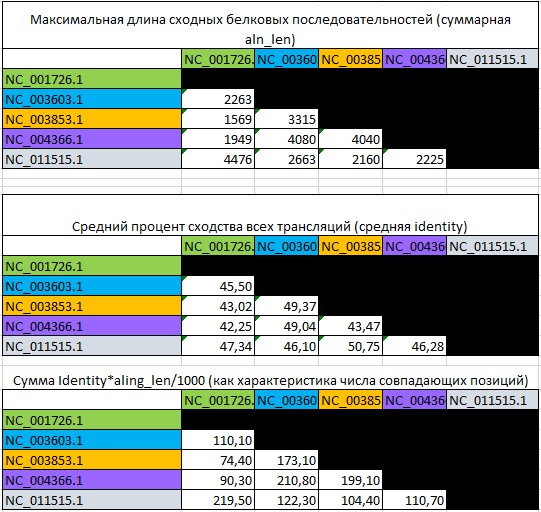
Рис.5 Матрицы некоторых параметров сходства геномов данных вирусов
Из общей таблицы сразу видно, что достоверных находок последовательностей из генома Velvet tobacco mottle virus (NC_014509.2) против геномов других вирусов нет. В необработанной выдаче blast (см. соответствующий лист excel-файла) находки последовательностей NC_014509.2 относительно других геномов присутствуют, но недостоверные (слишком короткие случайные последовательности). В выдаче, обработанной скриптом, находок из NC_014509.2 нет. Их отсутствие свидетельствует о том, что Velvet tobacco mottle virus, по-видимому, не родственен остальным пяти вирусам. Это очень странно, поскольку я брала вирусы одной таксономической группы (Umbraviruses) опираясь на данные сайта http://www.ictvonline.org/. Почему так - вопрос остается открытым. Пришлось изъять Velvet tobacco mottle virus из дальнейшего изучения сходства геномов.
Далее я анализировала оставшиеся 5 геномов.
Мне показался наиболее инфомативным параметр суммы Identity*aling_len/1000, условно отражающий количество совпадающих позиций в геноме. Надо сказать, что сходство геномов исходя из этого параметра близко к таковому, исходя из максимальной длины сходных белковых последовательностей.
Чтобы приблизительно классифицировать геномы по сходству, я построила дерево геномов исходя из матрицы значений параметра суммы Identity*aling_len/1000, пользуясь алгоритмом невзвешенного попарного среднего (UPGMA). Последовательные стадии построения дерева приведены в excel-файла на листе UPGMA.
Результат:
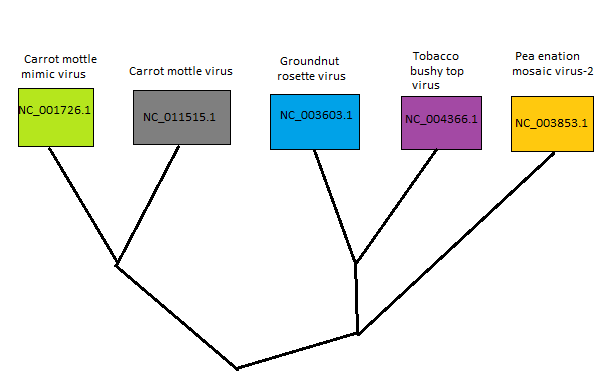
Рис.6 Дерево, характеризующее сходство геномов данных вирусов
Итоговые результаты:
1) Не выявлено гомологии генома Velvet tobacco mottle virus геномам других пяти вирусов (Pea enation mosaic virus-2, Carrot mottle virus, Tobacco bushy top virus, Groundnut rosette virus, Carrot mottle mimic virus).
2) Геномы этих пяти вирусов гомологичны и в разной степени сходны между собой.
3) По грубой оценке (по условному количеству совпадающих позиций в геноме) более всего схожи между собой пары Carrot mottle virus и Carrot mottle mimic virus, Tobacco bushy top virus и Groundnut rosette virus.
4) Pea enation mosaic virus-2 больше похож на Tobacco bushy top virus и Groundnut rosette virus, чем на Carrot mottle virus и Carrot mottle mimic virus.