Практикум 9. EMBOSS
Упражнения
1) (seqret) Несколько файлов в формате fasta собрать в единый файл
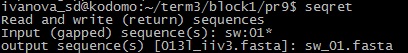
(Из SwissProt скачиваются все файлы, которые начинаются на 01, и помещаются в один файл.
Полученный файл3) (seqret) Из файла с хромосомой в формате .gb вырезать три кодирующих последовательности по указанным координатам "от", "до", "ориентация" и сохранить в одном fasta файле
4) (transeq) Транслировать кодирующие последовательности, лежащие в одном fasta файле, в аминокислотные, используя указанную таблицу генетического кода. Результат - в одном fasta файле.
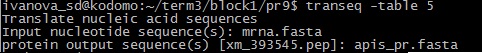
На вход подана нуклеотидная последовательность мРНК митохондриальной цитрат-синтазы 1 пчелы медоносной. Соответственно, выбрана таблица генетического кода №5 (для митохондрильных генов беспозвоночных). Программа транслирует последовательность начиная с первого нуклеотида (а не ищет старт-кодон), поэтому все транслировалось неправильно, со множеством стоп-кодонов.
5) (transeq) Транслировать данную нуклеотидную последовательность в шести рамках.
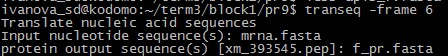
В выходном файле 6 последовательностей, как и должно быть.
10) (shuffle) Перемешать буквы в данной нуклеотидной последовательности
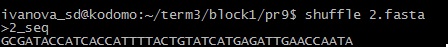
Путем перемешивания старой последовательности получена новая.
Практикум 9. EMBOSS
Задание: сравнить аннотации генов белков в одной хромосоме археи с трансляциями длинных открытых рамок считывания
Аннотация SNP
Я выбрала свою архею из первого семестра - Picrophilus torridus DSM 9790 и сохранила последовательность ее единственной хромосомы в формате genbank.
Пункт 1. Получение списка трансляций открытых рамок с помощью команды getorf пакета EMBOSS
1. Для этой задачи требовалось использовать программу getorf, прописав следующие опции:
таблицу генетического кода для данного генома (можно посмотреть в записи хромосомы): -table 11
минимальную длину открытой рамки - 180 п.н.: -minsize 180
кольцевая или линейная хромосома: кольцевая, -circular
выходные последовательности - трансляции открытых рамок от стоп кодона до стоп кодона: -find 0
Итого:
getorf -table 11 -minsize 180 -circular -find 0 PTgen.fasta out.fasta
2. Далее нужно было получить список координат и ориентаций найденных открытых рамок с помощью infoseq
Для infoseq использовались параметры: -name (получение ID открытой рамки), -description (координаты в геноме и др.), -sprotein1 - length (длина трансляции в остатках).
Итого:
infoseq -only -name -sprotein1 -length -description out.fasta > orf1.txt
Пункт 2. Получение таблицы аннотированных генов белков
Я не нашла предлагаемого в задании файла .ptt, поэтому скачала таблицу с необходимыми данными, перейдя по ссылке Genes со страницы генома своего организма на NCBI и поставив отметку напротив Protein-coding.
Далее я свела обе таблицы в одну. Скачать таблицу
Примеры антипараллельных открытых рамок с пересечением >= 150 п.н.
В таблице, полученной с помощью getorf, оказалось множество примеров больших пересечений открытых рамок считывания, как на одной цепи, так и на противоположных. То есть, судя по всему, подобные пересечения ORF в геноме не редкость. Из примерно 6050 найденных ORF 2350 (то есть больше трети) имели перекрытие > 150 п.н.
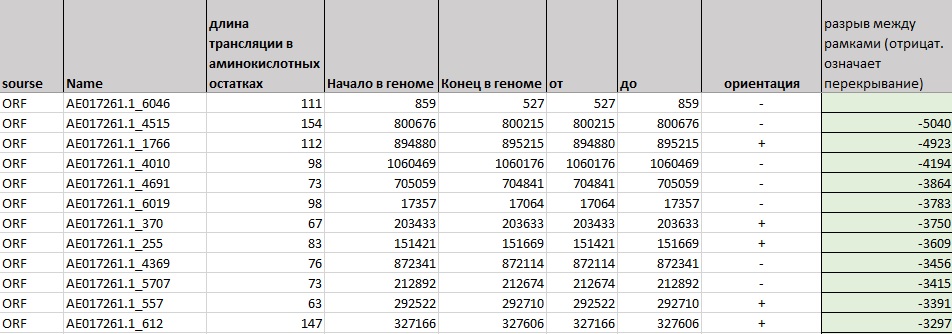
Рис. 1 Верхушка таблицы, полученной с помощью getorf. Применена сортировка по условной величине разрыва ("от" следующей ORF минус "до" предыдущей, значения закреплены за следующей ORF, то есть в ячейке приведено готовое посчитанное значение, а не формула с ссылкой на какие-то ячейки). Примечание: стоит заметить, что эта сортировка условна, поскольку теряется порядок найденных рамок, а вместе с ним и смысл значения разрыва. Однако она позволяет посмотреть, какие длины разрывов вообще есть и как они распределены по величине, а затем можно восстановить последовательность рамок.
Однако это вовсе не обязательно означает, что реально существующие гены действительно пересекаются. У моей археи в таблице с аннотированными генами больших пересечений между границами генов не обнаружилось. Наибольшее пересечение = 83 п.н.
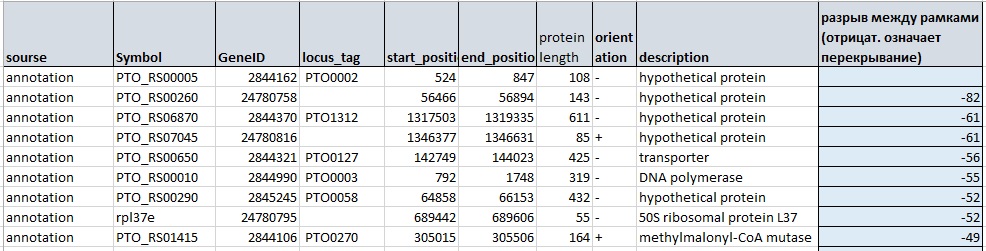
Рис. 2 Верхушка таблицы с аннотациями генов, применена сортировка по условной величине разрыва ("от" следующего гена минус "до" предыдущего). Видно, что величина перекрывания генов не превышает 100 п.н.
Я все же решила посмотреть поближе на несколько мест пересечений из первой таблицы (полученной с помощью getorf, рис.1).
1) Пара рамок AE017261.1_1616 и AE017261.1_4515
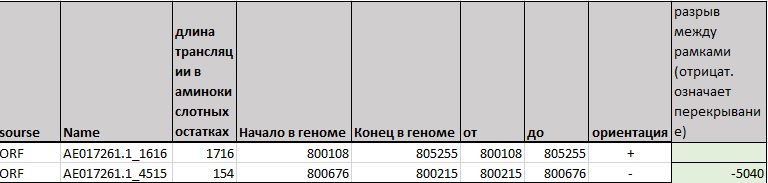
Рис. 3 Пара рамок AE017261.1_1616 и AE017261.1_4515
Оказалось, что в этом месте в ДНК присутствует множество ORF на обеих цепях и в разных рамках считывания (судя по тому, что отличаются позиции не только старт-, но и стоп-кодонов. Однако в данном регионе есть расоложен только 1 ген, то есть какого-либо перекрытия генов нет.
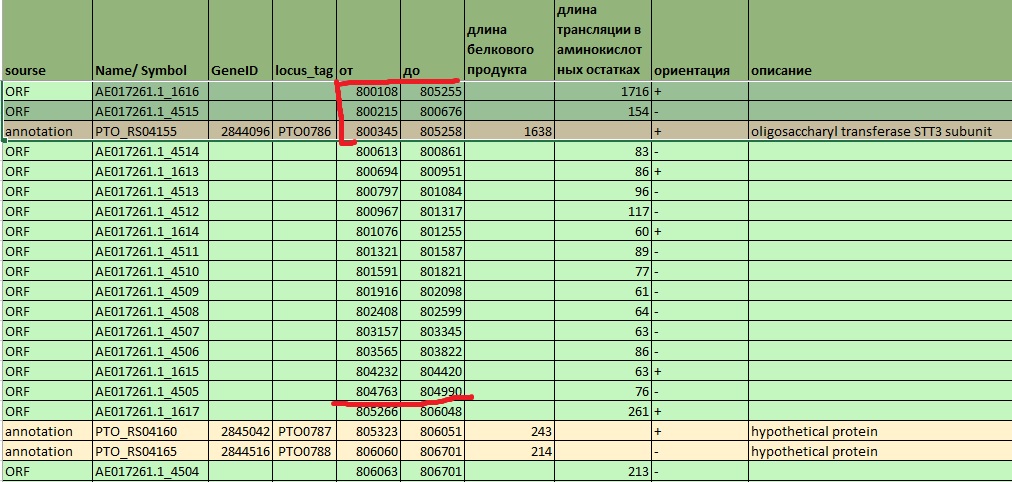
Рис. 4 Существующие гены и найденные ORF неподалеку от AE017261.1_1616 и AE017261.1_4515
2) Пара рамок AE017261.1_4328 и AE017261.1_1766
Этот случай практически аналогичен предыдущему, кроме того факта, что несколько вариантов рамок присутствует только на одной цепи.
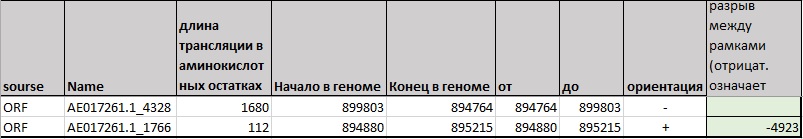
Рис. 5 Пара рамок AE017261.1_4328 и AE017261.1_1766
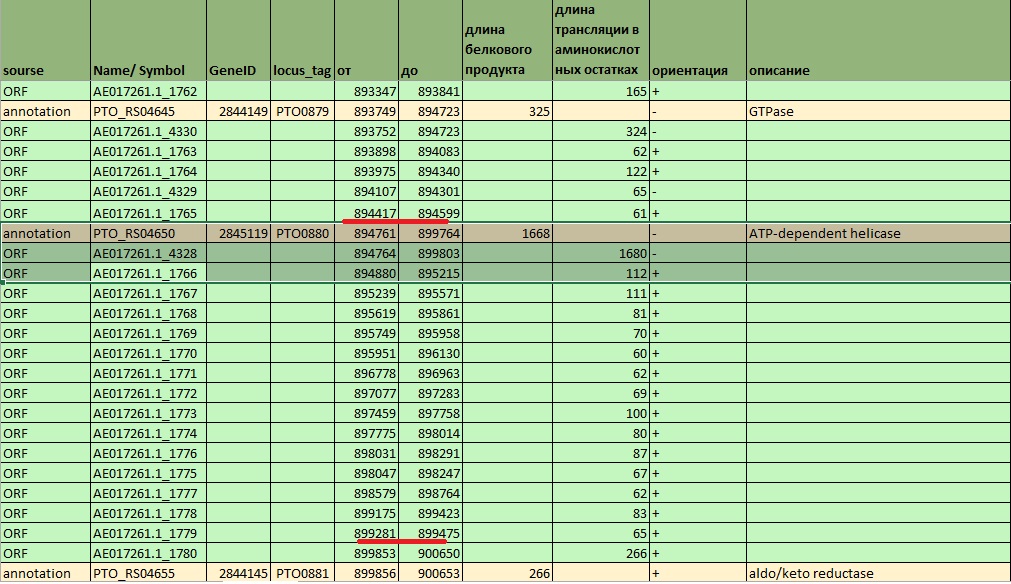
Рис. 6 Существующие гены и найденные ORF неподалеку от AE017261.1_4328 и AE017261.1_1766
Примеры расхождений в таблицах
Во-первых, сразу видно, что аннотации генов и предсказания getorf расходятся во мнении относительно того, включать ли стоп-кодон в, соответственно, ген или ORF. В открытую рамку считывания стоп кодон не включается, в отличие от гена, поэтому предсказания ORF заканчиваются на 3 п.н. раньше, чем аннотации генов.
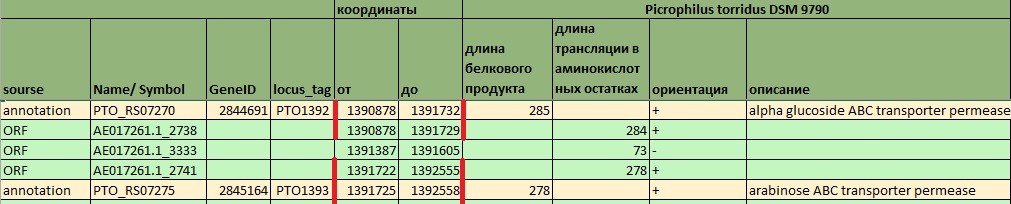
Рис.7а Предсказания ORF заканчиваются на 3 п.н. раньше, чем аннотации генов. У второй пары ген - предсказанная ORF различаются также и старт-кодоны. NB! Странно, что что у первой пары разная длина трансляции, а именно, различающаяся на 1, ведь стоп-кодон не транслируется, а старт-кодоны у них совпадают.
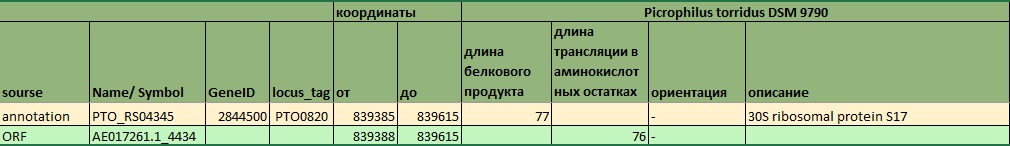
Рис.7б Предсказания ORF заканчиваются на 3 п.н. раньше, чем аннотации генов. (Обе структуры расположены на минус-цепи).
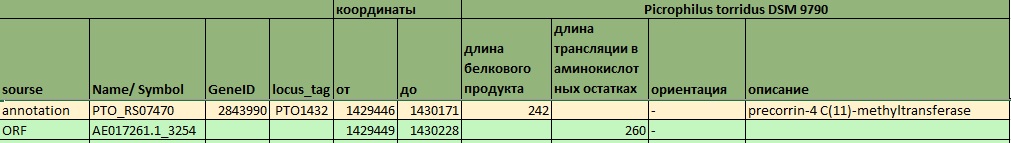
Рис. 8 Предсказания ORF заканчиваются на 3 п.н. раньше, чем аннотации генов. Обратите внимание, что ген и рамка расположены на минус-цепи, соответственно стоп кодон ("от") в данной нумерации располагается раньше старт-кодона ("до"). Позиции старт-кодонов в данном случае тоже различаются.
Во-вторых, исчезающе мало оказалось примеров полного совпадения координат аннотированных генов и предсказанных ORF ( не учитывая описанного выше отличия на 3 п.н. в стоп-кодоне). В большей степени это касается старт-кодонов. Это еще раз подтверждает, что определение старт-кодона, с которого начинается ORF, - нетривиальная задача, в том числе и потому, что у бактерий встречается несколько вариантов стартовых кодонов.
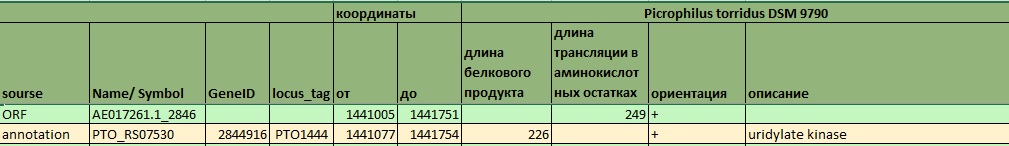
Рис.9 Различающиеся старт-кодоны в аннотации гена и ORF
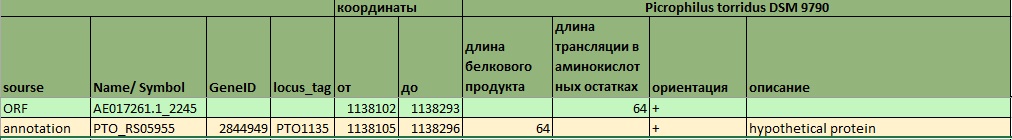
Рис. 10а
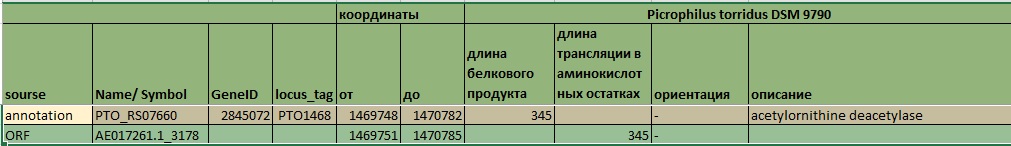
Рис. 10б
Рис. 10: Старт-кодоны различаются на 1 кодон (3.п.н.). Формально получается, что ORF и аннотация смещены друг односительно друга на 1 кодон. Судя по всему, в месте начала гена друг за другом шли два возможных старт-кодона, и программа выбрала не тот, который является стартовым в реальности.
В целом, случаи расхождения между двумя таблицами довольно однообразны и заключаются в разных старт- или, реже, стоп-кодонах.
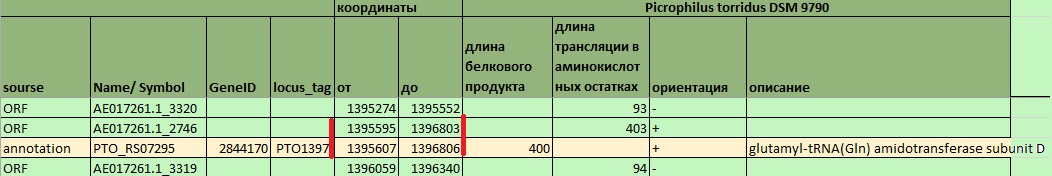
Рис.11 Различающиеся старт-кодоны
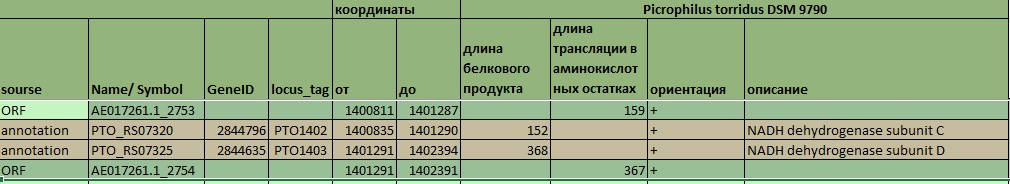
Рис.12 Рис.11 Различающиеся в первой паре и совпадающие во второй паре старт-кодоны