Поиск гомологичных последовательностей с помощью BLAST
Выбраный белок и найденные гомологи
Белок, который я описывал в практикуме номер 7, отсутсвует в базе данных Swiss-Prot. Всвязи с этим был найден и выбран другой белок. Для его поиска я воспользовался поисковым запросом "Bifunctional protein GlmU" AND (taxonomy_name:Rhodospirillaceae), а также применил фильтр для поиска только в Swiss-Prot, чтобы найти белок с таким же названием у родственной группы. По данному запросу был найден 1 результат (чтобы результатов было больше, можно искать по запросу "Bifunctional protein GlmU" AND (taxonomy_name:Rhodospirillales), тогда находок будет 6). Исследуемым будет белок с ID: GLMU_RHORT (AC: Q2RPX0).
Для поиска гомологов с помощью BLAST были установлены следующие параметры (непредставленные параметры оставались изначальными):
- Database (база данных): UniProtKB/Swiss-Prot(swissprot)
- Max target sequences (максимальное число находок): 1 000
- Expect threshold (порог на E-value): 0.05
- Word size (длина слова): 2
- Matrix (используемая матрица): BLOSUM62
- Gap Costs (штафы за гэп): Existence (за открытие): 11 Extension(за удлинение): 1
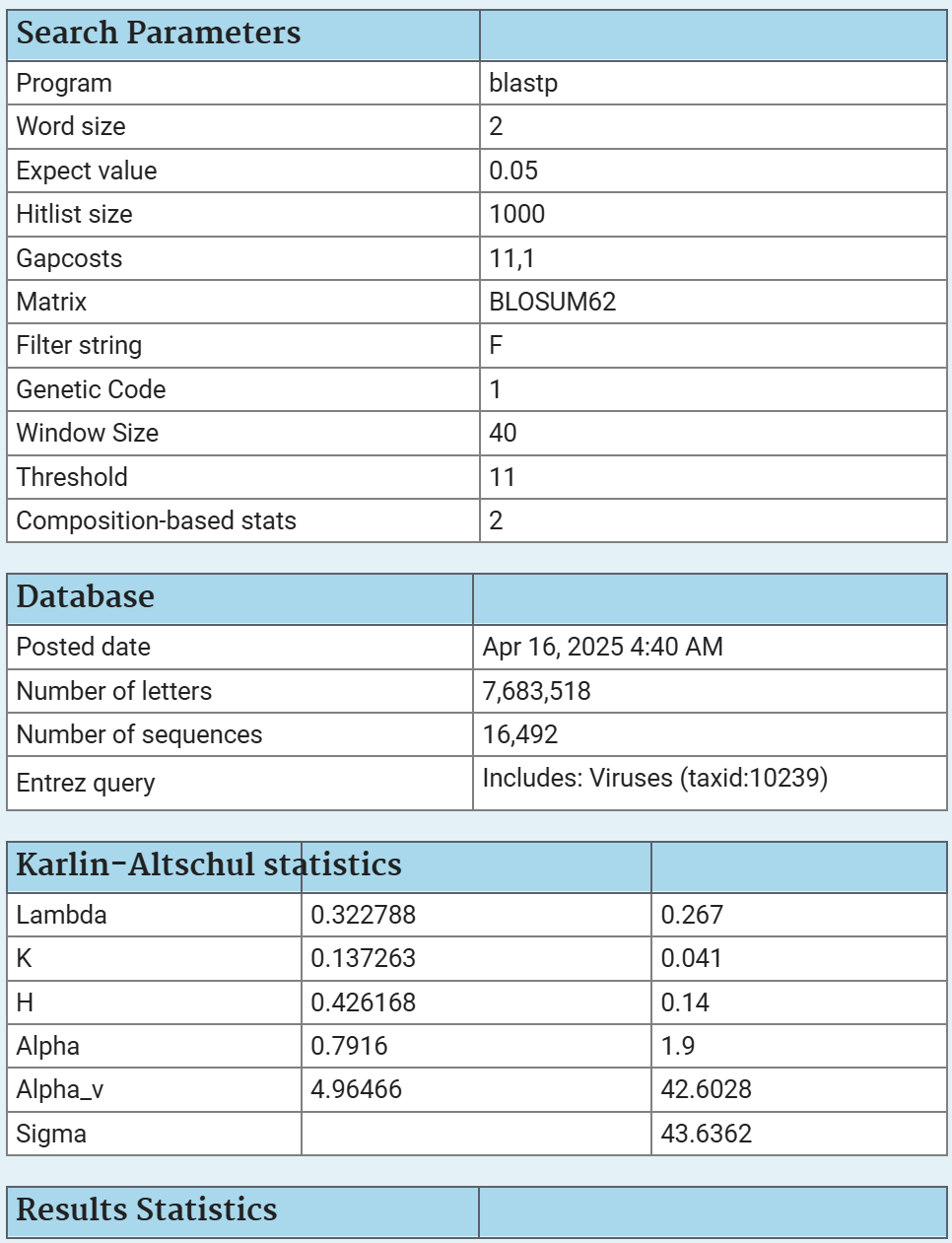
Парметры подробнее на рисунке 1.
В результате было найденно 773 последовательности. Ознакомиться с ними подробнее можно в файле.
Из всех находок я отобрал 6 (ниже представлены их AC):
- Q2W7U1
- A5VF26
- Q163N8
- Q6FZH5
- Q11HG1
- P28017
По выравниванию этих белков, оказалось, что из всех можно выделить один, а именно GLMU_PRING (AC: P28017). Во-первых, длина этого белка значительно меньше, чем у остальных. Во-вторых, у него отсутсвуют значимые части (консервативные участки в местах связывания и в каталитическом центре). По этим причинам он был вырезан из выравнивания. Остальные белки можно считать гомологичными, так как они сохраняют консервативные участки.
Белок из 7 практикума
Если всё таки делать выравнивание с белком из 7 практикума, взяв его последовательность из базы данных UniProt, то преминив те же параметры BLAST в результате будет полученно 703 последовательности, из которых были выбраны (ниже представлены AC):
- Q2W7U1
- A4WU43
- A1AZN6
- A5G0T8
- P08632
Было сделанно выравнивание и загружено в Jalview. Оказалось, что белок Nodulation protein L (AC: P08632) сильно отличается от остальных, и количество схожих участков у него мало. Из-за этого он был вырезан из выравнивания. Остальные белки оказались похожими друг на друга и их можно назвать гомологичными.
Гомологи зрелого вирусного белка, вырезанного из полипротеина
Мною был выбран полипротеин вируса под названием Porcine epidemic diarrhea virus (strain CV777) (PEDV) (вирус эпидемической диареи свиней; ID: R1AB_PEDV7; AC: P0C6Y4). В нём есть зрелый протеин под названием RNA-directed RNA polymerase nsp12, который располагается с 4 101 по 5 027 аминокислотный остаток.
С помощью EMBOSS был выбран участок цепи, соответсвующий данному зрелому белку.
descseq 'sw:R1AB_PEDV7[4101:5027]' segment.fasta -name 'RNA-directed RNA polymerase nsp12' -description '4101..5027'
Для поиска гомологов с помощью BLAST были установлены следующие параметры (непредставленные параметры оставались изначальными):
- Database (база данных): UniProtKB/Swiss-Prot(swissprot)
- Max target sequences (максимальное число находок): 1 000
- Expect threshold (порог на E-value): 0.05
- Word size (длина слова): 2
- Matrix (используемая матрица): BLOSUM62
- Gap Costs (штафы за гэп): Existence (за открытие): 11 Extension(за удлинение): 1
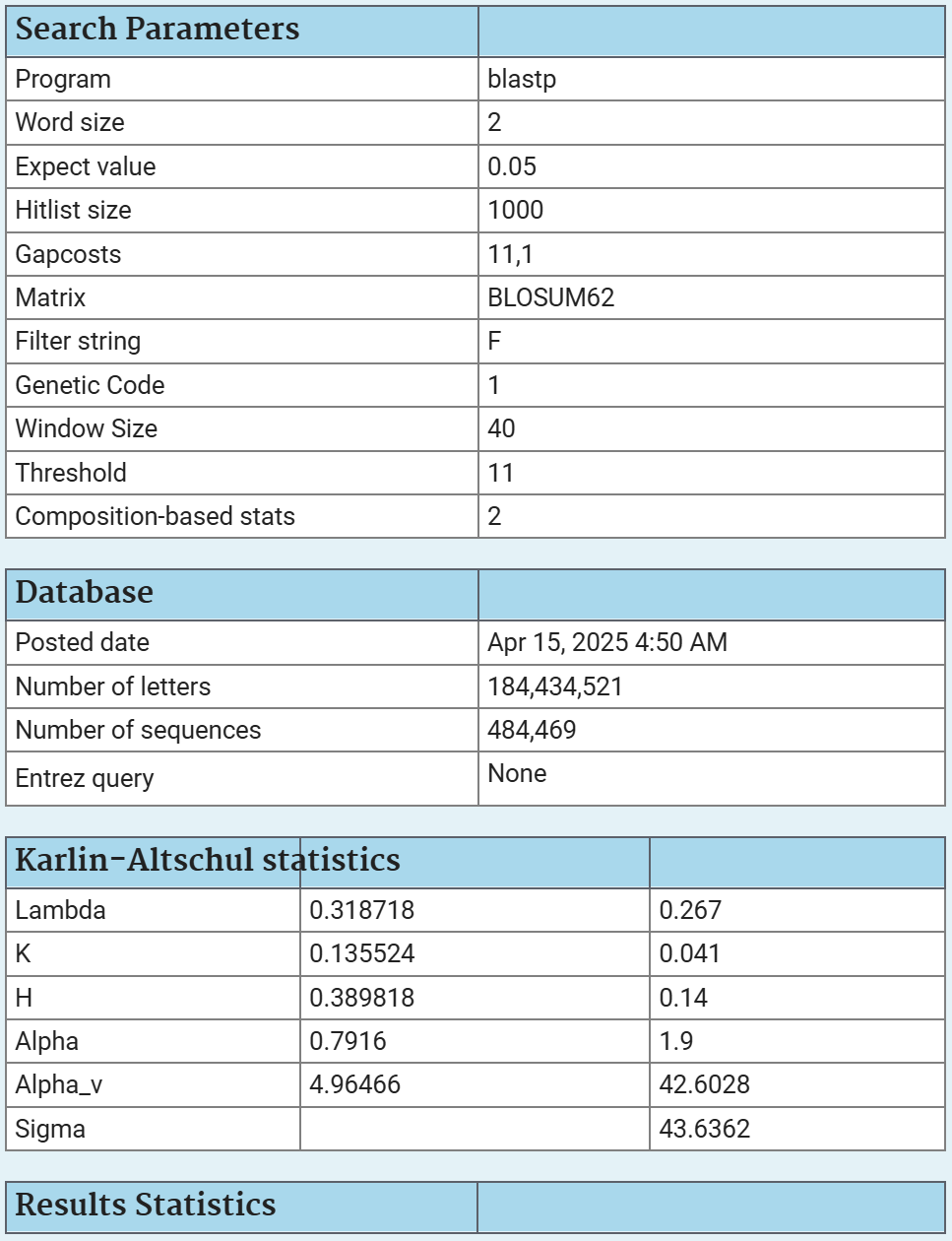
Парметры подробнее на рисунке 2.
В результате было найденно 46 последовательностей. Ознакомиться с ними подробнее можно в файле.
Из всех находок я отобрал 6 (ниже представлены их AC):
- P0C6W0
- P0C6Y0
- P0C6W5
- P0C6X4
- P0C6V7
- P0C6X5
По выравниванию видно, что последовательности имеют очень большой процент идентичности, что свидетельствует о их гомологичности. Наибольшее внимание вызывает последовательность RIAB_BEV (AC: P0C6V7). В консервативных участках у неё есть совпадения в нескольких позициях, но значимая часть отличается. Также в выравнивании у данной последовательности встречается много гэпов. По моему мнению, нет оснований утверждать достоверно, что данная последовательность гомологична остальным. По этим причинам, она была вырезана из выравнивания.
Исследование зависимости E-value от объёма банка
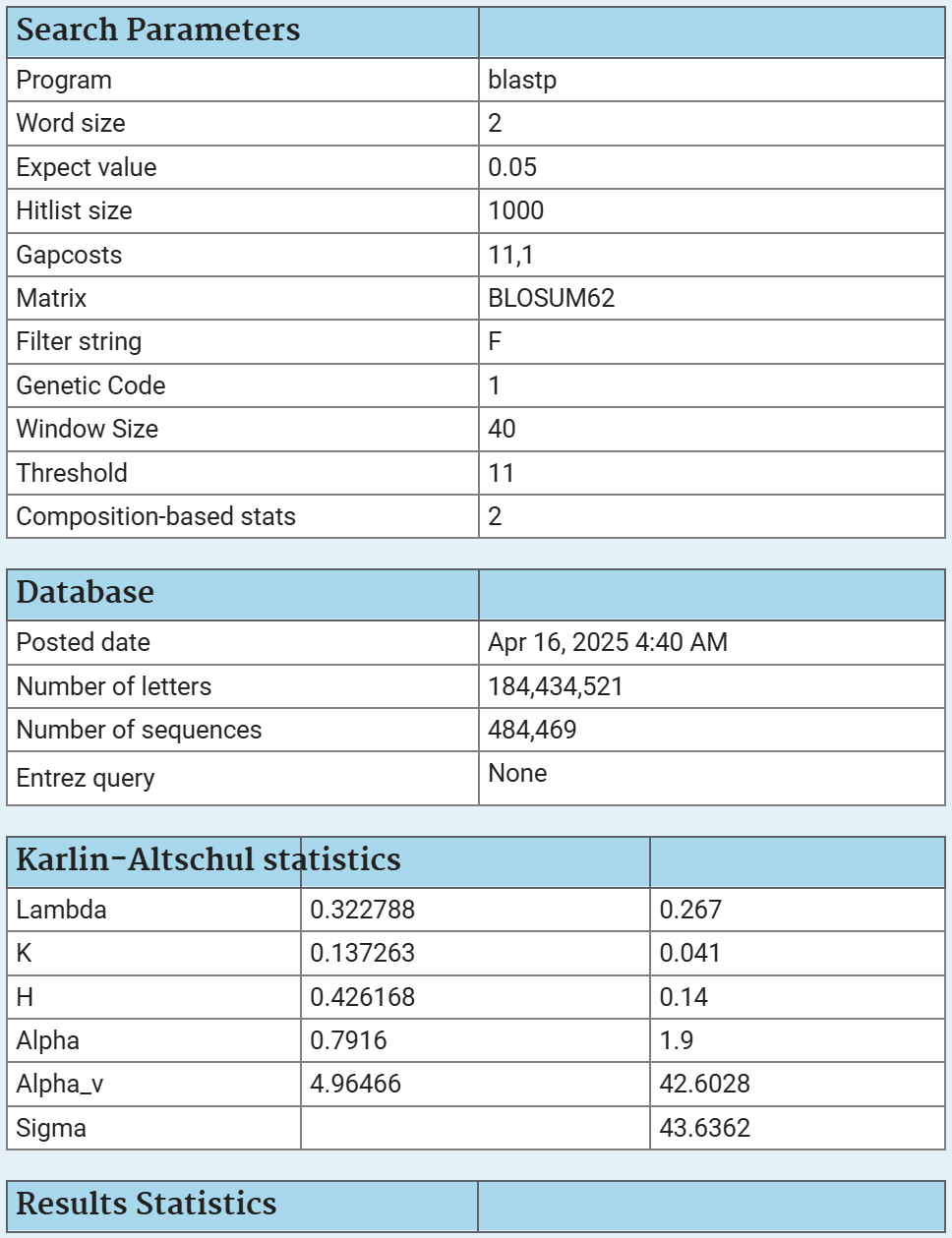
Сделав точно такой же запрос в BLAST, как и для вирусного белка, но применив фильтр по организмам (Viruses (taxid:10239)), я получил другое количество результатов, а именно 55. Если сравнить параметры (рисунок 2; рисунок 3), то можно заметить, что снизилось число "букв" (Number of letters) и последовательностей (Number of sequences), то есть выборка стала происходить из меньшего числа последовательностей.
Вместе с этим, значения E-value снизились. Если смотреть на последнюю находку из предыдущего запроса (AC: O56075) и на такую же находку из нового запроса, то можно увидеть разницу в значениях. В первом случае E-value составило 0.047, а во втором случае 0.002.
Чтобы узнать долю вирусных белков в базе данных нужно поделить значения E-value. Таким образом доля вирусных белков составляет 0.0426.
Если считать по другой находке, а именно по первой отличающейся (AC: Q9WQ77), у которой значения E-value по первому запросу 3 × 10−152, а по второму запросу 10−153, то доля получится 0,033.
Кроме того, можно ещё посчитать по количеству последовательностей (Number of sequences) на рисунке 3. Тогда получится значение 0.034
Из всех этих вычислений можно сделать вывод, что примерная доля вирусных белков в базе данных Swiss-Prot составляет от 0.03 до 0.045.