Features of Membrane Proteins
Задание 0. Характеристики некоторых ТМ белков
| PDB ID | Тип (спираль, баррель) |
Какая мембрана (внутренняя или внешняя, организм, органелла) |
Толщина гидрофобной части мембраны (Å) | Число ТМ участков в ТМ единице | Число остатков в одном ТМ участке (медиана, min, max) |
| 2BS2 страница |
спираль | Внутренняя мембрана грамотрицательных бактерий; Участвует в анаэробном дыхании (превращает фумарат в сукцинат) |
31.4 ± 1.3 | 5 | 26, 23, 27 |
| 1R3J страница |
спираль | Цитоплазматическая мембрана грамположительных бактерий; Действует как калиевый ионный канал. |
34.8 ± 1.2 | 3 | 26, 9, 32 |
| 4A2N страница |
спираль | Мембрана архебактерий (заякорен с внутренней стороны) | 30.6 ± 1.2 | 5 | 22, 19, 24 |
| 1UUN страница |
баррель | Заякорен в мембрану с внешней стороны (грамположительные бактерии) | 40.7 ± 2.1 | 2 | 9.5, 9, 10 |
| 1PHO страница |
баррель | Наружная мембрана грамотрицательных бактерий; Поглощение неорганического фосфата, фосфорилированных соединений и некоторых других отрицательно заряженных растворенных веществ. |
24.1 ± 1.2 | 16 | 9, 6, 13 |
| 2O4V страница |
баррель | Наружная мембрана грамотрицательных бактерий; Анион-специфичный сайт связывания имеет более высокое сродство к фосфат-, чем к хлорид-ионам. Порин O имеет более высокое сродство к полифосфатам (триполифосфату и пирофосфату), а порин P имеет более высокое сродство к ортофосфату. |
23.9 ± 1.1 | 16 | 9, 7, 13 |
Судя по представленным в таблице данным для выбранных ТМ белков толщина липидного бислоя составляет 23-36 Å; наружная мембрана грамотрицательных бактерий тоньше внутренней, которая, в свою очередь, тоньше цитоплазматической мембраны грамположительных бактерий; одна трансмембранная α-спираль содержит, как правило, больше а.о. (в среднем 22 а.о.), чем один β-тяж (примерно 9 а.о.).
Задание 1. Отбор гомологов белка FRDC_WOLSU
Выданный белок Fumarate reductase (Succinate dehydrogenase) принадлежит организму Wolinella succinogenes (грамотрицательная протеобактерия). C-цепь белка входит в состав цитохромB субъединицы фермента (Fumarate reductase cytochrome b subunit, FRDC_WOLSU) fasta-sequence.
Был произведен поиск гомологов белка с помощью PSI-BLAST (как описано в подсказках). При поиске гомологов были исключены белки из филума Proteobacteria.
После 4 итераций PSI-BLAST (порог на E-value 1e-5, максимальное количество хитов 500) было отобрано 11 хитов из разных таксономических групп. Затем названия последовательностей были отредактированы с сохранением указания на сам белок и соответствующий ему организм. Отбирались белки с наибольшим Query cover и для наибольшего разнообразия в пределах групп прокариот (нашлись только прокариоты). seqs.fasta
Задание 2. Анализ структуры белка FRDC_WOLSU
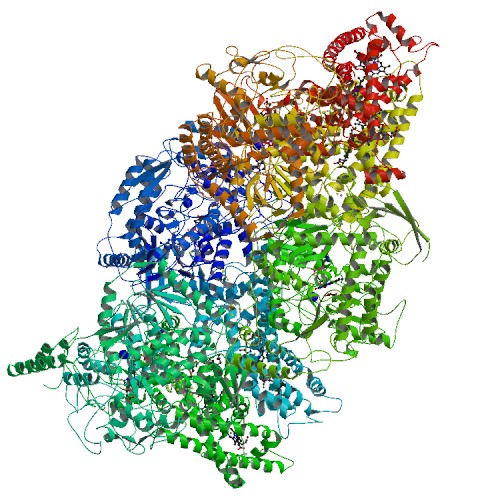
В базе данных OPM находим выданный белок 1E7P.
В базе данных TCBD отсутствует данная структура. По ссылке из OPM можно определить: 5.A.3.* - The Prokaryotic Molybdopterin-containing Oxidoreductase (PMO) Family. Однако поиск по названию в самой TCBD дает следущее: 3.D.10.* The Prokaryotic Succinate Dehydrogenase (SDH) Family. Очевидно, что вторая находка подходит. Из этого семейства в базе есть только один белок: Succinate:menaquinone oxidoreductase, SdhABC (pmf consuming when starting with succinate; pmf generating when starting with fumarate (the reverse reaction)) и это не тот. который нужен.
| PDB ID | Организм | Тип мембраны | TC-код | EC-код фермента | Наклон спиралей (бета-тяжей) к нормали | Количество трансмембранных втоичных структур | Название белка |
| 1E7P | Wolinella succinogenes (грамотрицательная протеобактерия) | Внутренняя мембрана | 3.D.10.* | 1.3.99.1 | 2 ± 0 | 10 | Fumarate reductase (Succinate dehydrogenase) cytochrome b subunit |
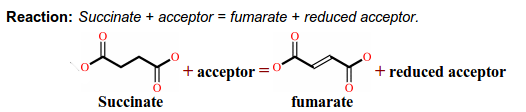
Схема катализируемой реакции:
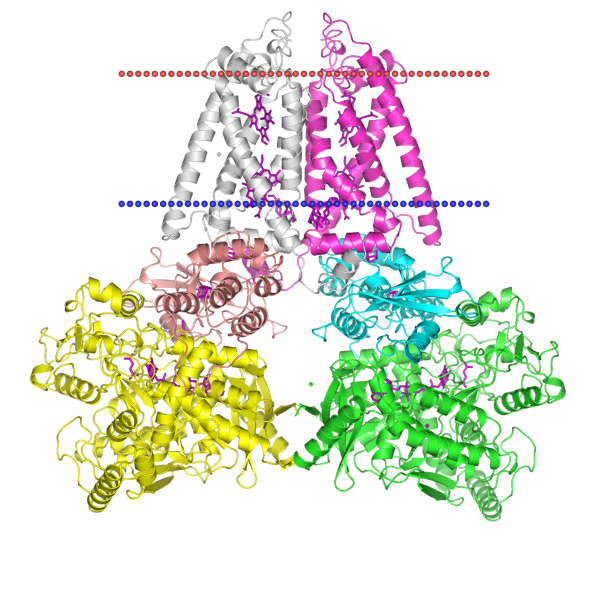
Представляет собой белковый комплекс из нескольких субъединиц. С-цепь принадлежит к субъединице cytochrome b:
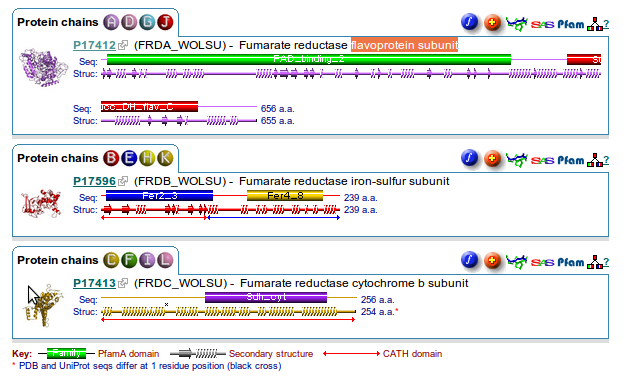
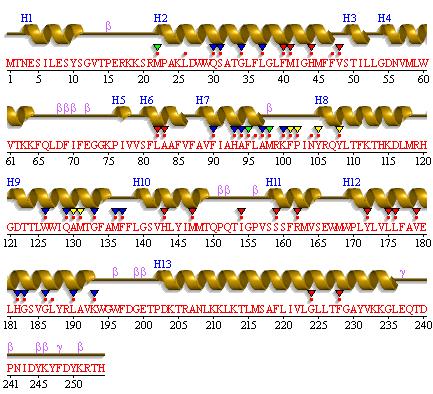
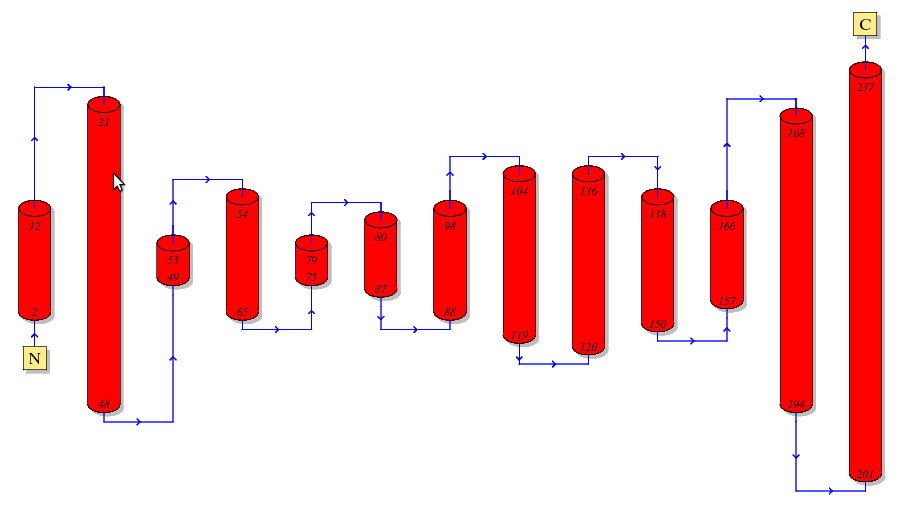
В поисках более подробной информации о TC-коде я наткнулась на схемы вторичной структуры С-цепи белка:
Задание 3. Анализ множественного выравнивания трансмембранных белков
Построим множественное выравнивание отобранных гомологов с помощью Muscle (align.fasta):
muscle -in seqs.fasta -out align.fasta
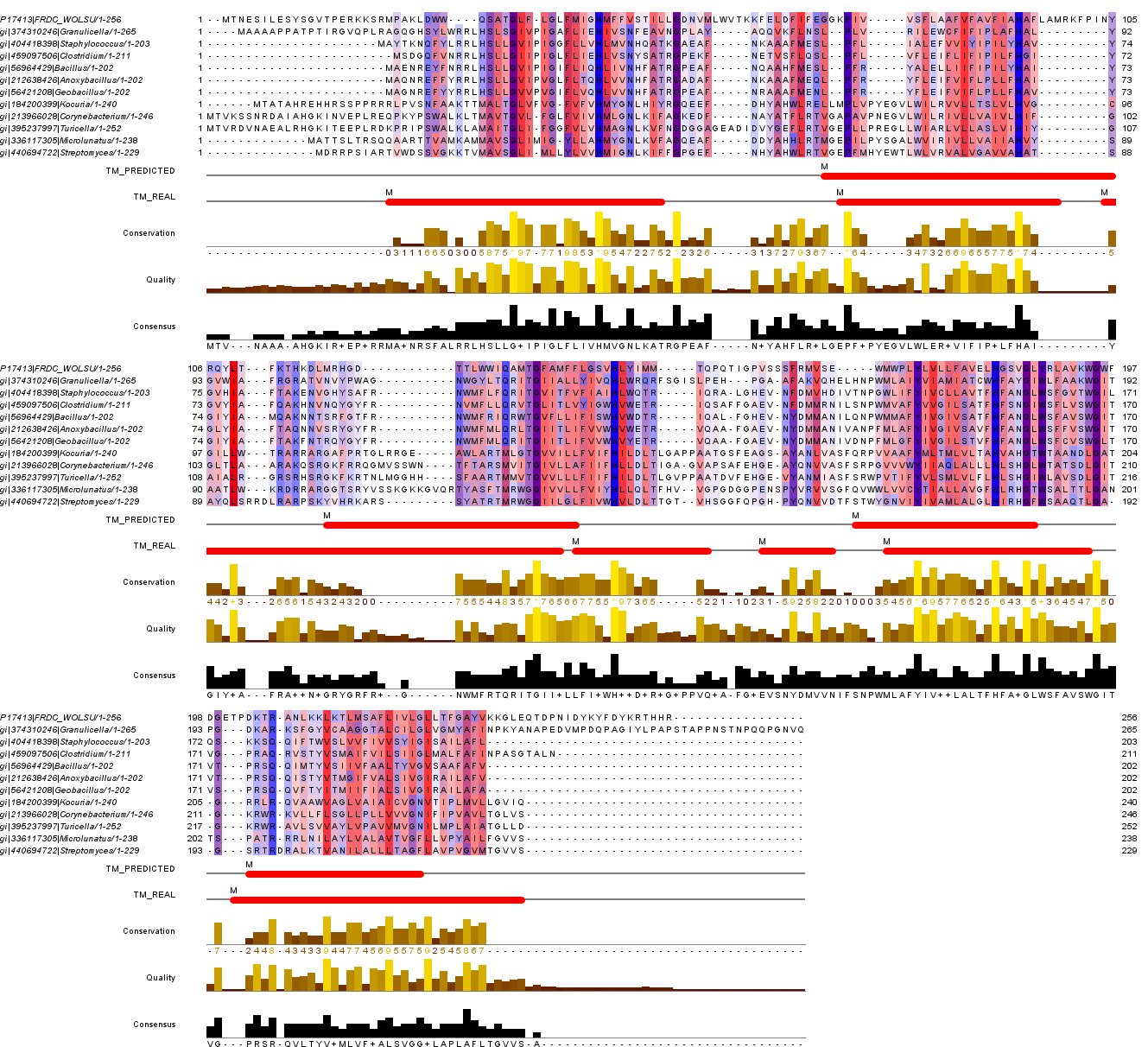
Загрузим полученное множественное выравнивание в программу JalView. К выравниванию добавим аннотации TM_REAL и TM_PREDICTED. TM_REAL содержит указания на участки последовательности, отвечающие трансмембранным спиралям в данном белке (1E7P:C), которая была прикреплена к его последовательности FRDC_WOLSU в выравнивании в программе JalView. TM_PREDICTED содержит предсказанные с помощью TMHMM участки трансмембранных спиралей в гомологе из Granulicella mallensis.
В поле TM_REAL получилось 7 спиралей (jal.jar). Хотя в базе данных OPM для белка 1E7P указано 10 вторичных стуртур, а на картинке выше мы видим целых 13 спиралей (минус 1 не ТМ - 12 штук).
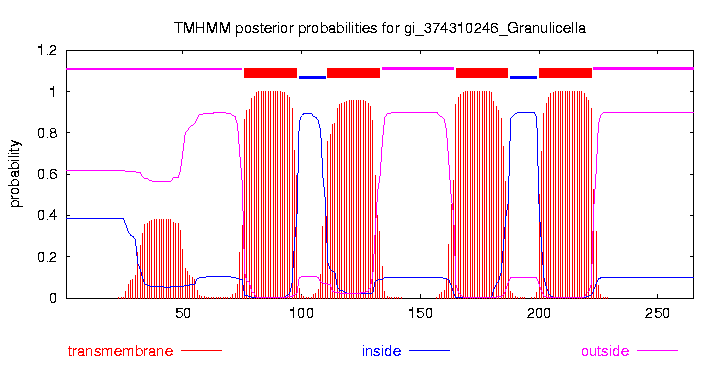
Предсказание с помощью TMHMM:
# gi_374310246_Granulicella Length: 265 # gi_374310246_Granulicella Number of predicted TMHs: 4 # gi_374310246_Granulicella Exp number of AAs in TMHs: 96.97039 # gi_374310246_Granulicella Exp number, first 60 AAs: 7.96999 # gi_374310246_Granulicella Total prob of N-in: 0.38421 gi_374310246_Granulicella TMHMM2.0 outside 1 75 gi_374310246_Granulicella TMHMM2.0 TMhelix 76 98 gi_374310246_Granulicella TMHMM2.0 inside 99 110 gi_374310246_Granulicella TMHMM2.0 TMhelix 111 133 gi_374310246_Granulicella TMHMM2.0 outside 134 164 gi_374310246_Granulicella TMHMM2.0 TMhelix 165 187 gi_374310246_Granulicella TMHMM2.0 inside 188 199 gi_374310246_Granulicella TMHMM2.0 TMhelix 200 222 gi_374310246_Granulicella TMHMM2.0 outside 223 265
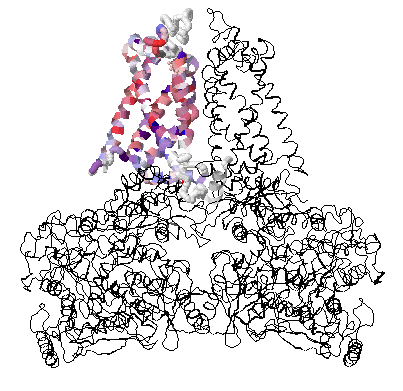
Выбранная цветовая схема позволяет визуально различать гидрофобные и гидрофильные остатки (Color -> Hydrophobicity), а также консерватиность отатков (Color -> By Conservation 20%). Цвета на структуре отвечают выбранной цветовой схеме. На каринке белок расположен так, чтобы его часть, ориентированная в n-сторону мембраны - сверху, а ориентированная в p-сторону - снизу.
Участки, относящиеся к ТМ спиралям, в основном консервативны. На глаз наиболее часто в спиралях встречаются остатки аланина, метионина, валина, лейцина, изолейцина, глицина, фениалаланина и пролина - в основном гидрофобные остатки. Среди участков между спиралями встречаются в основном неконсервативные участки, хотя консервативные тоже есть(порог 20%).
В ТМ спиралях более менее часто встречаются остатки серина, треонина, гистидина, аспарагина, тирозина (полярны). Как правило, эти остатки консервативны и, наверное, их присутствие связано с функциями белка.
Результат программы TMHMM и реальнаяструктурная информацию не особо совпадают. Я могу объяснить это тем, что гомологи нашлись какие-то кривые или я просто выбрала неудачный гомолог для сравнения. Присутствуют участки, где две аннотации пересекаются, но явного совпадения не видно. Предсказанные ТМ структуры иногда даже не попадают на консервативные позиции.
Наверх