| Число находок с E-value < 0,001 | |
| E-value лучшей находки | |
| Название последовательности с лучшей находкой | AE017333 Bacillus licheniformis DSM 13, complete genome |
| Координаты лучшей находки (от-до) | |
| Доля последовательности DACC_BACSU, вошедшая в выравнивание с лучшей находкой |
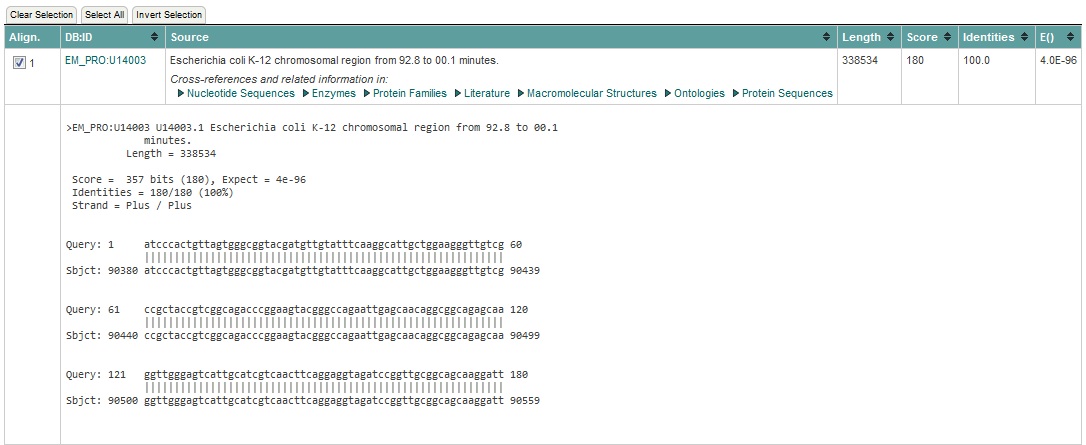
FT gene 90080..91030 FT /gene="miaA" FT CDS 90080..91030 FT /codon_start=1 FT /transl_table=11 FT /gene="miaA" FT /product="tRNA delta-2-isopentenylpyrophosphate (IPP) FT transferase" FT /note="CG Site No. 18160; alternate gene name trpX" FT /db_xref="GOA:P16384" FT /db_xref="InterPro:IPR002627" FT /db_xref="InterPro:IPR018022" FT /db_xref="PDB:2ZM5" FT /db_xref="PDB:2ZXU" FT /db_xref="PDB:3FOZ" FT /db_xref="UniProtKB/Swiss-Prot:P16384" FT /protein_id="AAA97067.1" FT /translation="MSDISKASLPKAIFLMGPTASGKTALAIELRKILPVELISVDSAL FT IYKGMDIGTAKPNAEELLAAPHRLLDIRDPSQAYSAADFRRDALAEMADITAAGRIPLL FT VGGTMLYFKALLEGLSPLPSADPEVRARIEQQAAEQGWESLHRQLQEVDPVAAARIHPN FT DPQRLSRALEVFFISGKTLTELTQTSGDALPYQVHQFAIAPASRELLHQRIEQRFHQML FT ASGFEAEVRALFARGDLHTDLPSIRCVGYRQMWSYLEGEISYDEMVYRGVCATRQLAKR FT QITWLRGWEGVHWLDSEKPEQARDEVLQVVGAIAG" FT misc_feature 90378..95221 FT /note="corresponds to GenBank Accession Number U00005 FT (ECOHFLA)" FT misc_feature 90378..91667 FT /note="corresponds to GenBank Accession Number D00743 FT (ECOHFQ)" FT promoter 91027..91054 FT /note="promoter matrix score of 55; putative"
FT gene complement(1998340..1999815) FT /gene="dacC" FT /locus_tag="BSU18350" FT CDS complement(1998340..1999815) FT /codon_start=1 FT /transl_table=11 FT /gene="dacC" FT /locus_tag="BSU18350" FT /product="D-alanyl-D-alanine carboxypeptidase" FT /function="16.13: Shape" FT /EC_number="3.4.16.4" FT /note="Evidence 1a: Function experimentally demonstrated in FT the studied strain; PubMedId: 11160090, 14731276, 9733705, FT 9864321; Product type e: enzyme" FT /db_xref="GOA:P39844" FT /db_xref="InterPro:IPR000667" FT /db_xref="InterPro:IPR012338" FT /db_xref="PDB:1W5D" FT /db_xref="PDB:2J9P" FT /db_xref="SubtiList:BG10969" FT /db_xref="UniProtKB/Swiss-Prot:P39844" FT /inference="ab initio prediction:AMIGene:2.0" FT /protein_id="CAB13718.1" FT /translation="MKKSIKLYVAVLLLFVVASVPYMHQAALAAEKQDALSGQIDKILA FT DHPALEGAMAGITVRSAETGAVLYEHSGDTRMRPASSLKLLTAAAALSVLGENYSFTTE FT VRTDGTLKGKKLNGNLYLKGKGDPTLLPSDFDKMAEILKHSGVKVIKGNLIGDDTWHDD FT MRLSPDMPWSDEYTYYGAPISALTASPNEDYDAGTVIVEVTPNQKEGEEPAVSVSPKTD FT YITIKNDAKTTAAGSEKDLTIEREHGTNTITIEGSVPVDANKTKEWISVWEPAGYALDL FT FKQSLKKQGITVKGDIKTGEAPSSSDVLLSHRSMPLSKLFVPFMKLSNNGHAEVLVKEM FT GKVKKGEGSWEKGLEVLNSTLPEFGVDSKSLVLRDGSGISHIDAVSSDQLSQLLYDIQD FT QSWFSAYLNSLPVAGNPDRMVGGTLRNRMKGTPAQGKVRAKTGSLSTVSSLSGYAETKS FT GKKLVFSILLNGLIDEEDGKDIEDQIAVILANQ"
| Число находок с E-value < 0,001 | |
| E-value лучшей находки | |
| Название последовательности с лучшей находкой | AE017333 Bacillus licheniformis DSM 13, complete genome |
| Координаты лучшей находки (от-до) |