сахарофосфатный остов A-формы ДНК
все нуклеотиды (G-красный, A-зеленый ,T-синий, C- желтый)
все аденины
атом N7 во всех гуанинах и/или только в первом по последовательности
|
сахарофосфатный остов A-формы ДНК |
все нуклеотиды (G-красный, A-зеленый ,T-синий, C- желтый) |
|
все аденины |
атом N7 во всех гуанинах и/или только в первом по последовательности |
|
сахарофосфатный остов B-формы ДНК |
все нуклеотиды (G-красный, A-зеленый ,T-синий, C- желтый) |
|
все аденины |
атом N7 во всех гуанинах и/или только в первом по последовательности |
|
сахарофосфатный остов Z-формы ДНК |
все нуклеотиды (G-красный, A-зеленый ,T-синий, C- желтый) |
|
все аденины (в последоывательности нет аденинов) |
атом N7 во всех гуанинах и/или только в первом по последовательности |
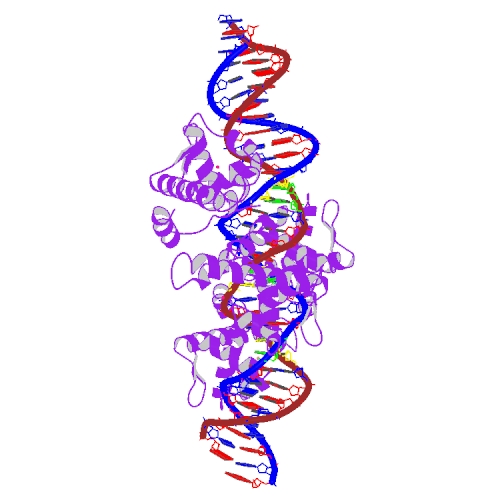 1DDN |
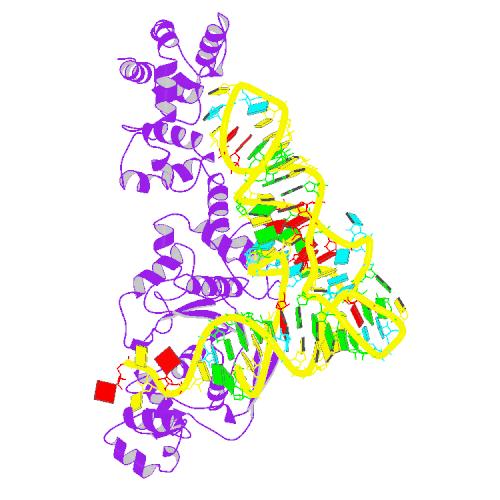 1G59 |
|
в ДНК, входящей в 1DDN, разрывов нет Файл с координатами только ДНК. |
в РНК, входящей в 1DDN, разрывов нет Файл с координатами только РНК. |
|
В молекуле B-ДНК белым изображен тимин 17, красным обозначены атомы, обращеные в сторону большой бороздки, синим обозначены атомы, обращеные в сторону малой бороздки. |
В молекуле A-ДНК белым изображен тимин 17, красным обозначены атомы, обращеные в сторону большой бороздки, синим обозначены атомы, обращеные в сторону малой бороздки. |
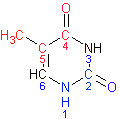
| B-форма ДНК | A-форма ДНК | |
| Атомы обращенные в сторону большой бороздки | T11.CM5, T11.C5, T11.C4, T11.O4 | T11.N1, T11.C2, T11.O2 |
| Атомы обращенные в сторону малой бороздки | T11.N1, T11.C2, T11.O2 | T11.CM5, T11.C5, T11.C4, T11.O4 |
| Остальные атомы основания | T11.N3, T11.C6 | T11.N3, T11.C6 |
|
A-форма |
B-форма |
*Z-форма |
Тип спирали (правая или левая) |
правая |
правая |
правая |
Шаг спирали (A) |
28.03 |
33.75 |
43.50 |
Число оснований на виток |
11 |
10 |
12 |
Ширина большой бороздки |
16.81(С12) |
17.21(A3) |
18.30(C10) |
Ширина малой бороздки |
7.98(С12) |
11.69(A3) |
9.86(G17) |
| α P-O5' |
β O5'-C5' |
γ C5'-C4' |
δ C4'-C3' |
ε C3'-O3' |
ξ O3'-P |
χ C1'-N |
|
| А-Днк для Т11 | -51.71 | 174.79 | 41.72 | 79.10 | -147.78 | -75.12 | -157.20 |
| А-Днк в презентации | -62 | 173 | 52 | 88 или 3 | 178 | -50 | 160 |
| В-Днк для Т11 | -29.87 | 136.38 | 31.10 | 143.42 | -140.77 | -160.52 | -97.95 |
| А-Днк в презентации | -63 | 171 | 54 | 123 или 131 | 155 | -90 | -117 |
Strand I base alpha beta gamma delta epsilon zeta chi 1 G --- 174.8 41.7 79.0 -147.8 -75.1 -157.2 2 A -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 3 T -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 4 C -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 5 G -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 6 A -51.7 174.8 41.7 79.0 -147.8 -75.1 -157.2 7 T -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 8 C -51.7 174.8 41.7 79.0 -147.8 -75.0 -157.2 9 G -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 10 A -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 11 T -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 12 C -51.7 174.8 41.7 79.1 -147.7 -75.1 -157.2 13 G -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 14 A -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 15 T -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 16 C -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 17 G -51.7 174.8 41.7 79.0 -147.8 -75.1 -157.2 18 A -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 19 T -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 20 C -51.7 174.8 41.7 79.1 --- --- -157.2 Strand II base alpha beta gamma delta epsilon zeta chi 1 C -51.7 174.8 41.7 79.0 --- --- -157.2 2 T -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 3 A -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 4 G -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 5 C -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 6 T -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 7 A -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 8 G -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 9 C -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 10 T -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 11 A -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 12 G -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 13 C -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 14 T -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 15 A -51.7 174.8 41.7 79.0 -147.8 -75.1 -157.2 16 G -51.7 174.8 41.7 79.1 -147.7 -75.1 -157.2 17 C -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 18 T -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 19 A -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 20 G --- 174.8 41.7 79.1 -147.8 -75.1 -157.2Тоже самое я сделала для Z- и В-форм, и получила следующую информацию:
Strand I base alpha beta gamma delta epsilon zeta chi 1 G --- 136.4 31.1 143.4 -140.8 -160.5 -98.0 2 A -29.9 136.3 31.2 143.3 -140.8 -160.5 -98.0 3 T -29.9 136.3 31.1 143.3 -140.8 -160.5 -97.9 4 C -29.9 136.4 31.1 143.4 -140.8 -160.5 -98.0 5 G -29.9 136.3 31.2 143.3 -140.8 -160.5 -98.0 6 A -29.9 136.4 31.1 143.4 -140.8 -160.5 -98.0 7 T -29.9 136.3 31.2 143.3 -140.8 -160.5 -98.0 8 C -29.9 136.3 31.1 143.3 -140.8 -160.5 -97.9 9 G -29.9 136.4 31.1 143.4 -140.8 -160.5 -98.0 10 A -29.9 136.3 31.2 143.3 -140.8 -160.5 -98.0 11 T -29.9 136.4 31.1 143.4 -140.8 -160.5 -98.0 12 C -29.9 136.3 31.2 143.3 -140.8 -160.5 -98.0 13 G -29.9 136.3 31.1 143.3 -140.8 -160.5 -98.0 14 A -29.9 136.4 31.1 143.4 -140.8 -160.5 -98.0 15 T -29.9 136.3 31.2 143.3 -140.8 -160.5 -98.0 16 C -29.9 136.4 31.1 143.4 -140.8 -160.5 -98.0 17 G -29.9 136.3 31.2 143.3 -140.8 -160.5 -98.0 18 A -29.9 136.3 31.1 143.3 -140.8 -160.5 -98.0 19 T -29.9 136.4 31.1 143.4 -140.8 -160.5 -98.0 20 C -29.9 136.3 31.2 143.3 --- --- -98.0 Strand II base alpha beta gamma delta epsilon zeta chi 1 C -29.9 136.4 31.1 143.4 --- --- -98.0 2 T -29.9 136.3 31.2 143.3 -140.8 -160.5 -98.0 3 A -29.9 136.4 31.1 143.4 -140.8 -160.5 -98.0 4 G -29.9 136.3 31.1 143.3 -140.8 -160.5 -98.0 5 C -29.9 136.3 31.2 143.3 -140.8 -160.5 -98.0 6 T -29.9 136.4 31.1 143.4 -140.8 -160.5 -98.0 7 A -29.9 136.3 31.2 143.3 -140.8 -160.5 -98.0 8 G -29.9 136.4 31.1 143.4 -140.8 -160.5 -98.0 9 C -29.9 136.3 31.1 143.3 -140.8 -160.5 -97.9 10 T -29.9 136.3 31.2 143.3 -140.8 -160.5 -98.0 11 A -29.9 136.4 31.1 143.4 -140.8 -160.5 -98.0 12 G -29.9 136.3 31.2 143.3 -140.8 -160.5 -98.0 13 C -29.9 136.4 31.1 143.4 -140.8 -160.5 -98.0 14 T -29.9 136.3 31.1 143.3 -140.8 -160.5 -97.9 15 A -29.9 136.3 31.2 143.3 -140.8 -160.5 -98.0 16 G -29.9 136.4 31.1 143.4 -140.8 -160.5 -98.0 17 C -29.9 136.3 31.2 143.3 -140.8 -160.5 -98.0 18 T -29.9 136.4 31.1 143.4 -140.8 -160.5 -98.0 19 A -29.9 136.3 31.1 143.3 -140.8 -160.5 -98.0 20 G --- 136.3 31.2 143.3 -140.8 -160.5 -98.0Для Z-ДНК:
Strand I base alpha beta gamma delta epsilon zeta chi 1 G --- 179.0 -173.8 94.9 -103.6 -64.8 58.7 2 C -139.5 -136.7 50.9 137.6 -96.5 81.9 -154.3 3 G 52.0 179.0 -173.8 94.9 -103.6 -64.8 58.7 4 C -139.5 -136.8 50.8 137.6 -96.5 82.0 -154.3 5 G 51.9 179.0 -173.8 94.9 -103.6 -64.8 58.7 6 C -139.5 -136.8 50.9 137.6 -96.5 82.0 -154.3 7 G 51.9 179.0 -173.8 94.9 -103.6 -64.8 58.7 8 C -139.5 -136.7 50.9 137.6 -96.5 81.9 -154.3 9 G 52.0 179.0 -173.8 94.9 -103.6 -64.8 58.7 10 C -139.5 -136.8 50.8 137.6 -96.5 82.0 -154.3 11 G 51.9 179.0 -173.8 94.9 -103.6 -64.8 58.7 12 C -139.5 -136.8 50.9 137.6 -96.5 82.0 -154.3 13 G 51.9 179.0 -173.8 94.9 -103.6 -64.8 58.7 14 C -139.5 -136.7 50.9 137.6 -96.5 81.9 -154.3 15 G 52.0 179.0 -173.8 94.9 -103.6 -64.8 58.7 16 C -139.5 -136.8 50.8 137.6 -96.5 82.0 -154.3 17 G 51.9 179.0 -173.8 94.9 -103.6 -64.8 58.7 18 C -139.5 -136.8 50.9 137.6 -96.5 82.0 -154.3 19 G 51.9 179.0 -173.8 94.9 -103.6 -64.8 58.7 20 C -139.5 -136.7 50.9 137.6 --- --- -154.3 Strand II base alpha beta gamma delta epsilon zeta chi 1 C -139.5 -136.7 50.9 137.6 --- --- -154.3 2 G 51.9 179.0 -173.8 94.9 -103.6 -64.8 58.7 3 C -139.5 -136.8 50.9 137.6 -96.5 82.0 -154.3 4 G 51.9 179.0 -173.8 94.9 -103.6 -64.8 58.7 5 C -139.5 -136.8 50.8 137.6 -96.5 82.0 -154.3 6 G 52.0 179.0 -173.8 94.9 -103.6 -64.8 58.7 7 C -139.5 -136.7 50.9 137.6 -96.5 81.9 -154.3 8 G 51.9 179.0 -173.8 94.9 -103.6 -64.8 58.7 9 C -139.5 -136.8 50.9 137.6 -96.5 82.0 -154.3 10 G 51.9 179.0 -173.8 94.9 -103.6 -64.8 58.7 11 C -139.5 -136.8 50.8 137.6 -96.5 82.0 -154.3 12 G 52.0 179.0 -173.8 94.9 -103.6 -64.8 58.7 13 C -139.5 -136.7 50.9 137.6 -96.5 81.9 -154.3 14 G 51.9 179.0 -173.8 94.9 -103.6 -64.8 58.7 15 C -139.5 -136.8 50.9 137.6 -96.5 82.0 -154.3 16 G 51.9 179.0 -173.8 94.9 -103.6 -64.8 58.7 17 C -139.5 -136.8 50.8 137.6 -96.5 82.0 -154.3 18 G 52.0 179.0 -173.8 94.9 -103.6 -64.8 58.7 19 C -139.5 -136.7 50.9 137.6 -96.5 81.9 -154.3 20 G --- 179.0 -173.8 94.9 -103.6 -64.8 58.7Из приведенных выше данных видно, что для каждой формы ДНК характерны свои значения торсионных углов. Ниже приведена таблица с характерными значениями.
| α P-O5' |
β O5'-C5' |
γ C5'-C4' |
δ C4'-C3' |
ε C3'-O3' |
ξ O3'-P |
χ C1'-N |
|
| А-Днк | -51.7 | 174.8 | 41.7 | 79.1 | -147.8 | -75.1 | -157.2 |
| В-Днк | -29.9 | 136.4 | 31.1 | 143.4 | -140.8 | -160.5 | -98.0 |
| Z-Днк углы в C | -139.5 | -136.7 | 50.9 | 137.6 | -96.5 | 81.9 | -154.3 |
| Z-Днк углы в G | 52.0 | 179.0 | -173.8 | 94.9 | -103.6 | -64.8 | 58.7 |
Значения угла α близки в А- и В-формах; значения угла β положительны и близки в А- и В-формах и в G Z-формы, но отрицательны в С Z-формы; значения угла положительны и близки в А- и В-формах и в С Z-формы, но отрицательны в G Z-формы; значения угла δ положительны, но не близки во всех формах; значения угла &epsilo; во всех формах отрицательны и близки в А- и В-формах; значения угла ξ не близки во всех формах; значения угла χ близки в А-форме и в С Z-формы;Торсионные углы в молекуле тРНК 1G59
Strand I base alpha beta gamma delta epsilon zeta chi 1 G --- -161.2 55.0 98.3 -155.1 -75.7 -163.9 2 G -78.8 -165.2 44.9 96.0 -151.7 -71.0 -155.8 3 C -65.5 174.8 48.7 92.0 -152.0 -75.1 -146.4 4 C -57.5 175.0 44.6 91.2 -159.7 -84.2 -151.5 5 C -64.4 170.1 55.5 88.1 -158.5 -69.8 -162.5 6 C -64.6 -177.7 55.1 96.5 -162.4 -76.1 -163.7 7 A -75.6 175.3 74.8 130.5 --- --- -145.1 8 G --- 151.0 30.0 93.2 -147.9 -78.5 -173.7 9 G -69.3 177.2 61.9 92.2 -158.5 -66.6 -166.9 10 G -61.2 -173.6 40.7 90.2 -159.8 -99.2 -157.4 11 G 126.3 -151.0 -166.9 104.6 -119.8 -27.5 179.1 12 G -110.5 56.4 178.7 112.4 -118.9 -89.0 166.5 13 U 9.3 148.6 -19.7 92.5 -131.1 -75.9 -152.2 14 U -71.9 158.4 68.3 89.9 --- --- -146.5 15 A --- 172.6 51.8 92.5 -147.5 -58.4 -167.8 16 G -79.3 -179.5 57.9 90.6 -156.5 -77.8 -157.2 17 G -60.6 177.9 43.0 92.2 -156.5 -75.3 -157.2 18 C -76.6 174.7 60.8 88.5 -159.3 -69.1 -153.4 19 C -68.9 -178.7 54.0 87.1 -156.5 -73.1 -153.3 20 G -64.2 171.0 62.2 89.1 -155.9 -81.0 -163.1 21 A -59.9 -178.7 41.1 87.0 --- --- -146.0 22 G --- 166.4 48.1 97.2 -147.7 -72.6 -174.0 23 U -66.5 -172.1 46.0 93.2 -153.5 -78.6 -159.6 24 C -61.7 -177.5 43.0 90.7 -152.1 -73.5 -146.3 25 U 118.5 -152.1 -146.8 91.1 -159.5 -80.7 -172.2 26 A -63.5 -171.1 49.9 92.5 -134.0 -68.6 -163.1 27 G -76.2 161.0 71.7 98.9 --- --- -147.0 28 G --- -109.6 52.4 121.6 --- --- -59.1 Strand II base alpha beta gamma delta epsilon zeta chi 1 C 152.4 -141.2 163.0 105.5 --- --- -166.6 2 U -75.0 168.0 67.8 95.6 -153.5 -116.4 -161.1 3 G -57.7 175.8 44.9 94.5 -151.9 -73.1 -166.4 4 G -74.3 168.6 68.3 93.7 -155.8 -80.9 -169.3 5 G -55.1 174.2 40.2 90.2 -158.7 -73.3 -163.5 6 G -55.6 174.4 40.3 95.7 -158.1 -81.5 -153.4 7 U -60.5 176.0 48.0 94.3 -150.2 -77.8 -158.8 8 C -56.4 169.8 48.0 91.6 -150.4 -72.6 -168.6 9 C -60.8 167.2 47.2 92.9 -155.4 -81.0 -161.6 10 C -50.7 169.7 42.9 95.0 -152.6 -85.8 -144.8 11 C -72.7 -162.9 44.6 100.3 -153.9 -83.7 -156.3 12 C --- -166.4 35.7 100.6 -158.5 -83.2 -175.2 13 A --- 143.7 115.0 115.2 --- --- -133.9 14 G --- -116.2 50.9 109.8 --- --- -99.6 15 C -60.9 169.3 48.0 96.2 --- --- -159.1 16 C -61.3 171.1 58.6 89.4 -137.8 -71.2 -164.2 17 C -70.2 169.5 58.1 91.1 -156.2 -74.7 -152.3 18 G -72.1 173.9 59.9 88.2 -150.1 -84.4 -165.2 19 G -64.2 171.1 52.3 86.6 -152.4 -73.8 -164.9 20 C -67.2 -170.2 46.4 86.9 -148.6 -77.0 -151.6 21 G -67.9 175.9 46.3 87.4 -154.5 -62.7 -159.6 22 C -74.9 -174.3 59.6 92.0 -147.0 -65.1 -161.8 23 A -65.3 178.9 47.2 90.0 -162.6 -75.1 -161.8 24 G -52.3 170.8 52.0 91.7 -154.3 -76.0 -164.5 25 G --- 153.8 38.2 87.0 -150.3 -82.3 -166.4 26 U --- -150.8 52.9 96.0 --- --- -141.0 27 C --- 105.3 170.0 134.9 --- --- -134.4 28 C --- -151.0 -179.2 106.6 --- --- 143.6Мне кажется, что эта молекула тРНК больше похожа на A-форму ДНК.
Strand I base alpha beta gamma delta epsilon zeta chi 1 T --- -118.5 -94.9 154.7 -128.2 -115.0 -120.2 2 A 62.6 -129.3 -138.0 149.9 -122.8 -71.6 -125.3 3 T -11.4 -72.8 -105.2 152.5 -172.9 -77.1 -130.4 4 A -77.2 -167.8 44.6 149.5 -175.8 -124.9 -91.6 5 A 55.9 -160.3 -83.1 148.9 -171.7 -112.2 -119.3 6 T 51.8 -162.2 -79.0 152.3 -169.1 -112.4 -129.0 7 T 37.0 166.0 -45.2 154.4 174.7 -79.0 -108.5 8 A -61.5 -155.4 22.4 150.7 173.5 -103.0 -85.3 9 G -54.0 -170.8 33.7 150.0 -171.2 -95.7 -83.2 10 G -7.2 -95.9 -72.2 151.2 -175.5 -88.1 -146.2 11 A -49.0 -160.3 35.3 149.1 -166.5 -123.5 -115.3 12 T 39.0 -178.9 -61.1 151.8 -172.9 -105.4 -107.8 13 A -46.2 171.7 42.1 148.7 -153.7 -109.9 -100.4 14 G 11.8 -151.7 -63.2 157.6 -159.6 -106.1 -105.1 15 C 22.4 -159.3 -76.3 146.4 -174.8 -117.7 -117.5 16 T 47.6 -171.0 -63.1 145.1 -165.5 -100.7 -133.1 17 T -52.1 177.2 49.7 151.2 -164.8 -130.1 -103.5 18 T 40.0 -173.3 -64.9 156.0 -178.7 -73.8 -110.6 19 A -57.5 -169.7 28.0 150.4 -150.5 -137.6 -84.1 20 C 23.4 -160.0 -83.9 152.2 175.3 -108.6 -118.8 21 C -20.9 -166.2 14.3 151.5 -171.2 -130.0 -119.2 22 T -45.5 173.4 43.6 147.4 -179.1 -105.8 -112.7 23 A -50.3 -172.3 41.1 150.7 -157.0 -115.3 -97.3 24 A 32.3 -131.5 -86.0 151.8 -150.5 -99.7 -127.9 25 T 35.8 -140.3 -85.5 152.6 175.1 -87.2 -131.2 26 T 172.0 160.7 173.4 151.6 -134.6 -88.2 -138.6 27 A 8.3 -103.2 -92.2 152.0 -140.5 -89.4 -141.2 28 T 33.2 -139.0 -94.5 153.4 -147.8 -93.4 -123.1 29 T 28.3 -133.0 -84.0 153.9 -141.6 -94.3 -116.1 30 T 34.2 -116.8 -96.2 153.1 -121.3 -77.2 -117.9 31 T -0.7 -64.9 -111.5 145.0 -96.6 -134.2 -142.4 32 A 95.5 -125.0 -179.0 153.6 --- --- -108.3 Strand II base alpha beta gamma delta epsilon zeta chi 1 A -53.6 -174.8 42.2 159.9 --- --- -95.0 2 T 26.6 -178.8 -62.7 150.5 167.0 -90.2 -99.8 3 A 28.6 -122.5 -94.0 156.4 -155.5 -98.2 -138.5 4 T 55.8 179.2 -59.9 158.3 -149.4 -112.6 -111.1 5 T -41.4 -168.5 44.5 151.4 -179.3 -127.3 -113.6 6 A -63.3 -167.9 33.1 149.0 159.5 -107.4 -96.0 7 A 39.6 -150.1 -79.5 153.4 -179.8 -83.7 -109.8 8 T -51.4 -179.7 47.8 148.8 -156.4 -117.7 -108.6 9 C -18.2 -170.2 9.8 149.0 -169.0 -122.5 -125.4 10 C 26.8 -146.6 -101.8 147.3 -176.3 -108.5 -123.9 11 T 16.5 -165.6 -53.7 159.9 -144.7 -116.2 -101.3 12 A -49.3 178.6 42.4 147.6 -161.0 -110.9 -103.8 13 T -52.2 173.0 53.7 148.7 -173.3 -113.3 -107.8 14 C -9.8 -178.0 4.3 150.8 -171.5 -126.5 -121.7 15 G 28.7 -147.5 -95.5 144.6 -171.8 -110.3 -130.4 16 A 1.4 -128.7 -71.9 158.9 -149.2 -101.8 -114.7 17 A -45.4 175.7 42.0 149.7 -144.4 -104.4 -101.9 18 A -46.4 179.4 37.8 146.7 -177.9 -117.4 -99.5 19 T -43.8 -156.6 25.0 146.5 -170.8 -109.8 -108.7 20 G 16.2 -109.6 -79.4 147.4 -175.3 -88.0 -142.6 21 G -60.3 -175.7 39.6 150.3 -161.3 -116.7 -82.5 22 A -64.8 -152.5 23.9 149.8 175.7 -104.2 -86.8 23 T 45.0 163.7 -51.8 154.0 173.3 -75.9 -109.9 24 T 44.6 -156.3 -75.0 152.7 -175.0 -106.4 -130.4 25 A 46.6 -160.4 -75.1 150.5 -166.2 -119.3 -117.2 26 A -59.4 -167.5 46.1 148.1 -170.3 -122.0 -97.1 27 T 38.1 161.7 -57.5 156.9 169.5 -91.9 -99.6 28 A 23.3 -108.9 -97.6 153.3 -167.3 -115.5 -122.0 29 A 52.5 -140.8 -165.4 144.6 -124.4 -95.6 -122.2 30 A 90.3 -142.6 173.7 160.2 -80.1 -169.0 -115.8 31 A -57.9 125.5 63.2 153.4 -88.5 168.3 -98.1 32 T --- 147.9 28.4 135.1 -135.3 179.5 -83.9Подсчет средних значений углов в файле.
Strand I Strand II Helix
1 (0.009) D:.501_:[..G]G-----C[..C]:.572_:D (0.010) |
2 (0.010) D:.502_:[..G]G-*---U[..U]:.571_:D (0.016) |
3 (0.010) D:.503_:[..C]C-----G[..G]:.570_:D (0.008) |
4 (0.009) D:.504_:[..C]C-----G[..G]:.569_:D (0.006) |
5 (0.009) D:.505_:[..C]C-----G[..G]:.568_:D (0.009) |
6 (0.008) D:.506_:[..C]C-----G[..G]:.567_:D (0.008) |
7 (0.009) D:.507_:[..A]Ax----U[..U]:.566_:D (0.015) |
8 (0.011) D:.549_:[..G]G-----C[..C]:.565_:D (0.012) |
9 (0.008) D:.550_:[..G]G-----C[..C]:.564_:D (0.010) |
10 (0.009) D:.551_:[..G]G-----C[..C]:.563_:D (0.014) |
11 (0.007) D:.552_:[..G]G-----C[..C]:.562_:D (0.011) |
12 (0.011) D:.553_:[..G]G----xC[..C]:.561_:D (0.009) |
13 (0.017) D:.554_:[..U]U-*--xA[..A]:.558_:D (0.008) |
14 (0.019) D:.555_:[..U]Ux**+xG[..G]:.517_:D (0.022) x
15 (0.011) D:.538_:[..A]A-*---C[..C]:.532_:D (0.013) |
16 (0.013) D:.539_:[..G]G-----C[..C]:.531_:D (0.008) |
17 (0.008) D:.540_:[..G]G-----C[..C]:.530_:D (0.009) |
18 (0.010) D:.541_:[..C]C-----G[..G]:.529_:D (0.008) |
19 (0.010) D:.542_:[..C]C-----G[..G]:.528_:D (0.006) |
20 (0.009) D:.543_:[..G]G-----C[..C]:.527_:D (0.012) |
21 (0.009) D:.544_:[..A]Ax*---G[..G]:.526_:D (0.009) |
22 (0.010) D:.510_:[..G]G-----C[..C]:.525_:D (0.012) |
23 (0.018) D:.511_:[..U]U-----A[..A]:.524_:D (0.009) |
24 (0.011) D:.512_:[..C]C-----G[..G]:.523_:D (0.008) |
25 (0.016) D:.513_:[..U]U-*--xG[..G]:.522_:D (0.007) |
26 (0.010) D:.514_:[..A]A-**-xU[..U]:.508_:D (0.015) |
27 (0.010) D:.515_:[..G]Gx**+xC[..C]:.548_:D (0.008) x
28 (0.012) D:.518_:[..G]G-----C[..C]:.556_:D (0.032) +
1)Стебли:| Акцепторный стебель | участок 1-7 комплементарен участку 72-66 |
| T-стебель | участок 49-53 комплементарен участку 65-61 |
| D-стебель | участок 10-13 комплементарен участку 25-22 |
| Антикодоновый стебель | участок 38-44 комплементарен участку 32-26 |
12 (0.011) D:.553_:[..G]G----xC[..C]:.561_:D (0.009) | 13 (0.017) D:.554_:[..U]U-*--xA[..A]:.558_:D (0.008) 24 (0.011) D:.512_:[..C]C-----G[..G]:.523_:D (0.008) | 25 (0.016) D:.513_:[..U]U-*--xG[..G]:.522_:D (0.007) | 26 (0.010) D:.514_:[..A]A-**-xU[..U]:.508_:D (0.015)
step i1-i2 i1-j2 j1-i2 j1-j2 sum 1 GG/UC 0.90( 0.00) 0.00( 0.00) 0.02( 0.00) 1.32( 0.00) 2.24( 0.00) 2 GC/GU 6.86( 4.14) 0.00( 0.00) 0.00( 0.00) 6.65( 4.06) 13.51( 8.20) 3 CC/GG 0.74( 0.16) 0.00( 0.00) 0.19( 0.00) 2.64( 1.17) 3.57( 1.33) 4 CC/GG 0.40( 0.00) 0.00( 0.00) 0.07( 0.00) 4.01( 2.73) 4.48( 2.73) 5 CC/GG 0.65( 0.17) 0.00( 0.00) 0.17( 0.00) 2.62( 1.17) 3.44( 1.34) 6 CA/UG 0.59( 0.00) 0.00( 0.00) 1.37( 0.63) 0.00( 0.00) 1.96( 0.63) 7 AG/CU 0.00( 0.00) 0.00( 0.00) 0.14( 0.00) 1.67( 0.48) 1.81( 0.48) 8 GG/CC 2.59( 1.07) 0.00( 0.00) 0.75( 0.00) 0.00( 0.00) 3.34( 1.07) 9 GG/CC 3.42( 1.95) 0.00( 0.00) 0.45( 0.00) 0.60( 0.00) 4.47( 1.95) 10 GG/CC 5.04( 3.70) 0.00( 0.00) 0.01( 0.00) 0.42( 0.00) 5.46( 3.70) 11 GG/CC 4.11( 2.49) 0.00( 0.00) 0.20( 0.00) 0.00( 0.00) 4.31( 2.49) 12 GU/AC 7.24( 4.67) 0.00( 0.00) 0.00( 0.00) 3.97( 1.25) 11.21( 5.92) 13 UU/GA 7.23( 4.29) 0.00( 0.00) 0.00( 0.00) 5.20( 2.93) 12.43( 7.22) 14 UA/CG 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 15 AG/CC 4.07( 2.23) 0.00( 0.00) 0.00( 0.00) 5.64( 2.61) 9.71( 4.84) 16 GG/CC 2.76( 1.22) 0.00( 0.00) 0.34( 0.00) 0.60( 0.01) 3.70( 1.22) 17 GC/GC 5.91( 2.77) 0.00( 0.00) 0.00( 0.00) 5.69( 2.76) 11.59( 5.53) 18 CC/GG 0.11( 0.00) 0.00( 0.00) 0.81( 0.00) 3.36( 1.93) 4.28( 1.93) 19 CG/CG 0.03( 0.00) 0.00( 0.00) 5.06( 2.27) 0.00( 0.00) 5.09( 2.27) 20 GA/GC 2.60( 0.44) 0.00( 0.00) 0.00( 0.00) 4.42( 1.97) 7.02( 2.41) 21 AG/CG 0.00( 0.00) 0.00( 0.00) 0.81( 0.00) 1.37( 0.85) 2.17( 0.85) 22 GU/AC 5.14( 2.34) 0.00( 0.00) 0.00( 0.00) 3.26( 1.87) 8.40( 4.21) 23 UC/GA 0.28( 0.00) 0.00( 0.00) 0.00( 0.00) 3.76( 2.35) 4.04( 2.35) 24 CU/GG 1.38( 0.05) 0.00( 0.00) 0.00( 0.00) 2.90( 1.40) 4.27( 1.45) 25 UA/UG 0.00( 0.00) 2.56( 0.19) 6.17( 4.48) 0.00( 0.00) 8.73( 4.67) 26 AG/CU 4.86( 2.31) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 4.86( 2.31) 27 GG/CC 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00)Максимальное перекрывание у пары 2 GC/GU
