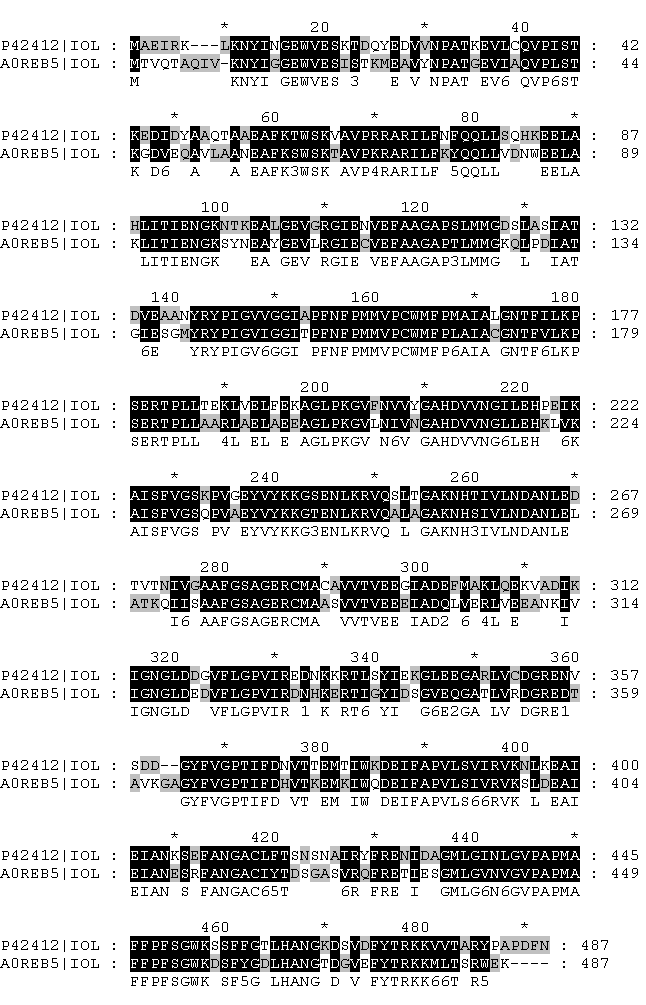
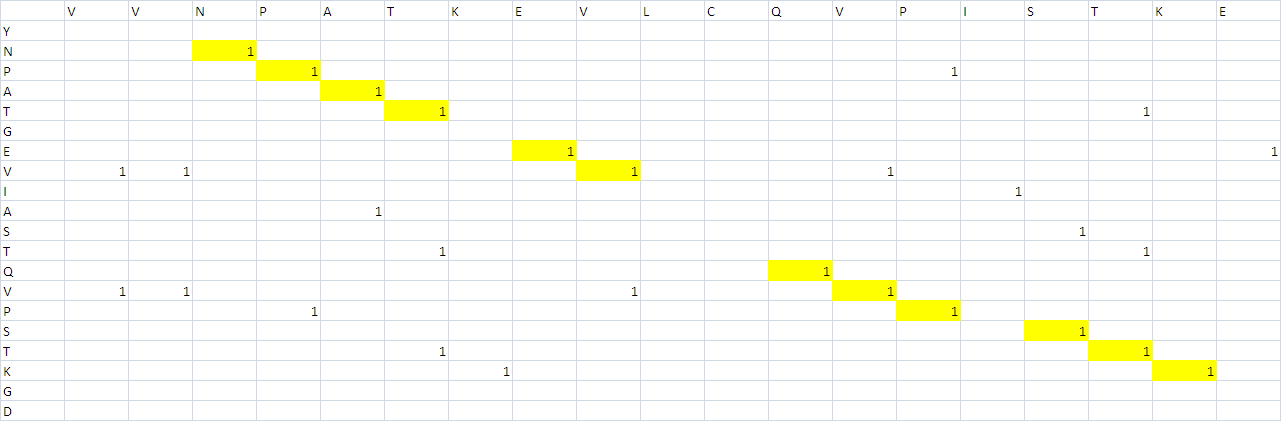
1. Выравнивание фрагментов последовательности двух родственных белков.
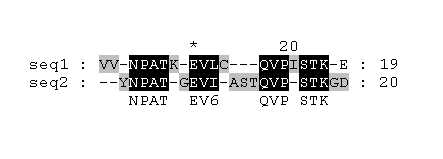
Процент идентичности 48%.
Процент сходства 56%.

| ID | Iola_Bacsu | Iola2_Bacah |
| Организм | Bacillus subtilis | Bacillus thuringiensis |
| Процент идентичности ("Identities") | 331/477 (69%) | |
| Процент сходства ("Positives") | 395/477 (83%) | |
| Число символов разрыва (гэпов) | 2 | |
| Число разрывов - идущих подряд символов разрыва | 1 | 0 |
| Суммарное число гэповых колонок | 2 | |
| 7-66 67-126 127-186 187-246 247-306 307-364 365-424 425-481 | 9-68 69-128 129-188 189-248 249-308 309-368 369-428 429-485 | |