Пример использования трехмерного QSAR анализа для предсказания активности низкомолекулярных соединений в отношении данного белка.
1. Для проведения 3DQSAR анализа мы будем использовать программы Open3DQSAR и Open3DALIGN (open3dqsar.sourceforge.net).
export PATH=$PATH:/home/preps/grishin/open3dtools/bin
Эти программы поддерживают как работу в интерактивном режиме, так и выполнение скриптов.
2. Теперь необходимо сделать выравнивание полученных конформеров.
Перекодируем в ascii. Получили aligned_ok.sdf.
Далее запустим 33DQSAR для регрессионной модели: pls
Exp. Cum. exp. Exp. Cum. exp.
PC var. X % var. X % var. Y % var. Y % SDEC r2
--------------------------------------------------------------------------
0 0.0000 0.0000 0.0000 0.0000 0.9494 0.0000
1 15.9480 15.9480 32.8386 32.8386 0.7780 0.3284
2 5.1333 21.0813 36.3625 69.2011 0.5269 0.6920
3 4.6235 25.7048 15.6991 84.9002 0.3689 0.8490
4 3.8908 29.5956 7.5246 92.4248 0.2613 0.9242
5 4.0108 33.6064 2.8661 95.2909 0.2060 0.9529
Как видно, модель сработала довольно неплохо.
Далее кросс-валидация: cv type=loo runs=20
PC SDEP q2
--------------------------
0 0.9658 -0.0348
1 0.9164 0.0683
2 0.9733 -0.0509
3 0.9667 -0.0368
4 0.9880 -0.0829
5 0.9497 -0.0006
А затем предскажем активности для тестовой выборки: predict
PC r2(pred) SDEP
--------------------------
0 0.0000 1.0362
1 0.2655 0.8881
2 0.3296 0.8484
3 0.2353 0.9061
4 0.2754 0.8821
5 0.2536 0.8953
3. Анализ, используя выравнивание и конформации, полученные с учетом структуры активного центра белка-мишени
Регрессионный анализ:
Exp. Cum. exp. Exp. Cum. exp.
PC var. X % var. X % var. Y % var. Y % SDEC r2
--------------------------------------------------------------------------
0 0.0000 0.0000 0.0000 0.0000 0.9494 0.0000
1 12.1342 12.1342 48.4736 48.4736 0.6815 0.4847
2 13.2295 25.3637 14.5885 63.0621 0.5770 0.6306
3 7.6412 33.0049 13.2040 76.2661 0.4625 0.7627
4 8.0257 41.0305 4.3684 80.6345 0.4178 0.8063
5 6.0521 47.0827 3.8642 84.4987 0.3738 0.8450
Кросс-валидация:
PC SDEP q2
--------------------------
0 0.9658 -0.0348
1 0.8027 0.2851
2 0.7664 0.3484
3 0.7061 0.4468
4 0.6735 0.4968
5 0.6401 0.5454
Предсказание:
PC r2(pred) SDEP
--------------------------
0 0.0000 1.0362
1 0.3451 0.8385
2 0.3226 0.8529
3 0.2998 0.8671
4 0.3012 0.8662
5 0.2693 0.8858
Как видно, значения r2 стали лучше.
4. Предскажем активности:
1) Переделаем модель с использованием всех имеющихся данных, а вещества с неизвестной активностью обозначим как тестовую выборку:
set object_list=60-85 attribute=TRAINING
set object_list=86-88 attribute=TEST
2) Построение модели:
Exp. Cum. exp. Exp. Cum. exp.
PC var. X % var. X % var. Y % var. Y % SDEC r2
--------------------------------------------------------------------------
0 0.0000 0.0000 0.0000 0.0000 0.9749 0.0000
1 12.8375 12.8375 44.4004 44.4004 0.7269 0.4440
2 14.5264 27.3638 14.3748 58.7753 0.6260 0.5878
3 6.9607 34.3245 11.2007 69.9760 0.5342 0.6998
4 8.4659 42.7904 5.4939 75.4699 0.4828 0.7547
5 4.7600 47.5503 5.7466 81.2166 0.4225 0.8122
3) Предсказание:
External predictions for dependent variable 1 (activity)
--------------------------------------------------------------------------------------------------------------------------------------
N ID Name Actual 1 2 3 4 5 Opt PC n
--------------------------------------------------------------------------------------------------------------------------------------
86 86 01 0.0000 7.0954 7.5090 7.3772 7.6623 7.8822 1
87 87 44 0.0000 6.9300 7.0808 6.9883 7.1990 7.4119 1
88 88 72 0.0000 5.5493 5.2836 5.1285 5.3788 5.3537 3
PC r2(pred) SDEP
--------------------------
0 0.0000 6.6604
1 0.0294 6.5616
2 -0.0102 6.6942
3 0.0265 6.5717
4 -0.0480 6.8183
5 -0.0950 6.9696
4) Кросс-валидация:
PC SDEP q2
--------------------------
0 0.9865 -0.0240
1 0.8305 0.2743
2 0.7666 0.3816
3 0.7394 0.4247
4 0.7339 0.4332
5 0.7380 0.4269
Пусть предсказание будет соответствовать 4ой компоненте (лучше проявила себя в кросс-валидации).
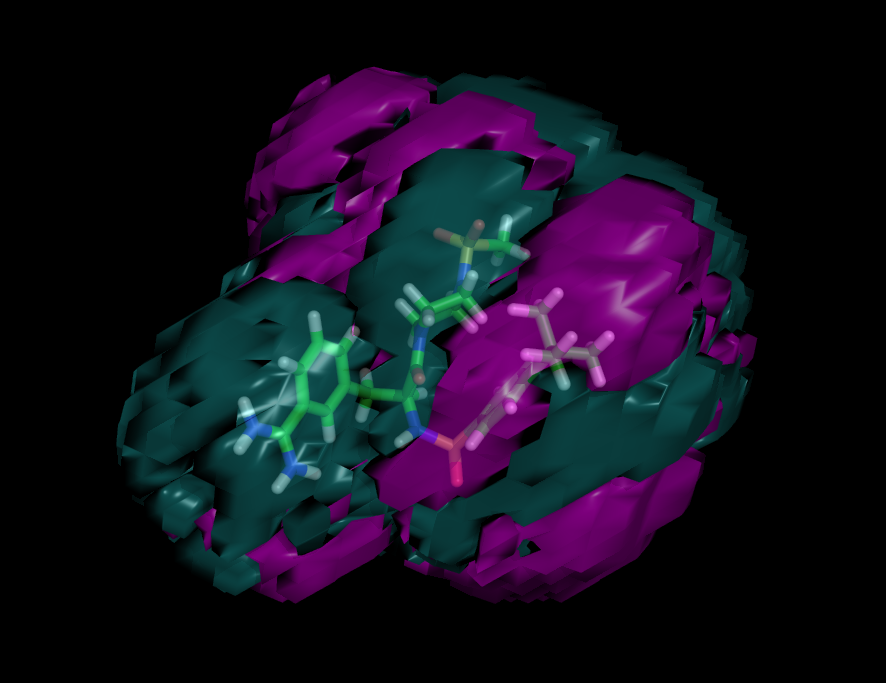
Теперь сделаем модель в Pymol о влиянии заместителей на активность соединений:
1) Используем команду isosurface positive, coefs_fld-01_y-01, 0.00001 и покрасим в розовый цвет полученную положительную поверхность и команду isosurface negative, coefs_fld-01_y-01, -0.00001 , отрицательную поверхность красим в циановый.
2) Используем команду set transparency, 0.5, чтоб изменить прозрачность поверхности.
Получаем такое изображение:
© Karavaeva Julia 2009
