Химическое строение нуклеиновых кислот. MarvinSketch
C помощью инструментов программы 3DNA, используя команду fiber, были построены структуры A, B и Z-формы ДНК. Для A- и B- формы в одной из нитей была повторена 5 раз последовательность "gatc", для Z- формы по умолчанию был задан повтор последовательности "gc". Полученные последовательности A- и B- форм были сохранены в файлах gatc-a и gatc-b, а структура дуплекса в Z-форме была сохранена в файле gatc-z.
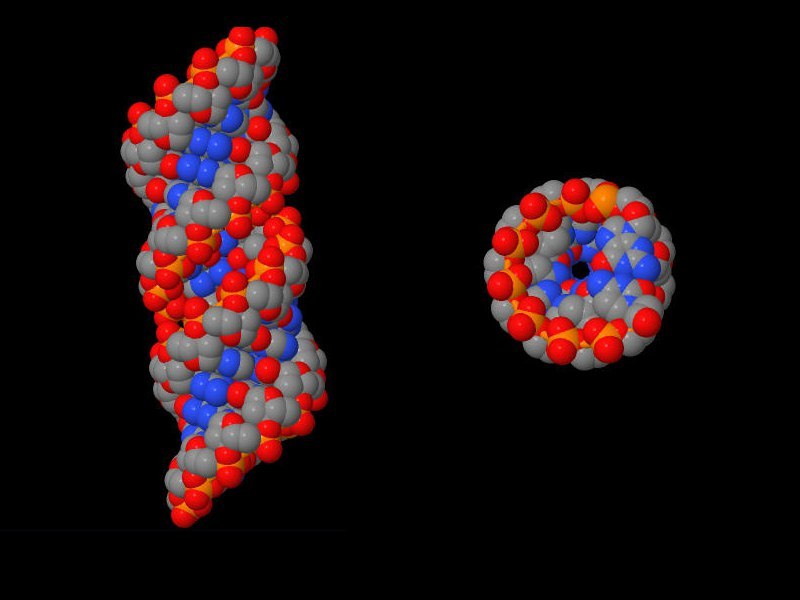
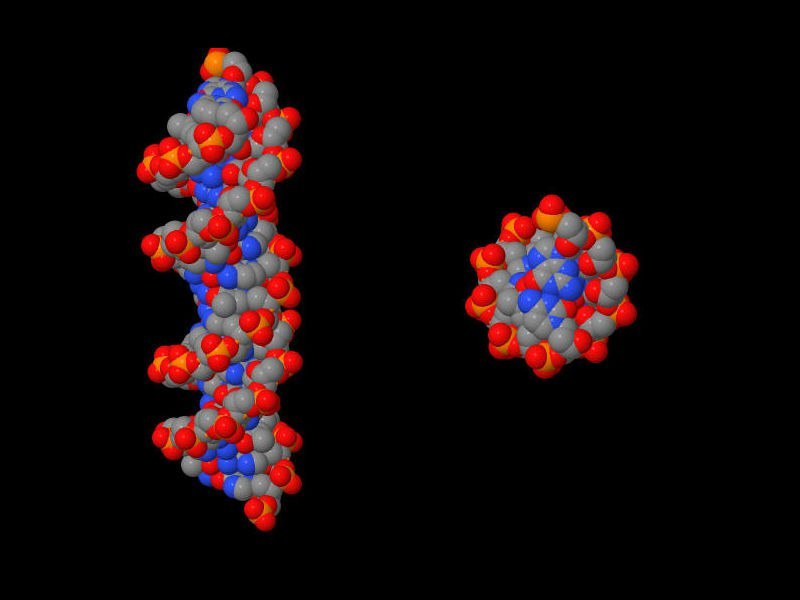
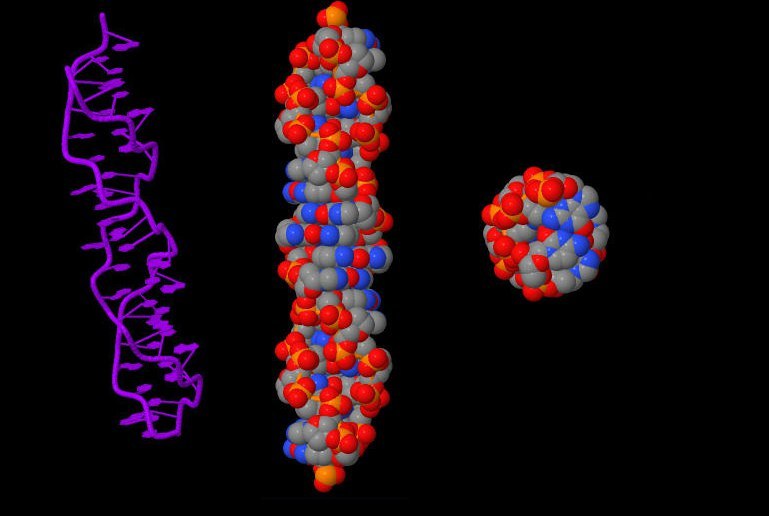
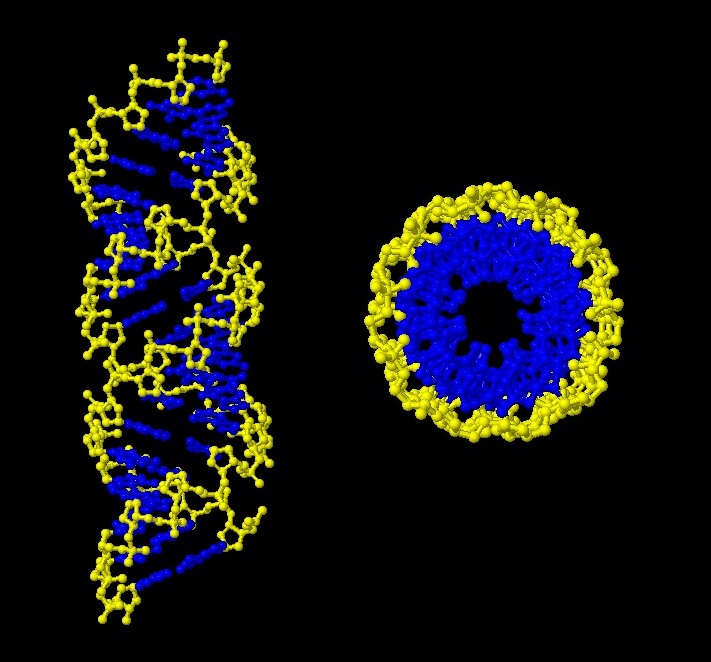
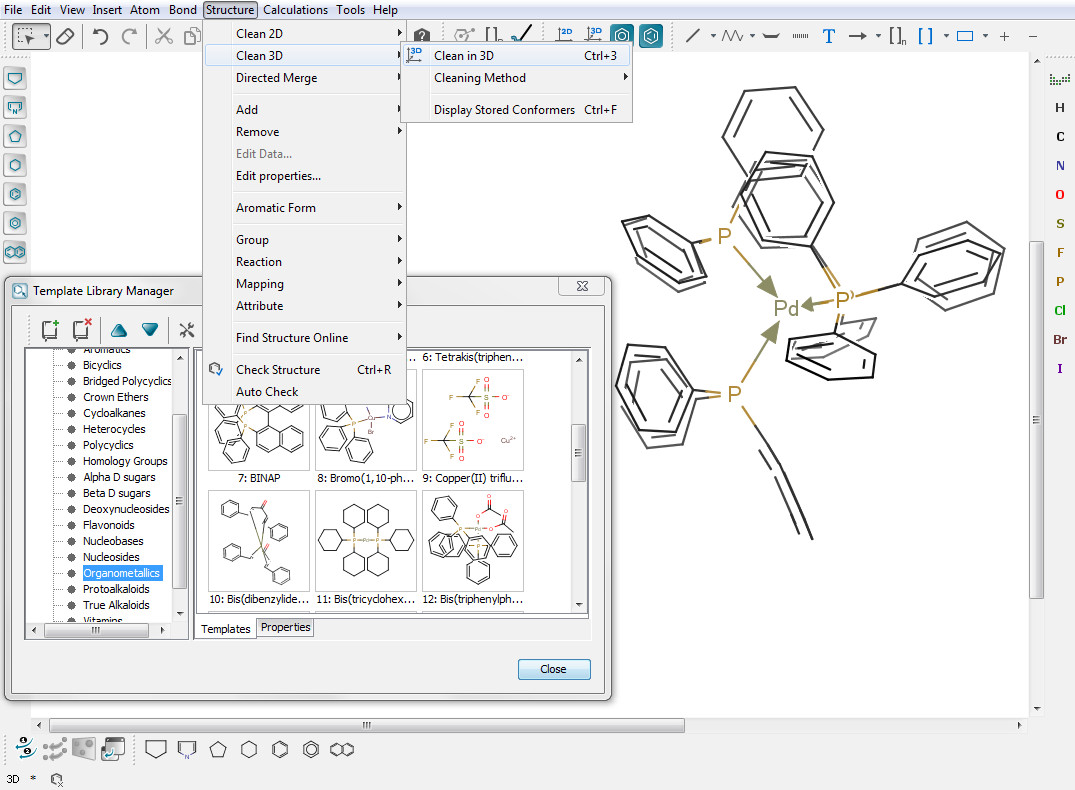
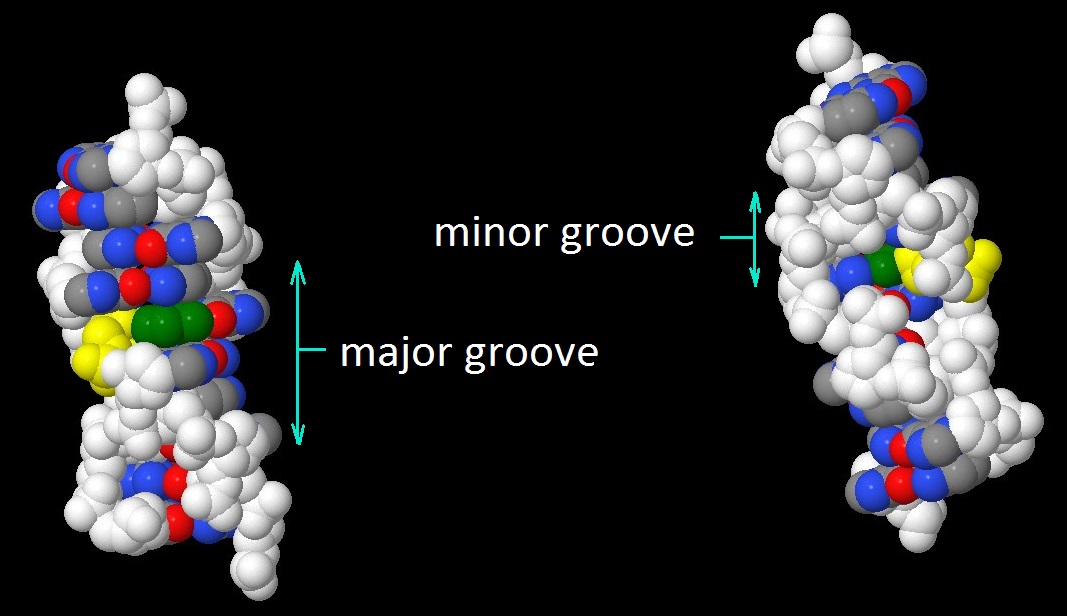
Сахарофосфатный остов ДНК выделен жёлтым, а основания синим. Jmol-команда select backbone.
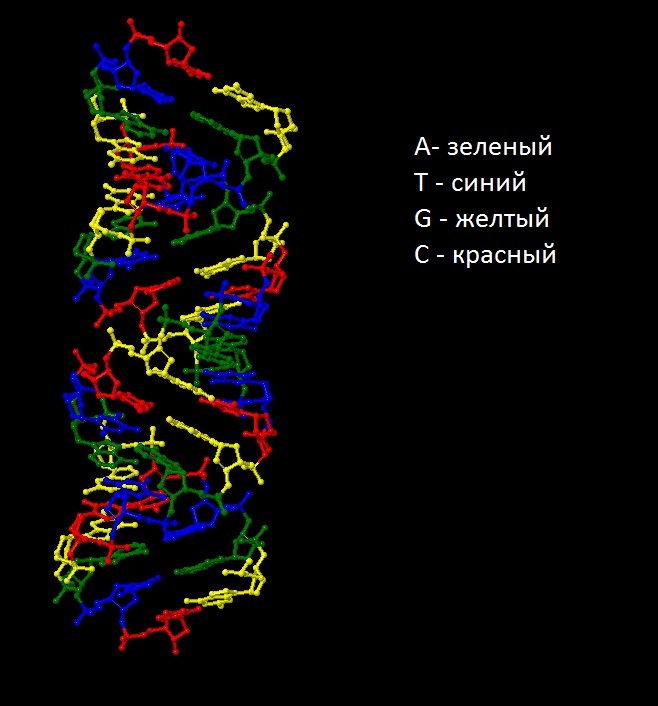
Нуклеотиды ДНК.
Выделенные цитозины.Select all, select DC, color yellow.
Таблица 1. Особенности А, В, Z-форм ДНК
| A-форма |
B-форма |
Z-форма |
||
Тип спирали (правая или левая) |
правая | правая | левая | |
Шаг спирали (Å) | 33.75 | 28.03 |
43.50 | |
Число оснований на виток | 11 | 10 | 12 | |
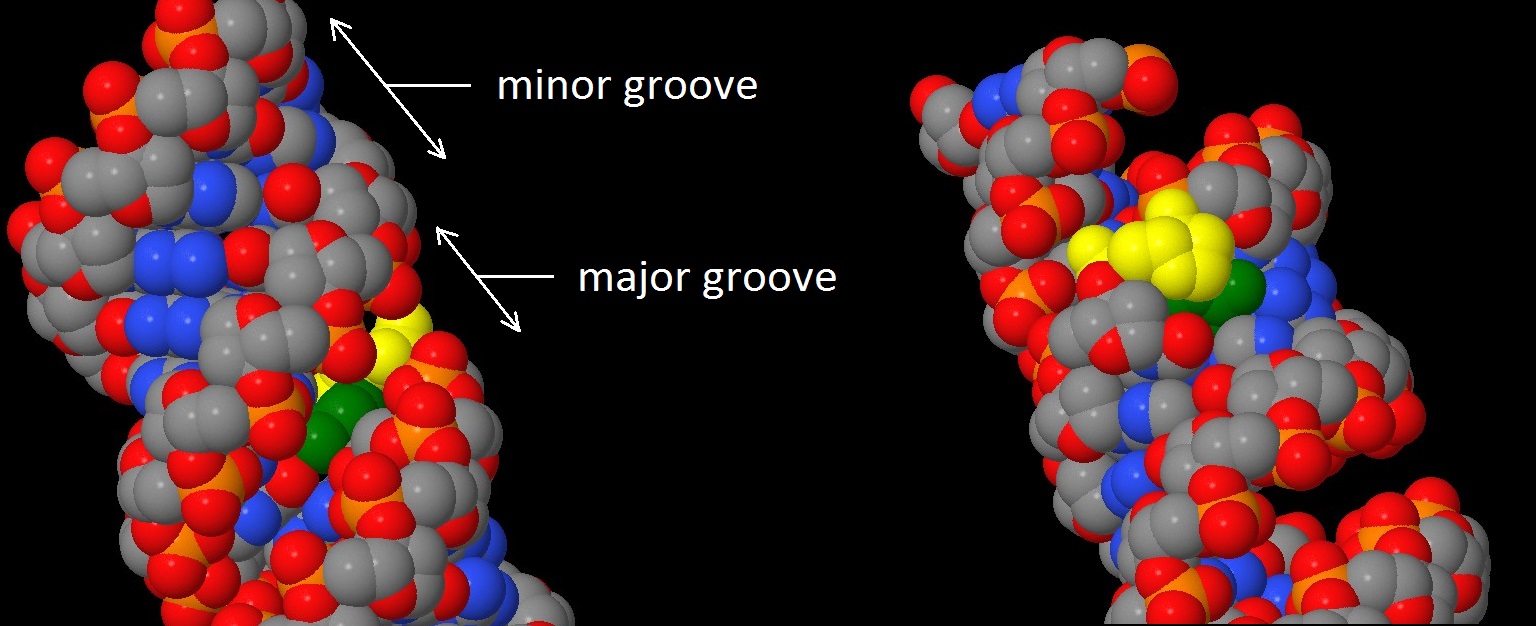
Ширина большой бороздки | 7.98([C]8:A.P-[T]27:B.P) |
17.91([C]24:B.P-[G]13:A.P) | 16.08([C]68:B.P-[C]52:A.P) | |
Ширина малой бороздки | 16.97([G]7:B.P-[T]7:A.P | 11.69([A]14:A.P-[T]31:B.P |
7.2([G]69:B.P-[G]55:A.P |
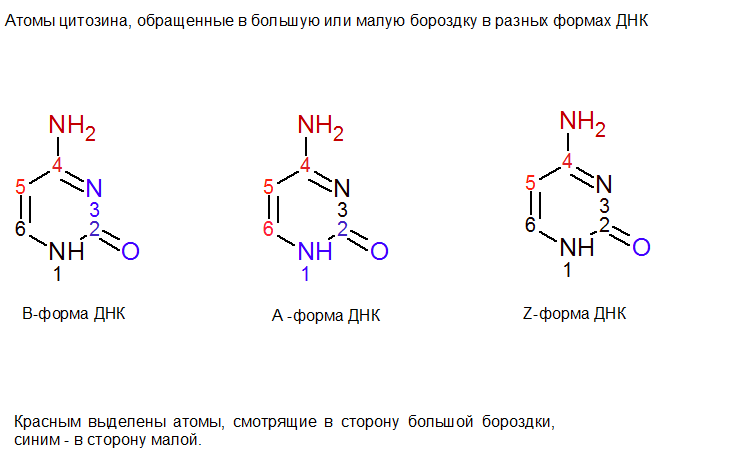
B-форма ДНК: 1)В сторону большой бороздки обращены атомы C4, C5, N4. 2)В сторону малой бороздки обращены атомы C2, O2, N3. 5 3)Остальные атомы основания (N1, C6) не обращены явно в какую-либо из бороздок и находятся в плоскости витка спирали. A-форма ДНК: 1)В сторону большой бороздки обращены атомы (глубокой) C4, C5, C6, N4. 2)В сторону малой бороздки обращены атомы O2,C2,N1. 3)Оставшийся атом основания - N3, - не обращен явно в какую-либо из бороздок и находится в плоскости витка спирали. Z-форма ДНК: 1)В сторону большой бороздки обращены атомы C5, C6, N4. 2)В сторону малой бороздки обращены атомы O2. 3)Остальные атомы основания (C6, C2, N1, N3) не обращены явно в какую-либо из бороздок и находятся в плоскости витка спирали.
Основания в А, В, Z-формах ДНК соответственно
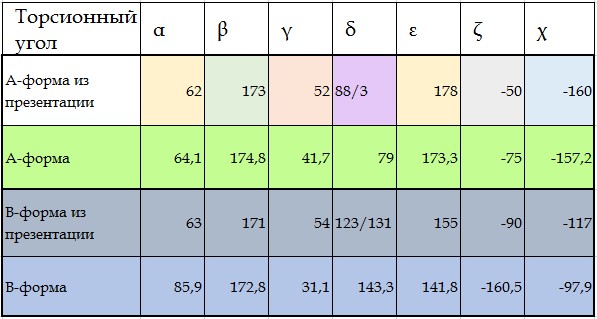
Торсионные углы цитидилового нуклеотида в разных формах ДНК.
Нужно было сравнить торсионные углы в структурах А- и В-форм. С помощью команды Settings->Torsion JMol были измерены торсионные углы нуклеотида, содержащего цитозин, в А- и В-форме.
Таблица 2.Торсионные углы цитидилового нуклеотида в разных формах ДНК. Получено с помощью Jmol
| a | b | g | d | e | ζ | ξ | |
| A-форма | 64.1 | 174.8 | 41.7 | 79.1 | 173.3 | -75 | -157.2 |
| B-форма | 85.9 | 172.8 | 31.1 | 143.3 | 141.8 | -160.5 | -97.9 |
Таблица 3.Торсионные углы цитидилового нуклеотида в разных формах ДНК. Получено из файлов ****.out
| a | b | g | d | e | ζ | ξ | |
| A-форма | -51.7 (62) | 174.8 (173) | 41.7 (52) | 79.1 (88/3) | -147.8 (178) | -75.1 (-50) | -157.2 (-160) |
| B-форма | -29.9 (61) | 136.3 (171) | 31.1 (54) | 143.3 (123/131) | -140.8(155) | -160.5(-90) | -97.9 (-117) |
| Z-форма | -139.5 | -136.8 | 50.8 | 137.6 | -96.5 | 82.0 | -154.3 |
Таблица 5.Торсионные углы для 1H4S (комплекс РНК-белок).Strand one.
| Base | a | b | g | d | e | ζ | ξ | |
| 1 | G | --- | -146.0 | 56.0 | 87.3 | -171.7 | -60.3 | -156.3 |
| 2 | G | 141.6 | -174.7 | -178.4 | 80.6 | -135.9 | -65.9 | -172.6 |
| 3 | A | -54.1 | 163.8 | 53.2 | 75.6 | -166.3 | -97.0 | -168.1 |
| 4 | G | 156.5 | -150.0 | -172.9 | 128.7 | --- | --- | -159.4 |
| 5 | G | --- | 146.7 | 51.6 | 84.1 | -134.3 | -73.9 | 177.1 |
| 6 | C | -65.6 | 165.8 | 53.8 | 81.9 | -154.7 | -73.9 | -171.9 |
| 7 | U | -66.3 | 177.6 | 55.7 | 83.3 | -157.0 | -13.0 | -153.8 |
| 8 | G | -137.7 | 79.1 | 173.7 | 88.7 | -128.9 | -83.0 | 175.6 |
| 9 | G | 140.9 | -141.8 | 176.1 | 81.0 | -129.1 | -79.4 | 174.0 |
| 10 | u | -62.1 | 176.5 | 45.7 | 78.8 | -128.1 | -73.1 | -162.6 |
| 11 | P | -52.9 | 163.7 | 50.7 | 78.6 | --- | --- | -149.0 |
| 12 | A | --- | -103.9 | 179.0 | 88.8 | -147.2 | -70.9 | -170.6 |
| 13 | C | -67.2 | 178.1 | 47.8 | 80.0 | -160.6 | -75.0 | -152.9 |
| 14 | G | -66.8 | 174.4 | 51.1 | 82.1 | -154.4 | -76.4 | -156.9 |
| 15 | A | -63.6 | 166.5 | 50.4 | 81.7 | -152.6 | -72.5 | -159.0 |
| 16 | G | -51.3 | 165.9 | 52.4 | 79.1 | -162.9 | -57.6 | -167.9 |
| 17 | G | 160.4 | -172.1 | 179.8 | 83.8 | -142.2 | -79.0 | 179.1 |
| 18 | G | -63.6 | 170.8 | 46.7 | 78.5 | --- | --- | -163.0 |
| 19 | G | --- | -128.9 | 71.0 | 91.6 | -153.1 | -68.4 | -171.9 |
| 20 | C | -70.2 | -179.4 | 46.6 | 82.0 | -155.0 | -69.3 | -160.6 |
| 21 | G | -59.0 | 178.1 | 43.9 | 78.6 | -159.5 | -73.4 | -154.6 |
| 22 | C | 148.9 | -155.6 | -174.5 | 85.0 | -141.5 | -77.5 | -174.1 |
| 23 | A | -71.4 | 167.8 | 64.3 | 75.7 | -152.6 | -61.5 | -174.0 |
| 24 | G | -59.7 | 177.2 | 54.1 | 87.7 | --- | --- | -150.9 |
| 25 | G | --- | -158.5 | 46.5 | 126.7 | --- | --- | -71.5 |
| average | -13.16 | 54.16 | 44.43 | 84.17 | -148.21 | -70.52 | -103.83 | |
Здесь можно посмотреть таблицу excel со значениями средних квадратичных отклонений, на основе которых был сделан вывод о "деформированном нуклеотиде" - в РНК это 22G (цепь 2),21C, 5U, 9G(цепь 1). В ДНК - 5 (цепь 1), 3С (цепь 2)
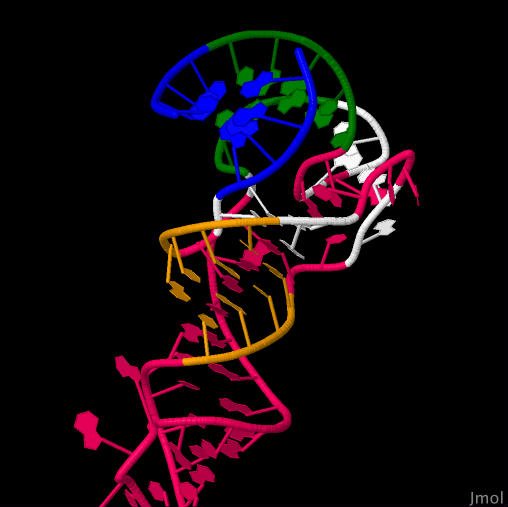
Рассмотрим вторичную структуру тРНК, ориентируемся по рисунку:
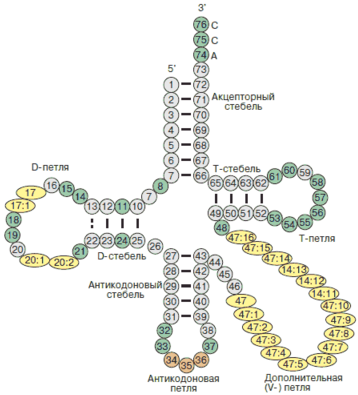
Посмотрев файл 1h4s.out, я обнаружила неканонические пары оснований - 5 G-U, 10 U-G, 10 5MU-G, 11 PSU-G, 18 G-A, 23 A-U, 24 G-C. 5MU и PSU.
Я обнаружила дополнительные водородные связи - пары 10 5MU-G, 11 PSU-G, 23 A-U, 24 G-C, 25 G-C, которые стабилизируют третичную структуру РНК.
Затем, просмотрев файл 1h4s.out, я выяснила некоторые параметры стекинг-взаимодействий. С наибольшей площадью перекрывания:
5 GC/GU 6.56( 3.49) 0.00( 0.00) 0.00( 0.00) 6.55( 3.64) 13.11( 7.12) 9 Gt/GC 7.60( 2.39) 0.00( 0.00) 0.00( 0.00) 5.65( 2.63) 13.24( 5.01) 10 tP/GG 7.22( 2.93) 0.00( 0.00) 0.00( 0.00) 6.02( 3.11) 13.24( 6.04)
С наименьшей площадью перекрывания:
11 PA/UG 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 18 GG/CA 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 24 GG/CC 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00)
Визуализация стекинг-взаимодействий с помощью stack2img
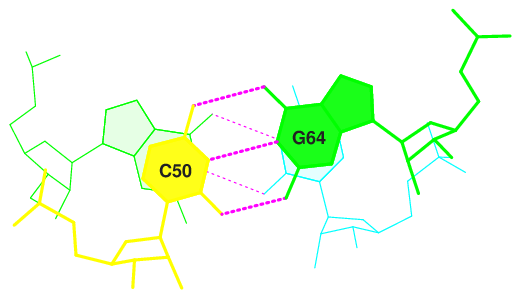
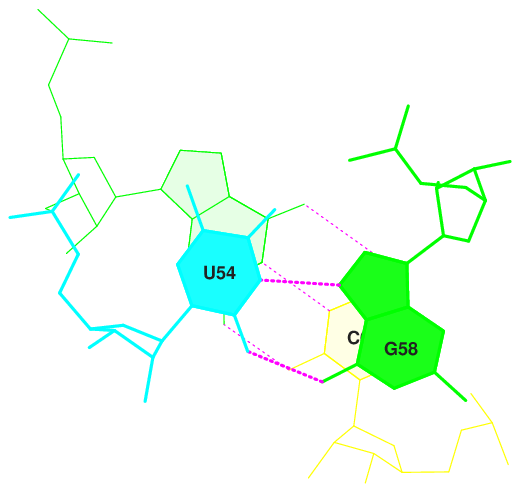
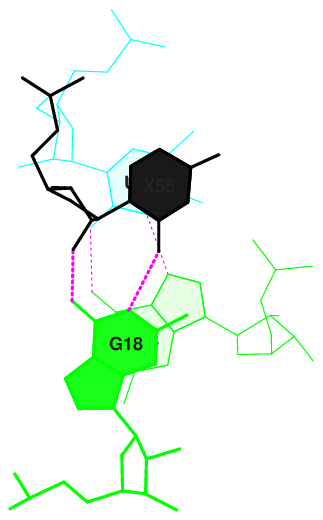
Рис.4.1.Стекинг-взаимодействия в парах с наибольшей площадью перекрывания (5,9,10).
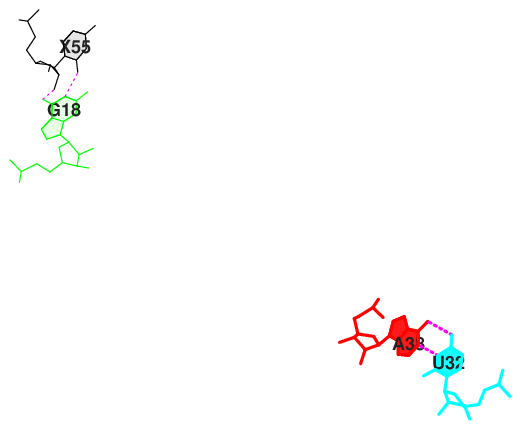
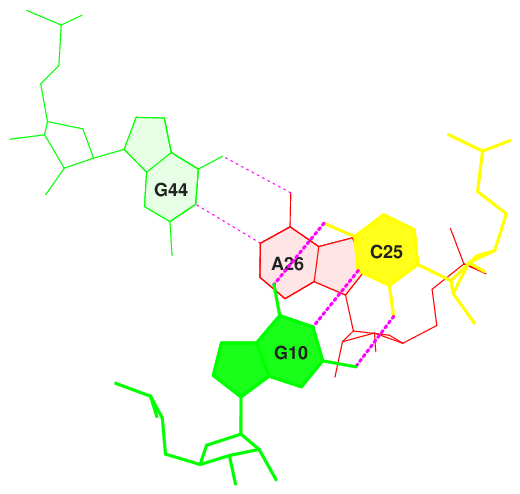
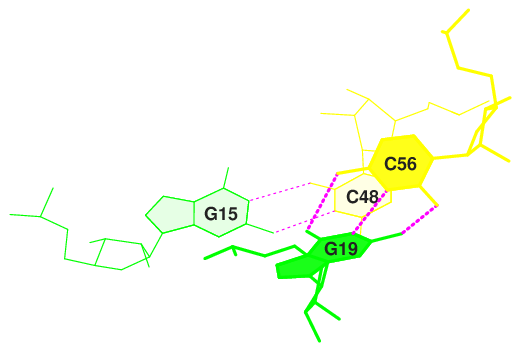
Рис.4.2.Стекинг-взаимодействия в парах с наименьшей площадью перекрывания (11,18,24).
Визуализация стекинг-взаимодействий с помощью Jmol
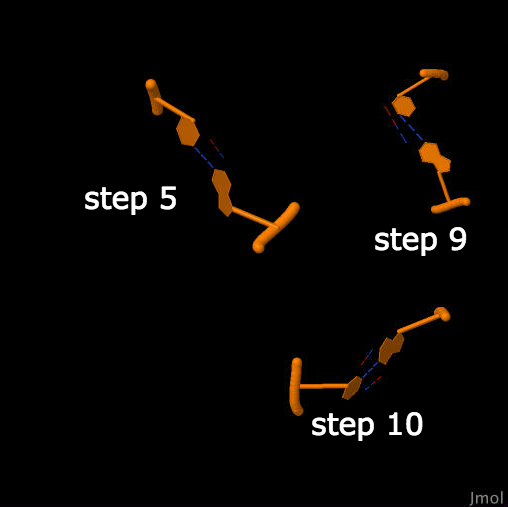
Как можно заметить из картинки, проверку не прошли пары 11 и 18.