#Получим запись заданного гена (мРНК версии) крысы в БД emblБыло обнаружено, что запись SwissProt и EMBL, во-первых, относятся к разным организмам (кишечной палочке и крысе, соответственно), а во-вторых, белки в этих двух БД транслируются с разных рамок. На лицо несоответствие.
entret embl:d89965 -auto
#Поиск всех рамок не короче 90 п.н. между старт и стоп-кодонами, результат запишем в файл d89965.fasta
getorf -minsize 90 -find 1 -sequence embl:d89965
#какой рамке соответствует занесённая в поле FT белковая последовательность?
blastp -query d89965.fasta -subject translation-FT.orf > alignment
#получим последовательность, на которую ссылается запись embl, запишем в файл hslv_ecoli.fasta
seqret sw:P0A7B8
#проверим, какой рамке соответствует запись SwissProt
blastp -query hslv_ecoli.fasta -subject trans.orf > alignment
#Скачаем из SwissProt последовательности алкогольдегидрогеназКонечный файл с аминокислотными последовательностями алкогольдегидрогеназ из заданных организмов: NEW_seqs_adh.
seqret sw:adh*_* > adh_seq.fasta
#Создадим список со ссылками на эти последовательности
infoseq -only -usa adh_seq.fasta >list_adh
#Сузим этот список при помощи скрипта
chmod +x script.sh
./script.sh
#Получим последовательности из суженного списка
seqret @NEW_list_adh NEW_seqs_adh

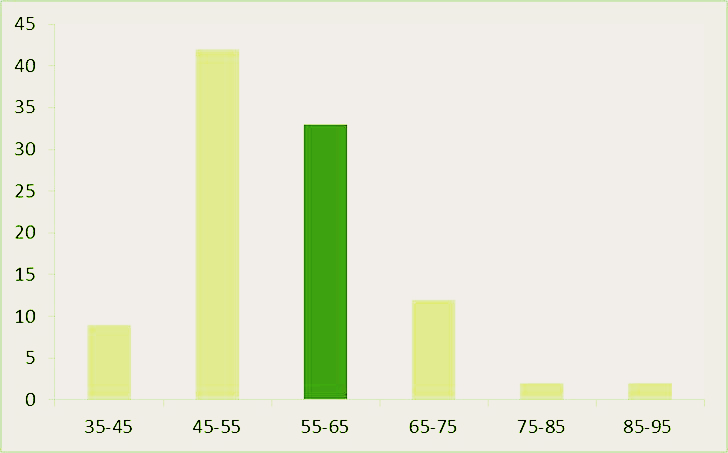
Рис. 1. Распределение весов выравниваний перемешанных последовательностей. Темнее столбик с весом неперемешанного выравнивания.
Последнее обновление: 16.09.2014