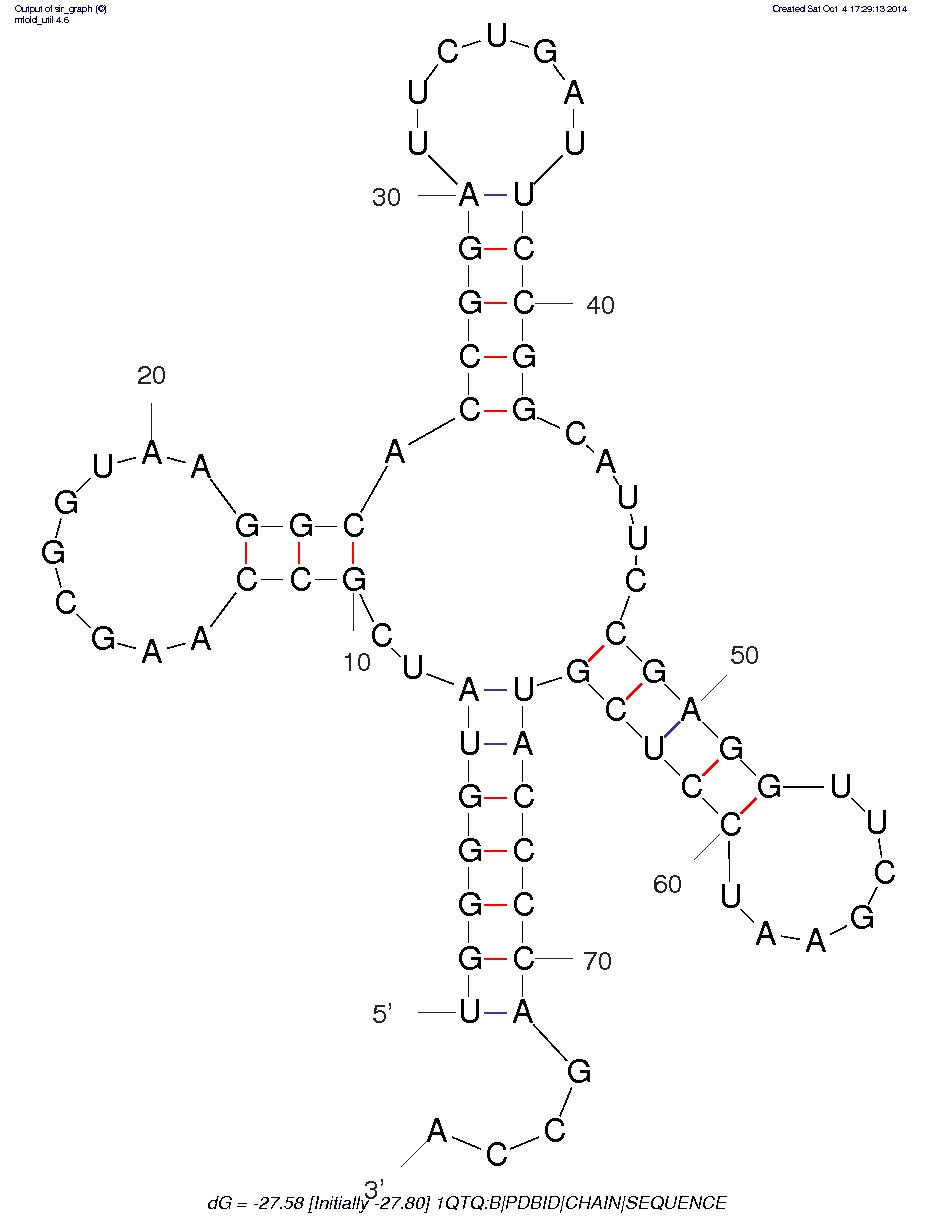
Предскажем вторичную структуры тРНК 1QTQ тремя способами:
Программа find_pair использовалась в предыдущем разделе, там же приведена ее выдача. Программа einverted и алгоритм Зукера работают с последовательностям в формате fasta. Программа einverted требует задать некоторые параметры, такие как штраф за гэп, очки за совпадения и несовпадение и т. п. Получен следующий результат (значение параметра minimum score threshold = 18, остальные параметры взяты по умолчанию):
SEQUENCE: Score 21: 7/7 (100%) matches, 0 gaps
1 tggggta 7
|||||||
71 accccat 65
В алгоритме Зукера можно менять множество разных параметров входа, но здесь имело смысл менять только параметр "P", который отражает отклонение энергии структуры от оптимального. Я воспользовался он-лайн версией программы mfold. После попыток применить разные значения параметра, я сделал вывод, что структура с подходящей конформацией получается при P=15, которая приведена ниже.

По результатам применения всех трех методов можно составить следующую таблицу.
| Участок структуры | Позиции в структуре (по результатам find_pair) | Результаты предсказания с помощью einverted | Результаты предсказания по алгоритму Зукера |
| Акцепторный стебель | 5'-902-907-3' 5'-966-71-3' Всего 6 пар |
Всего 7, предсказано 6 пар | 5'-1-7-3' 5'-65-71-3' Всего 7, предсказано 6 пар |
| D-стебель | 5'-910-912-3' 5'-923-925-3' Всего 3 пары |
Предскзано 0 пар из 3 реальных | 5'-10-12-3' 5'-22-24-3' Предсказано 3 парs из 3-x реальных |
| T-стебель | 5'-949-953-3' 5'-958-965-3' Всего 5 пар |
Предсказано 0 пар из 5 реальных | 5'-48-52-3' 5'-60-64-3' Предсказано 5 пар из 5 реальных |
| Антикодоновый стебель | 5'-926-933-3' 5'-937-944-3' Всего 8 пар |
Предсказано 0 пар из 8 реальных | 5'-26-30-3' 5'-38-42-3' Предсказано 5 пар из 8 реальных |
| Общее число канонических пар нуклеотидов | 24 | 7 | 20 |
Из таблицы видно, что алгоритм Зукера вцелом эффективнее чем программа einverted.
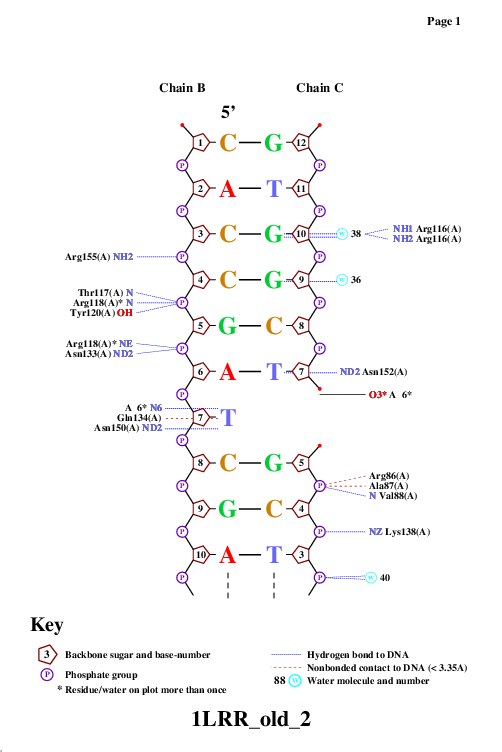
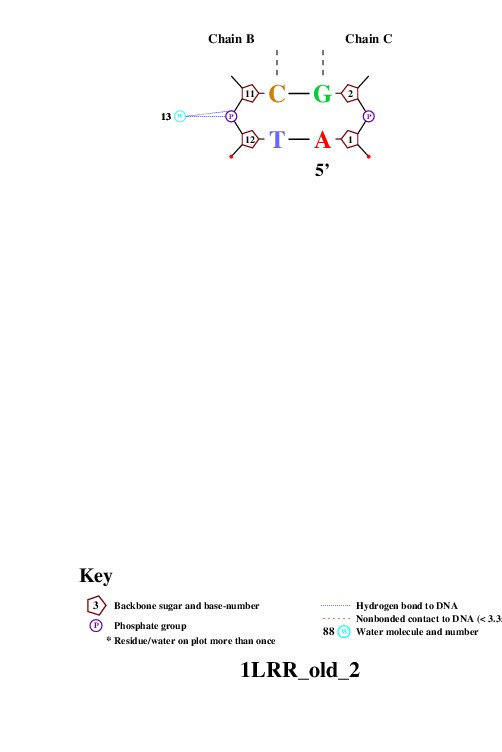
Программа Jmol позволяет работать с наборами атомов, и , в частности, позволяет фиксировать набор атомов командой define. Ниже приведен пример использования этой команды в скрипте, показывающем в структуре 1LRR полседовательно:
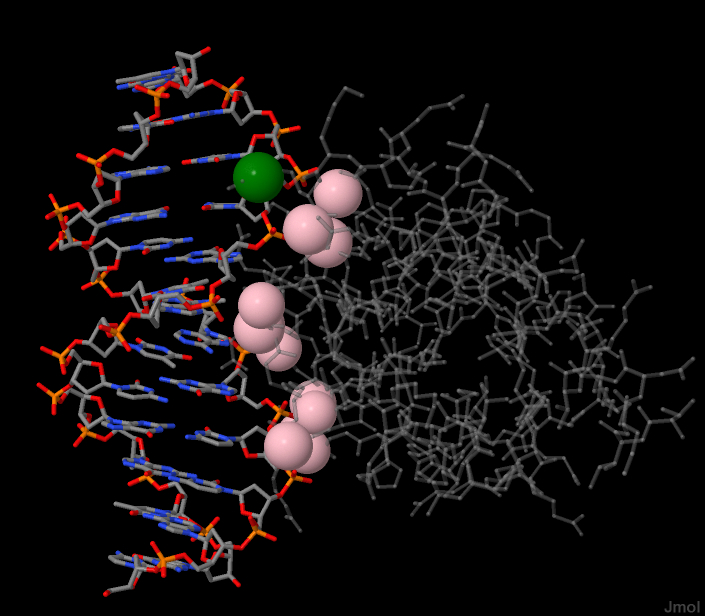
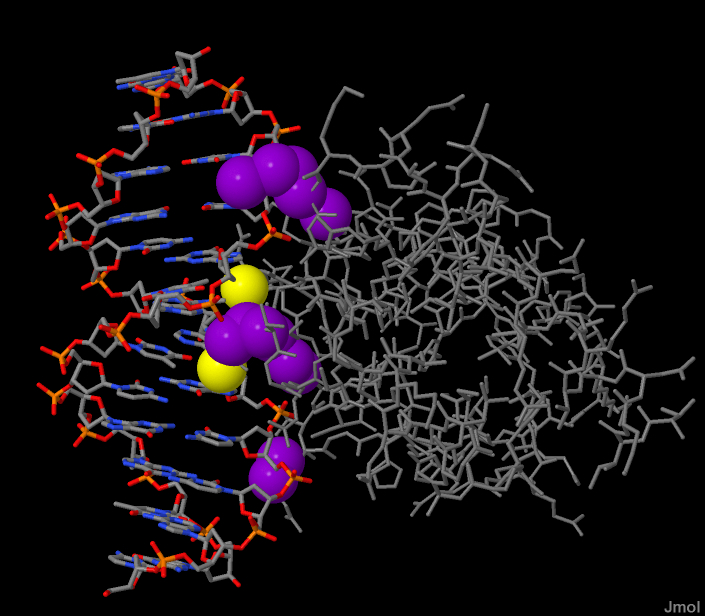
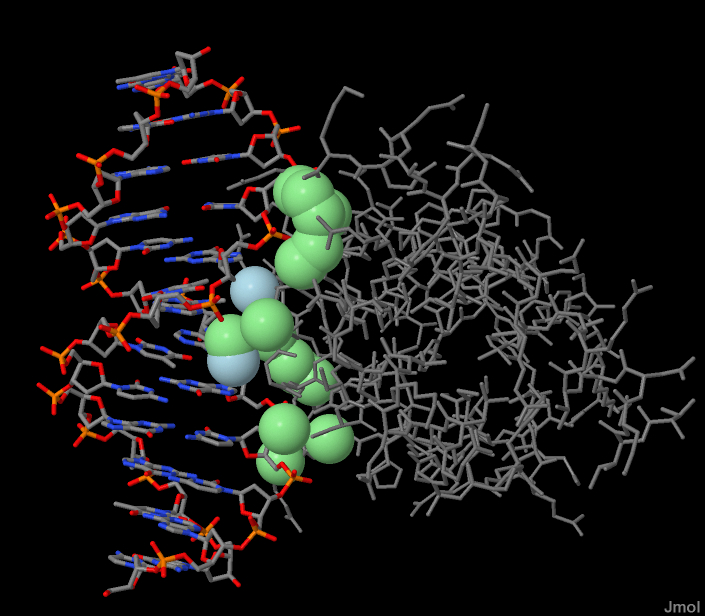
Исследуем контакты ДНК с белком в структуре 1LRR (в одной субъединице: цепи *D, *E, *F), используя команд define и select within. Классифицируем контакты на полярные и неполярные. Полярными будем счиат атомы кислорода и азота, а расстояние между ними в полярном контакте не более 3,5 ангстрем. Неполярными будем считать атомы углерода, фосфора и серы, расстояние неполярного контакта - не более 4,5 ангстрем.
| Контакты атомов белка с | Полярные | Неполярные | Всего |
| остатками 2'-дезоксирибозы | 1 | 13 | 14 |
| остатками фосфорной кислоты | 12 | 15 | 27 |
| остатками азотистых оснований со стороны большой бороздки | 2 | 2 | 4 |
| остатками азотистых оснований со стороны малой бороздки | 0 | 0 | 0 |
 |
 |
 |
| Полярные. Зеленые - с дезоксирибозой, розовые - с фосфатами. | Полярные. Желтые - атомами оснований большой бороздки. Неполярные. Фиолетовые - с дезоксирибозой. |
Неполярные. Зеленые - с фосфатами, голыбые - с атомами оснований большой бороздки, коричневые - малой бороздки. |
 |
 |
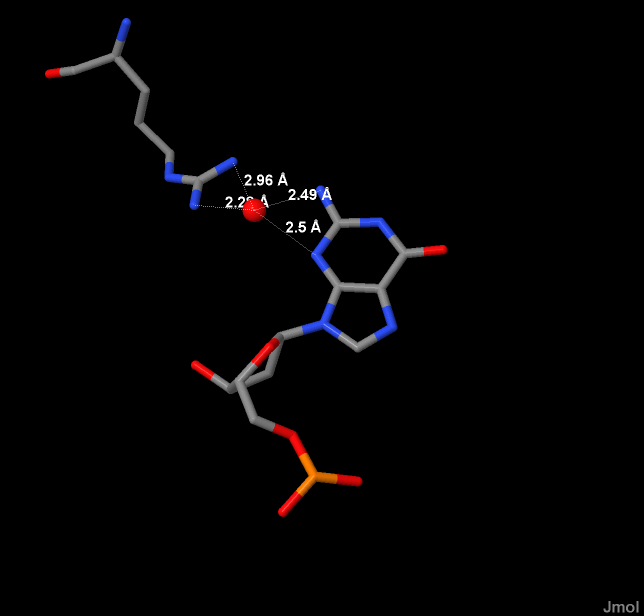
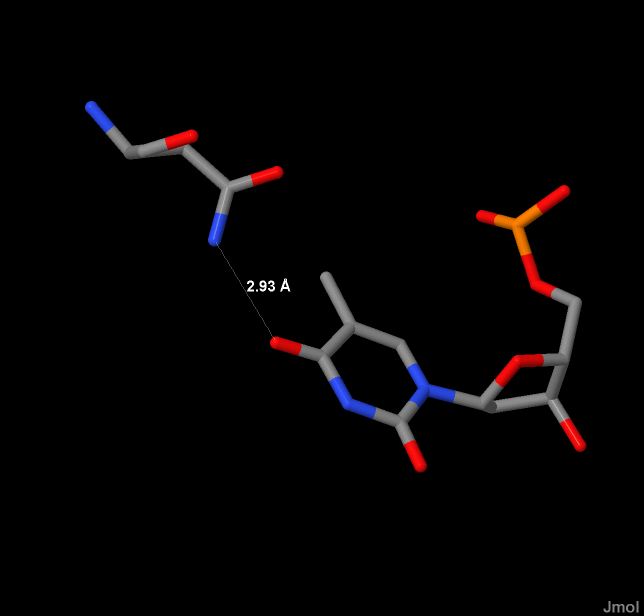
Наибольшее число контакотов с ДНК имеет Arg 116(A). Наиболее важным, по моему мнению, для распознавания последовательности ДНК является Asn 150(A), потому что он образует водородные связи через молекулу воды с азотистым основанием (а не с остовом) ДНК (тимином 7).
 |
 |
Последнее обновление: 29.09.2014