Эволюционные домены являются единицами непрерывной эволюции белков, то есть в процессе эволюции с ними просходят только локальные изменения: небольшие вставки и делеции, мутации отдельных аминокислотных остатков.
Для работы в этом задании был выбрано семейство доменов Mad3_BUB1_I (идентификатор PF08311).
Белки, содержащие этот домен, являются белками "чекпоинта", следовательно связанны с делением клетки. Было показано, что домен Mad3_BUB1_I необходим для связывания комплекса [BUB1 + MAD3] с CDC20p.
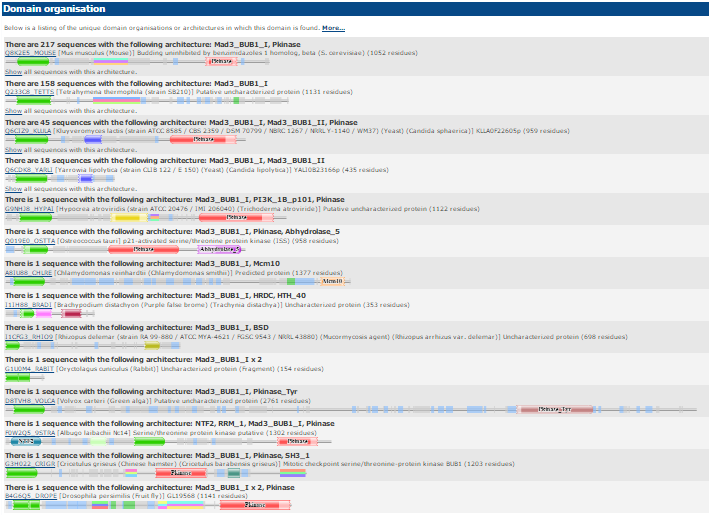
На рисунке 1 представлены все типы доменных архитектур, включающие в себя анализируемый домен Mad3_BUB1_I.

Для дальнейшей работы я выбрал две их них. Первая представлена двумя доменами: Pkinase и Mad3_BUB1_I, и включена в 217 последовательностей. Pkinase - консервативный домен, отвечающий за фосфорилирование белков, которое регулирует много клеточных процессов: метаболизм, транскрипцию, движение по клеточному циклу, реорганизацию цитоскелета, апоптоз и дифференциацию. Вторая архитектура - исходный домен Mad3_BUB1_I, включена в 158 последовательностей.
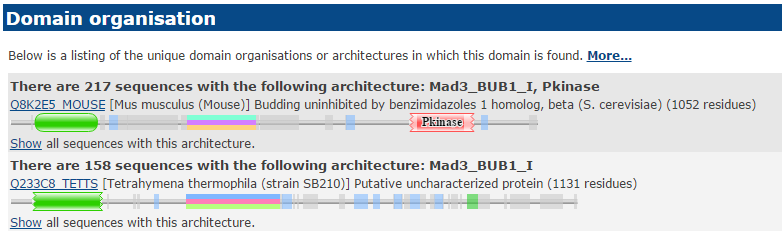
В качестве таксонов, в которых представлены домены этих белков я выбрала два одцарства (Fungi, Metazoa), общий для них таксон - Eukaryota.
Информация об архитектурах всех последовательностей, включающих выбранный домен представлена на первом листе (Domains) таблицы Excel. Данные были получены с помощью первого скрипта (swisspfam-to-xls.py), из данных по заданию.
Из этих данных были составлены доменные архитектуры для каждой последовательности. Результат был добавлен в ту же таблицу Excel на страницу 2 (Architecture), где для каждого домена (столбцы) показана встречаемость в каждой последовательности (строки). Колонка, соответсвующая исходному домену, выделена синим.
Также были посчитаны длины исходного домена в каждой архитектуре, результат представлен в таблице на второй странице (колонка "Длина").
Из базы данных Uniprot по идентификаторам были получены все последовательности, включающие домен Mad3_BUB1_I. С помощью второго из данных скриптов (uniprot-to-taxonomy.py) были получены данные о таксономии этих последовательностей. Они приведены на той же странице (Architecture) таблицы Excel.
Выравнивание всех этих последовательностей доменов можно увидеть в проекте Jalview (использовалась раскраска Clustalx с консервативностью 30%).
На странице Excel "Architecture" я отметил "+" те последовательности, архитектуры и таксоны которых соответствуют выбранным ранее. Далее убрал те последовательности, которые значительно отличались по длине от среднего значения (для Fungi ~129ак, для Metazoa ~125ак).
Затем из общего выравнивания были отобраны эти последовательности, а плоховыровненные были удалены, плюс удалены выступающие N- и C-концевые участки последовательностей. Далее произвел переименование по доменной архитектуре (цифра соответствует количеству доменов, буква - таксону). В проекте были сформированы 2 группы по количеству доменов и покрашены BLOSUM62.
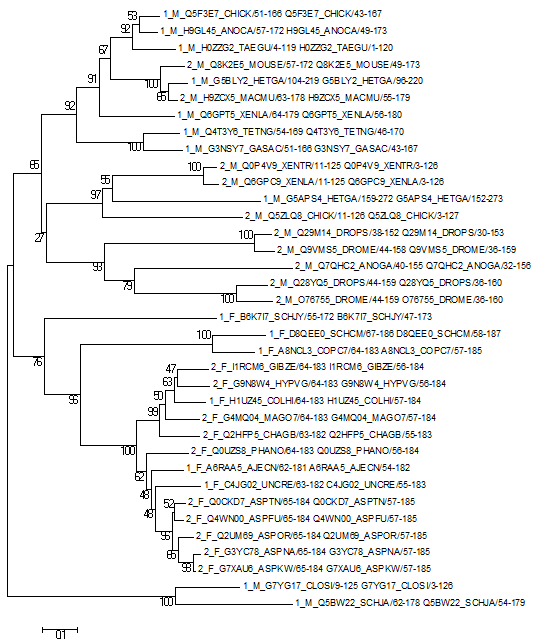
C помощью MEGA было построено дерево (методом Neighbor joining и со 100 бутстреп репликами). Полученное дерево (после удаления дубликатов) представлено на рисунке 3, а скобочную форму дерева можно посмотреть здесь.
Полное дерево было упрощено. Схематичное изображение представлено на рисунке 4.
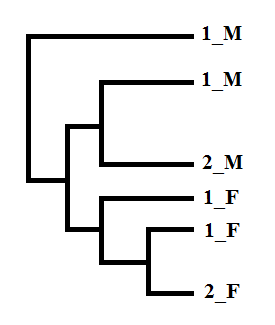
Видно, что последовательности разделились на относящиеся к Fungi и к Metazoa. Эволюционная линия формирования доменных структур одинакова для обоих таксонов. Предположительно, исходно была однодоменная архитектура. Далее возможно произошла потеря стоп-кодона между двумя рамками считывания, что привело к образованию новой двудоменной архитектуры. Однако у некоторых организмов утратился 1 домен, о чем свидетельствует дальнейшее разделение на 2 клады (1_* и 2_*).
Но есть еще один сценарий, при котором общий предок обладал двудоменной архитектурой. В местах разделения ветвей дерева белок, содержащий исходный домен, терял P-киназную активность. Но это маловероятно, так кат фосфорилирование играет важную роль в жизни клетки. Однако вероятность приобретения домена меньше, чем его трата.
В качестве подсемейства последовательностей я выбрал Fungi, так как они образуют одну кладу на дереве (рис 3). Выравнивание всех этих последовательностей (всего 27) можно посмотреть здесь. По ним с помощью программы hmm2biuld был построен профиль последовательностей и затем он был откалиброван программой hmm2calibrate. Результат работы этих двух программ можно увидеть здесь.
По этому профилю был проведен поиск по всем белкам базы данных Uniprot, включающим семейство доменов Mad3_BUB1_I с помощью программы hmm2search c параметрами по умолчанию. Всего было найдено 405 последовательностей с E_value от 2e-91 до 9,s1.
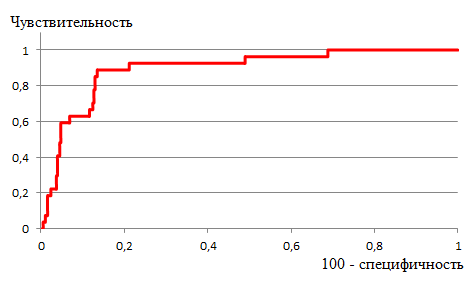
Для определения порога, по которому стоит относить последовательности к подсемейству, была построена ROC-кривая (рисунок 5) с помощью Excel. При пороге чувствительности 92,6% порог E_value составляет 2.7e-36, специфичность - 79%, характеристики представлены в таблице 1.
| На самом деле | приналежит семейству | не приналежит семейству | сумма |
|---|---|---|---|
| Выше порога по профилю | 25 | 80 | 105 |
| Ниже порога по профилю | 2 | 298 | 300 |
| Сумма | 27 | 378 | 405 |
Последнее обновление: 3.05.2015