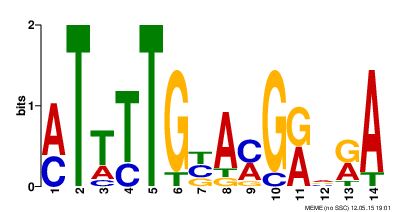
Имея набор промоторных областей генов E.coli и список экспериментально установленных сайтов связывания белка PurR, были найдены регуляторные мотив в полученных последовательностях с помощью программы MEME.
Результат работы в программе MEME представлен в этом файле.
Сначала я скачал полную последовательность ДНК бактерии Dinoroseobacter shibae DFL 12 с ftp-сервера NCBI.
По хромосомной таблице были определены координаты стартов всех генов, расположенных на "+" цепи и составлены координаты последовательностей, предположительно включающих последовательности Шайн-Дальгарно: [-35;0] позиции.
Затем, с помощью программы seqret пакета Emboss были получены сами последовательности:
| seqret @coords.txt newseq.fasta |
| ememe newsequences.fasta |
Где newsequences.fasta - это последовательности, предположительно включающие последовательности Шайн-Дальгарно, с различными идентификаторами, которые пришлось сократить до 1000, так как МЕМЕ отказывается обрабатывать болшее количество последовательностей.
В результате выполнения программы MEME была получена папка с файлами для первых 50 последовательностей, так как запущенная через kodomo программа MEME выдала странные данные. Интерес представляет LOGO, представленное на рисунке 1.
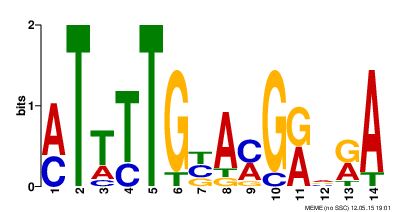
Рис. 1. Лого, простроенное программой MEME для 50 последовательностей.
Профиль представлен на html странице вывода.
Результаты так же были получены на официальном сайте MEME с теми самыми 1000 последовательностей.
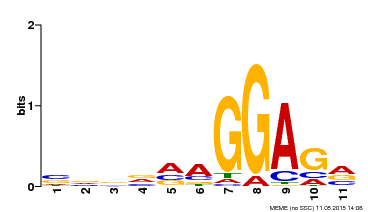
Рис. 2. Лого, простроенное программой MEME для 1000 последовательностей.
Программа MAST была запущена следующей командой:
| emast meme_out/meme.txt |
Результат представлен по ссылке.
Мною и моим однокурсником был изучен сервис PePPER. С результатом можно ознакомиться тут.
Последнее обновление: 3.05.2015