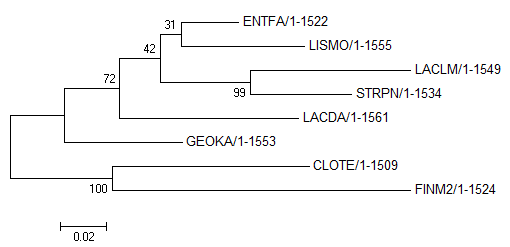
Рис.1. Maximum Likehood.
Для отобранных на предыдущем занятии бактерий требуется построить филогенетическое дерево, используя последовательности РНК малой субъединицы рибосомы (16S rRNA). Для этого сначала необходимо найти последовательности 16S рибосомальной РНК каждой из бактерий. Это можно сделать следующим способом. В записи EMBL, описывающей полный геном бактерии, найти соответствующее поле (FT с FTkey rRNA и упоминанием 16S rRNA в примечании). Затем вырезать нужный участок из записи EMBL с использованием программы seqret.
| Название | Мнемоника | AC записи EMBL | Начало | Конец | Последовательность (+/-) |
| Clostridium tetani | CLOTE | AE015927 | 176113 | 177621 | + |
| Finegoldia magna | FINM2 | AP008971 | 611796 | 613319 | + |
| Enterococcus faecalis | ENTFA | AE016830 | 248466 | 249987 | + |
| Geobacillus kaustophilus | GEOKA | BA000043 | 10421 | 11973 | + |
| Lactobacillus delbrueckii | LACDA | CR954253 | 45160 | 46720 | + |
| Listeria monocytogenes | LISMO | AL591974 | 37466 | 39020 | + |
| Streptococcus pneumoniae | STRPN | CP001845* | 15355 | 16888 | + |
| Lactococcus lactis | LACLM | AM406671 | 511423 | 512971 | + |
Полученные описанным выше образом последовательности были сохранены в соответствующем файле, при этом названия последовательностей были отредактированы так, чтобы они отвечали мнемонике организмов. Выравнивание последовательностей было получено с помощью программы JalView (Web Service > Alignment > Muscle with Defaults).
Затем выравнивание было импортировано в программу MEGA (как выравнивание нуклеотидных последовательностей), с помощью которой было построено дерево методом Maximum Likehood. Полученное дерево приведено на рисунке 1.
Как видно из сравнения с правильным деревом (приведено на рисунке 2), полученное дерево имеет с ним лишь две общие нетривиальные ветви ({CLOTE,FINM2} vs {LACDA,ENTFA,LACLM,STRPN,GEOKA,LISMO}; {LACLM,STRPN} vs {CLOTE,FINM2,LACDA,ENTFA,GEOKA,LISMO}).
Таким образом, качество реконструкции дерева по последовательности РНК малой субъединицы рибосомы оказалось ниже по сравнению с деревьями, реконструированными по белкам.
Для гомологов белка CLPX_BACSU в отобранных бактериях необходимо построить дерево.
Для поиска гомологов предложен файл proteo.fasta, содержащий записи банка Uniprot, относящиеся к исходному списку бактерий. Поиск гомологов можно произвести с помощью программы blastp, а затем отобрать находки, относящиеся к отобранным бактериям.
В файле gomolog.fasta получены последовательности гомологов CLPX_BACSU , относящиеся к отобранным бактериям.
Построить выравнивание полученных последовательностей можно, например, с помощью программы muscle:
Полученный файл с выравниванием можно импортировать в программу MEGA, чтобы построить дерево. Ниже приведено дерево, реконструированное на основе выравнивания последовательностей гомологов белка CLPX_BACSU в отобранных бактериях методом Neighbor-Joining.
Если считать, что два гомологичных белка являются ортологами, если они из разных организмов и разделение их общего предка на линии, ведущей к ним, произошло в результате видообразования, то на построенном дереве ортологами являются, например, HSLU_LACDA и HSLU_ENTFA, HLPX_LACLM и HLPX_STRPN.
Если паралогами называть два гомологичных белка из одного организма, то на основе реконструированного дерева можно сказать, что паралогами являются, например, Q891B9_CLOTE и Q899H3_CLOTE, Q1G869_LACDA и Q1GBM8_LACDA.
Пример дупликации гена на данном дереве отсутствует.
Пример видообразования изображен на картинке красным цветом