8-916-939-49-78
jiabicht@rambler.ru
vk.com/allweiss
BLOSSUM(_ _) матрицы используются для выравнивания цитоплазматических белков, а PHAT матрицы для выравнивания мембранных белков.
Цифры в названии матрицы BLOSUM62 означают порог кластеризации, т.е. последовательности из БД Blocks с идентичностью ≥ 45%, 50%, 62% и т.д. объединяются в кластеры, для них устанавливается определённый штрафной коэффициент при подсчёте пар аминокислотных остатков.
Матрицы типа BLOSUM и PHAT используют одну и ту же базу BLOCKS, но отличие в том, что BLOSUM использует цитоплазматические белки, а по матрицам PHAT для выравнивания берутся только трансмембранные белки или их участки.
Сравнение величин в восстановленной матрице, матрице BLOSUM62 и PHAT для аминокислоты Валина можно посмотреть здесь.
Обе программы, как needle, так и water, используют одинаковые обязательные параметры:
| Выравнивание белка с мутантом №1 | ||||
|---|---|---|---|---|
| Ручное выравнивание | 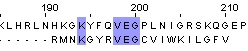
| Identity = 4/20*100% = 20% | Similarity = 8/20*100%= 40% | BLOSSUM62: 13.0 |
| Needle | 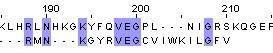
| Identity = 7/20*100% = 35% | Similarity = 12/20*100% = 60% | BLOSSUM62: 10.0 |
| Water | 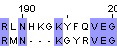
| Identity = 6/13*100 = 46.2% | Similarity = 9/13*100 = 69.2% | BLOSSUM62: 23.0 |
| Выравнивание белка с мутантом №2 | ||||
|---|---|---|---|---|
| Ручное выравнивание | 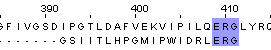
| Identity = 3/20*100% = 15% | Similarity = 8/20*100%= 40% | BLOSSUM62: 21.0 |
| Needle | 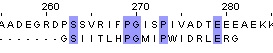
| Identity = 5/20*100 = 25% | Similarity = 9/20*100 = 45% | BLOSSUM62: 20.0 |
| Water | 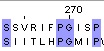
| Identity = 4/11*100 = 36.4% | Similarity = 7/11*100 = 63.6% | BLOSSUM62: 24.0 |
| Выравнивание белка с мутантом №3 | ||||
|---|---|---|---|---|
| Ручное выравнивание | 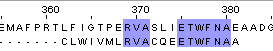
| Identity = 9/20*100% = 45% | Similarity = 11/20*100%= 55% | BLOSSUM62: 39 |
| Needle | 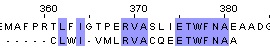
| Identity = 11/20*100% = 55% | Similarity = 12/20*100% = 60% | BLOSSUM62: 36.0 |
| Water | 
| Identity = 9/12*100% = 75.0% | Similarity = 9/12*100% = 75.0% | BLOSSUM62: 44 |