Главная страница
term3 🕒
A- и В- формы ДНК. Структура РНК
A- и В- формы ДНК. Структура РНК
TASK1. Модели структур A-, B- и Z-формы ДНК
A-форма ДНК
B-форма ДНК
Z-форма ДНК
TASK2
В сторону большой бороздки обращены атомы 8:B.N7, 8:B.C5, 8:B.C4, 8:B.N9, 8:B.C8
В сторону малой бороздки обращены атомы : 8:B.N6, 8:B.C6, 8:B.N1, 8:B.C2, 8:B.N3
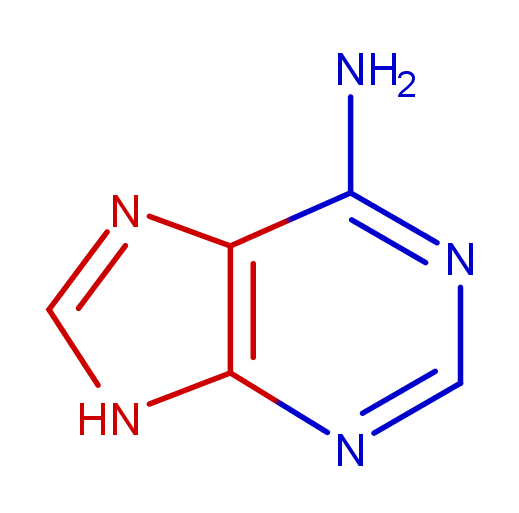
1. Adenin

| A-форма | B-форма | Z-форма | ||
| Тип спирали (правая или левая) | правая | правая | левая | |
| Шаг спирали Å | 27,6 | 36,7 | 20,62 | |
| Число оснований на виток | 11 | 10 | 12 | |
| Ширина большой бороздки Å | [DA]8:B.P;11.81 | [DC]9:A.P;14.86 | [8MG]4:A.P;11.43 | |
| Ширина малой бороздки Å | [DC]10:B.P;15.11 | [DC]9:A.P;9.77 | [8MG]10:B.P;8.19 | |
TASK3
Тяжи структуры данной тРНК похожи на А-ДНК. Это видно из ниже приведенной таблицы(для strain 1)
по наименьшей разнице между средним значением торсионных углов исследуемой тРНК и
из соответствующей таблицы, сделанной в EXCEL,а рядом находится ссылка на файл
EXCEL
| base | alpha | beta | gamma | delta | epsilon | zeta | chi | ||
| strain 1 | |||||||||
| tRNA | -47.64 | 35.91 | 64.33 | 84.18 | -137.36 | -71.67 | -138.97 | ||
| aDNA | -36.84 | 66.57 | 33.96 | 83.56 | -131.98 | -72.10 | -164.53 | ||
| Разница с тРНК | 10.8 | 30.66 | 30.37 | 0.61 | 5.373 | 0.43 | 25.55 | ||
| Среднее значение разницы | 14.8 | ||||||||
| bDNA | -64.04 | 71.18 | 65.52 | 125.45 | -2.12 | -79.19 | -115.98 | ||
| Разница | 16.4 | 35.27 | 1.18 | 41.27 | 135.23 | 7.52 | 22.9 | ||
| Среднее значение разницы | 37.125 | ||||||||
| zDNA | 46.52 | -95.62 | -0.98 | 121.13 | -118.36 | 42.24 | -49.45 | ||
| разница | 94.16 | 131.52 | 65.3 | 36.95 | 18.99 | 113.911 | 89.5 | ||
| среднее значение разницы | 78.67 | ||||||||
| base | alpha | beta | gamma | delta | epsilon | zeta | chi | |
| 1 G | --- | -163.5 | 44.9 | 81.1 | -148.1 | -79.6 | -178.1 | |
| 2 G | -61.1 | 178.0 | 49.6 | 84.4 | -141.1 | -79.5 | -172.4 | |
| 3 C | -56.7 | 166.8 | 49.4 | 77.3 | -139.4 | -82.6 | -170.6 | |
| 4 G | -63.6 | 163.7 | 59.0 | 79.1 | -167.2 | -69.5 | -169.4 | |
| 5 C | -65.4 | 179.9 | 58.0 | 81.2 | -154.4 | -84.0 | -165.8 | |
| 6 G | -54.2 | 165.9 | 58.9 | 84.8 | -160.2 | -62.0 | -166.8 | |
| 7 U | -75.8 | -175.9 | 69.4 | 116.4 | -49.4 | -104.3 | -131.9 | |
| 8 U | 71.5 | -168.5 | 53.4 | 89.2 | -140.0 | -79.5 | 180.0 | |
| 9 C | -51.8 | 163.6 | 47.9 | 81.4 | -157.1 | -67.1 | -167.1 | |
| 10 C | -71.3 | -170.9 | 48.4 | 83.8 | -162.5 | -49.1 | -154.1 | |
| 11 G | -155.1 | 148.8 | 146.0 | 88.6 | -139.4 | -63.3 | 178.6 | |
| 12 G | -72.9 | -178.5 | 55.9 | 80.4 | -167.3 | -69.5 | -162.8 | |
| 13 U | -67.9 | -179.2 | 53.0 | 83.9 | -135.6 | -56.8 | -163.5 | |
| 14 U | -66.8 | -178.6 | 46.0 | 78.0 | -146.1 | -76.3 | -142.4 | |
| 15 A | 156.5 | -176.4 | 170.7 | 82.7 | -127.4 | -75.3 | -175.1 | |
| 16 A | -78.5 | 177.2 | 53.3 | 81.5 | -156.0 | -58.2 | -158.7 | |
| 17 U | -65.6 | -179.8 | 47.3 | 79.6 | -143.6 | -89.2 | -160.3 | |
| 18 C | -54.0 | 155.8 | 57.6 | 82.0 | -149.7 | -72.5 | -162.9 | |
| 19 C | -76.6 | -174.8 | 50.4 | 82.2 | -157.5 | -73.1 | -162.7 | |
| 20 G | -54.8 | 172.1 | 46.4 | 79.0 | -152.6 | -65.9 | -171.4 | |
| 21 U | -60.8 | 174.0 | 54.8 | 80.7 | -160.3 | -99.9 | -163.6 | |
| 22 C | -53.8 | 170.9 | 52.4 | 82.5 | -138.5 | -88.2 | -173.9 | |
| 23 A | -174.4 | -174.4 | X48.9 | 82.8 | -147.2 | -77.0 | -153.9 | |
| 24 C | -70.1 | 173.6 | 51.2 | 80.0 | -148.8 | -72.8 | -160.2 | |
| 25 A | -63.4 | 172.5 | 53.4 | 82.0 | -148.3 | -64.7 | -163.5 | |
| 26 A | -56.2 | 174.0 | 51.4 | 88.2 | -178.0 | -81.3 | -149.2 | |
| 27 A | 152.8 | 179.4 | 178.9 | 87.2 | -154.6 | -78.7 | -168.2 | |
| 28 G | -64.4 | 172.3 | 52.3 | 88.2 | -151.4 | -71.4 | -156.0 | |
| 29 C | -63.7 | 167.6 | 53.6 | 81.1 | -138.6 | -80.6 | -146.0 | |
| 30 G | -155.3 | -175.1 | 76.0 | 96.7 | -59.3 | -155.0 | -124.6 | |
| 31 U | 144.1 | 152.9 | 55.8 | 83.6 | 61.5 | 105.1 | -171.7 | |
Strand II base alpha beta gamma delta epsilon zeta chi 1 C -45.7 166.2 55.5 82.0 -153.2 100.3 -160.5 2 C -57.6 174.0 48.7 77.5 -146.0 -77.3 -165.0 3 G -54.0 173.1 44.5 77.7 -151.1 -70.6 -164.1 4 C -51.8 167.7 53.1 77.3 -157.0 -81.3 -165.6 5 G -63.0 170.1 56.3 78.5 -149.4 -75.2 -166.9 6 C -70.9 176.4 54.5 80.2 -157.0 -69.2 -154.9 7 A -84.1 -160.2 52.7 88.0 -159.0 -63.4 -153.5 8 A -81.8 -170.5 63.0 84.7 -175.7 -59.6 -159.5 9 G -58.0 173.7 51.9 83.8 -166.1 -64.1 -164.7 10 G -57.7 165.7 48.8 77.7 -151.0 -74.9 -170.0 11 C -67.2 -164.6 40.7 83.3 -147.7 -79.2 -162.3 12 C -90.5 134.6 39.1 80.0 -152.4 -65.5 -176.2 13 A -58.8 -149.2 63.6 83.1 70.2 70.7 -111.4 14 G -73.8 151.5 -174.2 79.2 78.1 80.4 -152.4 15 U -71.2 177.5 62.4 85.2 -125.1 -57.3 -163.3 16 U -65.6 170.6 52.1 82.6 -151.7 -58.8 -156.1 17 A -38.1 164.8 46.7 80.1 -141.7 -59.7 -167.2 18 G -70.9 -177.7 52.4 76.5 -153.8 -77.4 -168.5 19 G -78.1 -169.6 50.0 82.8 -157.2 -63.4 -166.3 20 C -62.3 172.5 49.7 79.9 -152.7 -72.0 -166.2 21 G -55.2 -172.6 40.5 79.5 -155.9 -63.7 -164.0 22 A -69.6 175.6 55.3 77.2 -150.4 -65.8 -171.8 23 U -60.9 171.2 52.6 79.1 -145.0 -61.5 -160.5 24 G -68.2 -178.1 46.8 84.2 -150.6 -71.3 -159.6 25 U -65.1 168.3 49.7 80.9 -149.8 -79.0 -158.3 26 A 138.9 -111.7 153.0 105.3 -87.7 -67.4 -152.1 27 U -21.7 173.3 46.8 88.8 -121.1 -171.8 -152.5 28 G -38.3 140.4 54.0 87.0 71.6 -134.7 -139.5 29 C 89.1 167.0 44.3 87.1 -160.4 -38.8 -161.5 30 C 161.2 177.5 54.6 84.0 -135.2 -79.9 -170.3 31 C 147.2 172.9 55.5 81.2 -170.4 -55.8 -145.8 ****************************************************************************
TASK3.2
В исследуемой структуре тРНК 1u0b было обнаружено IV стебля:
I) 1- 7 нуклеотиды (Strand I) / 66 - 72 нуклеотиды (Strand II)
II) 49 - 53 нуклеотиды (Strand I) / 61 - 65 нуклеотиды (Strand II)
III) 39 - 42 нуклеотиды (Strand I) / 28 - 31 нуклеотиды (Strand II)
IV) 10 - 12 нуклеотиды (Strand I) / 23 - 25 нуклеотиды (Strand II)
Неканоническими парами оснований являются:
[..U]U-**--A[..A]
[..U]U-**+-G[..G]
[..A]A-**--U[..U]
[..A]A-**--U[..U]
[..U]U-*---G[..G]
[..C]C-**--A[..A]
[..A]A-**--A[..A]
[..A]A-**--U[..U]
[..G]G-**+-G[..G]
[..C]C-**+-C[..C]
[..G]G-*---C[..C]
[..U]U-**+-C[..C]
В cтруктуре тРНК отсутствуют участки, стабилизирующие третичную структуру посредством
дополнительных водородных связей в молекуле
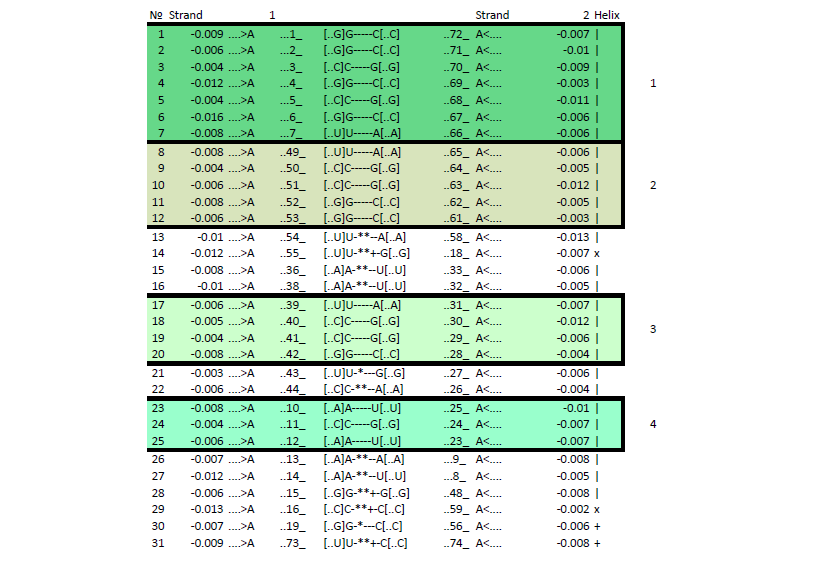
1u0b RNA

© Цыганов Кирилл, 2017