Для выполнения задания я воспользовалась программой, написанной Ксенией Кирцовой (страница с программой).
В 9 практикуме уже строила множественное выравнивание последовательностей с мнемоникой MURD (файл с последовательностями в формате fasta: jw.fasta). Взяла те же последовательности и построила выравнивания тремя разными программами (MAFFT, MUSCLE и T-Coffee).
MAFFT: запустила программу на kodomo: mafft jw.fasta > murdmafft.fasta
MUSCLE: запустила программу на kodomo: muscle -in jw.fasta -out murdmuscle.fasta
T-Coffee: построила выравнивание на сайте
Строила выравнивания не в Jalview, так как хотела посмотреть как это можно сделать по-другому (в Jalview это делается через Web Service => Alignment). Проект в Jalview со всеми выравниваниями.
Если посмотреть на таблицу из презентации к практикуму 12, то мы увидим, что из выбранных мною программ, наиболее близко к "идеальному выравниванию" строит программа MAFFT, выбрала сравнивать 2 оставшиеся программы с ней.
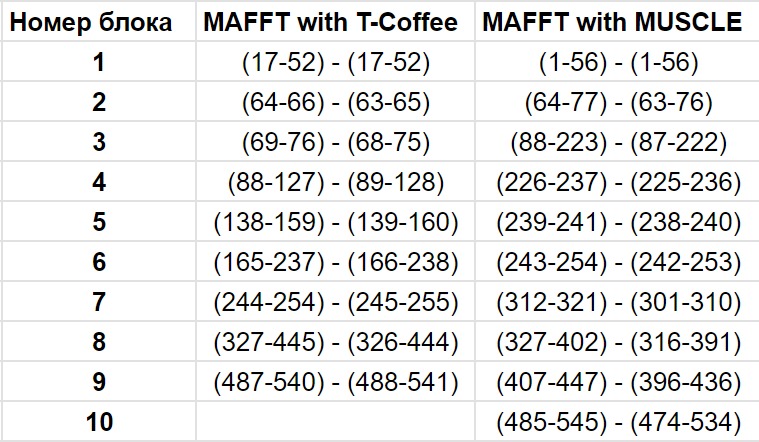
Таблица 1. Совпадающие блоки в сраниваемых выравниваниях, сделанных разными программами

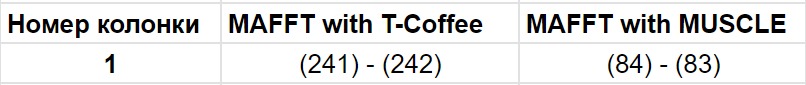
Также в каждом сравнении нашлись совпадающие одиночные колонки.
Таблица 2. Совпадающие одиночные колонки в сравниваемых выравниваниях
MAFFT with MUSCLE: Число совпадающих блоков – 10. Длина выравнивания, построенного программой MAFFT – 545, программой MUSCLE – 534. Средняя длина совпадающего блока при сравнении выравниваний с помощью программ – 42,10.
MAFFT with T-Coffee: Число совпадающих блоков – 9. Длина выравнивания, построенного программой T-Coffee – 546. Средняя длина совпадающего блока при сравнении выравниваний с помощью программ – 40,67.
Исходя из числа совпадающих блоков и средней длины совпадающего блока, можно сделать вывод, что выравнивание, построенное программой MUSCLE более близкое к референсному выравниванию, построенному программой MAFFT.
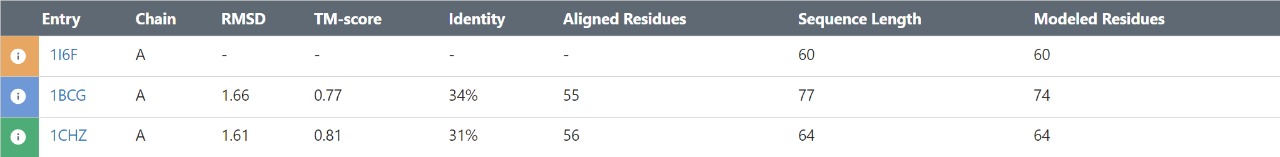
Выбрала семейство Scorpion toxin-like domain (ID: Toxin_3; AC: PF00537). В нем выбрала три белка: 1i6f, 1bcg, 1chz.
Пространственное выравнивание выполнила на сайте PDB. Референсным белком взяла 1i6f.
Таблица 3. Результат 3D выравнивания трех белков: 1i6f, 1bcg, 1chz
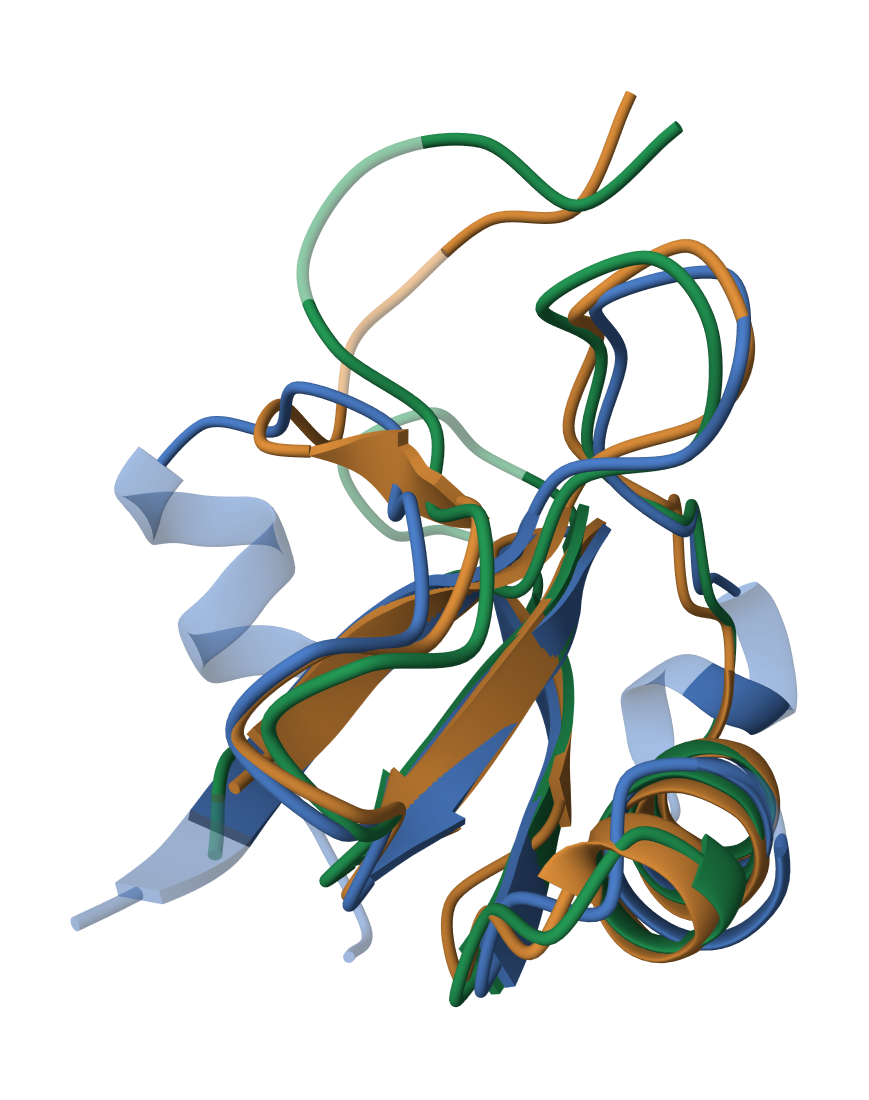
Далее объединила два выравнивания с сайта PDB в одно и построила множественное выравнивание последовательностей этих белков программой MAFFT в Jalview – проект в Jalview. Сравнила два выравнивания при помощи программы из задания 2. Длина выравнивания с сайта PDB – 83, выравнивания, сделанного программой MAFFT – 79. Процент выровненных колонок в выравнивании с сайта PDB – 77,11%, в выравнивании, сделанном программой MAFFT – 81,01%. Нашлось 6 совпадающих блоков и 2 совпадающие одиночные колонки. Если посмотреть и сравнить С-конец глазами, то видно, что теряется часть ни с чем не совпадающего участка белка 1bcg – самое критичное несовпадение.
T-Coffee (Tree-based Consistency Objective Function for Alignment Evaluation) – программа множественного выравнивания последовательностей. Впервые была представлена в 2000 году. Основана на прогрессивном выравнивании. Это была первая программа, которая внесла какие-либо значимые улучшения в алгоритм ClustalW
Алгоритм ClustalW основан на попарном выравнии последовательностей с дальнейшим построением матрицы расстояний. Эта матрица нужна для построения филогенетического древа, а затем, идя от ветвей к корню, постепенно строится множественное выравнивание. Эта программа имеет недостаток, если во время попарного выравнивания допускается ошибка, то она не исправится и распространится на остальные последовательности. T-Coffee старается минимизировать эффект таких ошибок, но при этом увеличивается время работы. Программа выполняет прогрессивное выравнивание таким образом, чтобы учитывать выравнивание между всеми парами во время генерации множественного выравнивания.
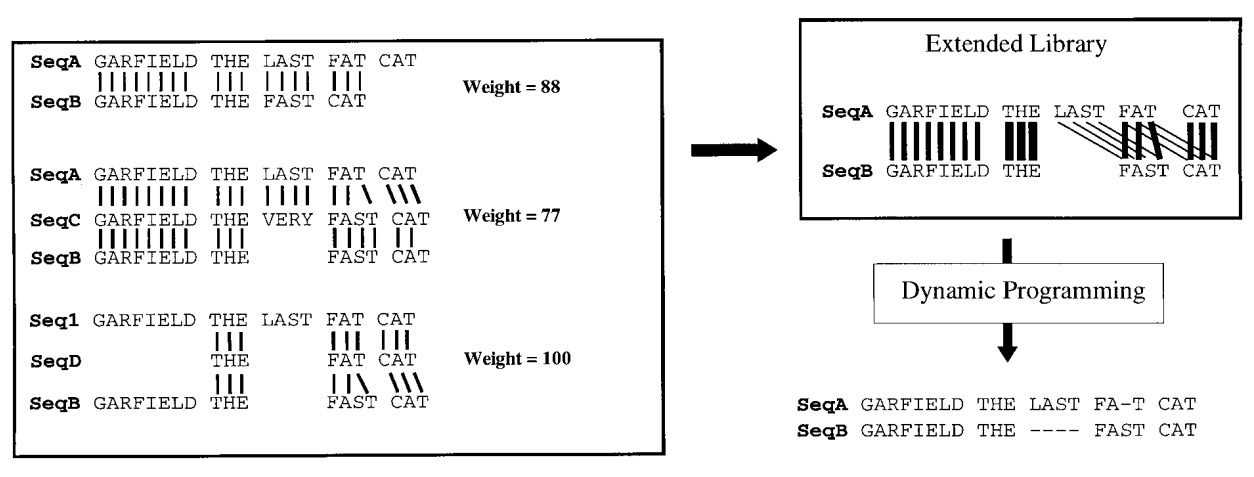
1. Создание библиотек попарных выравниваний (содержит все попарные выравнивания последовательностей; строится как глобальное попарное выравнивание, так и локальное => получаются две библиотеки: с глобальными попарными выравниваниями и с локальными попарными выравниваниями) Рис. 2. Алгоритм работы программы T-Coffee [1]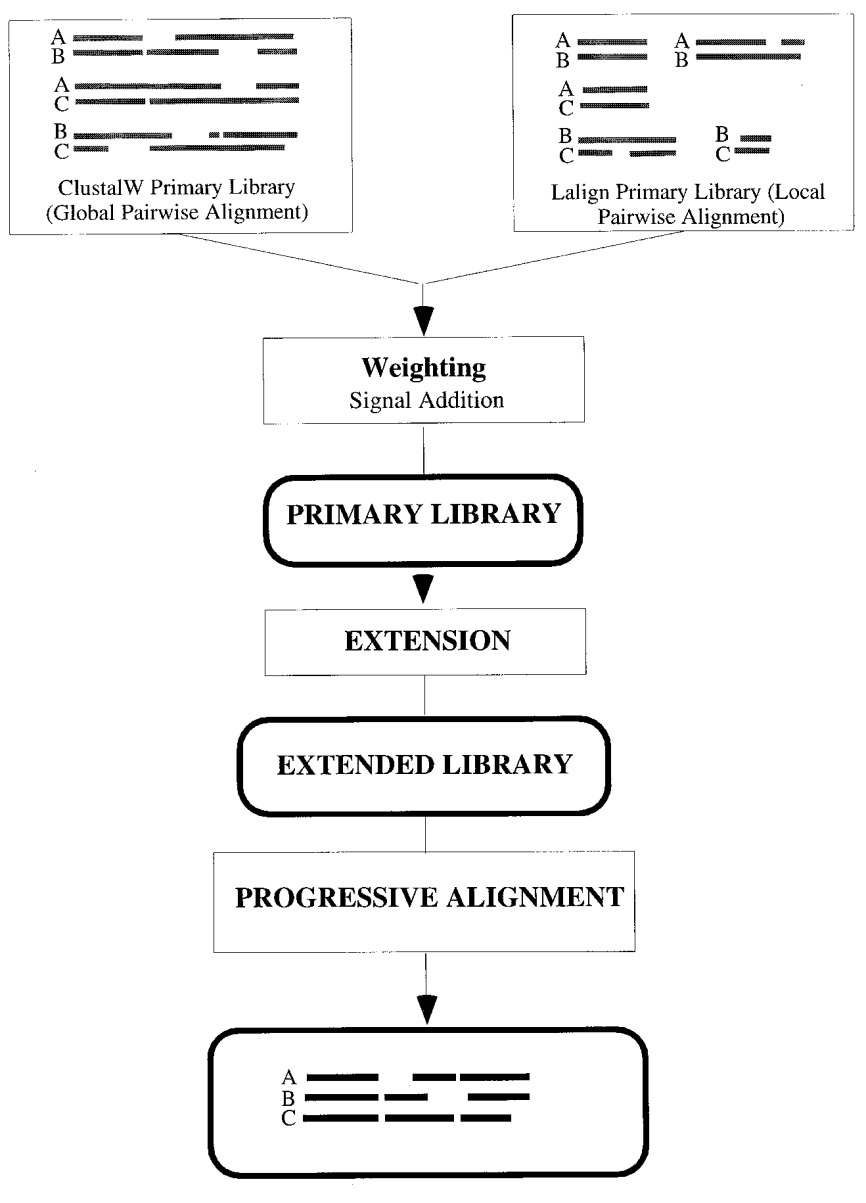
Рис. 3. Расширение библиотеки; толщина линии между парами выровненных остатков в расширенной библиотеке отмечает их "силу веса" [1]