В начале работы нашла идентификатор δ-субъединицы АТФ-синтазы в аннотации геномной сборки bColLiv1.pat.W.v2
В файле с последовательностями генома Columba livia (Сизый голубь) с аннотацией (GBFF) нашла идентификатор нуклеотидной записи, в которой аннотирован белок (δ-субъединица АТФ-синтазы): NC_088619
Последовательность белка скачала со страницы сборки генома.Далее получила последовательность гена (кодирующего δ-субъединицу АТФ-синтазы) с небольшой окрестностью.
Осуществила поиск BLAST через сайт NCBI для нахождения гомологов последовательности гена δ-субъединицы АТФ-синтазы по последовательностям геномов из таксона первичноротых: Araneae (выбрала эту группу, так как задание подразумевает поиск гомолога гена генетически далеких организмов; Сизый голубь – вторичноротое, далек от первичноротых).
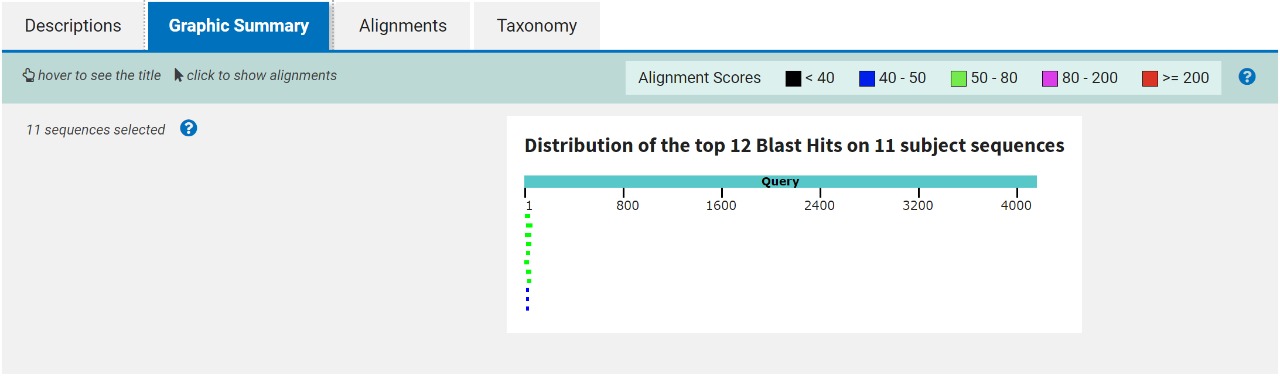
Выбрала алгоритм blastn (длина слова = 7, остальные параметры по умолчанию), так как он подходит для поиска гомологичных последовательностей и сравнения геномов не близкородственных организмов. База данных, по которой осуществлялся поиск: RefSeq Genome Database. Всего в refsec_genomes нашлось 4 сборки из таксона Araneae.Выдача содержала 11 находок из четырех разных организмов. Если мы откроем графическое представление выравнивания, то можно сразу увидеть, что выровнялись очень малые участки (скорее всего они выровнялись не на сам ген, а на его окрестность), что говорит о том, что δ-субъединица АТФ-синтазы у Columba livia и организмов таксона Araneae имеет существенные отличия.

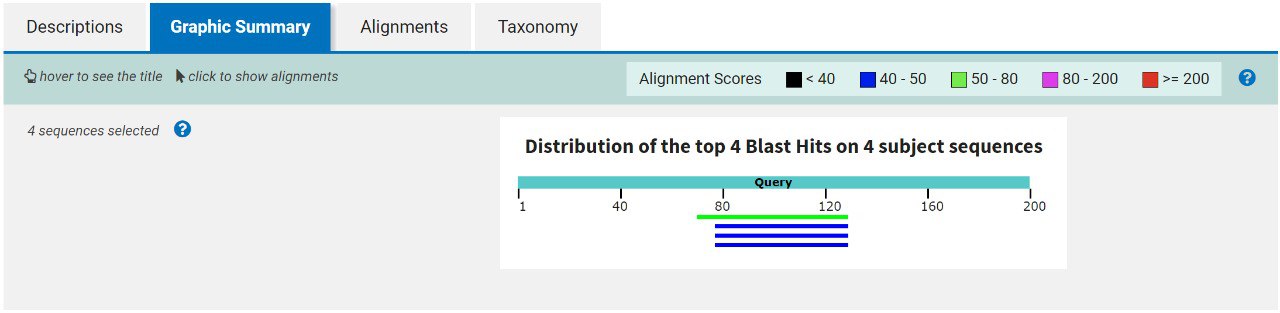
Далее выбрала алгоритм tblastn (длина слова = 5, остальные параметры также по умолчанию), с помощью которого искала белковые гомологи по выбраному таксону. Выдача содержала 4 находки из разных организмов. Из графического представления выравнивания видим, что процентр покрытия последовательности белка из Сизого голубя довольно низкий (у одной записи 29%, у остальный 26% покрытия). По этим данным также можно сделать вывод, что последовательность белка δ-субъединицы АТФ-синтазы из организмов таксона Araneae не имеет сильного сходства с последовательностью этого же белка из организма Columba livia.
Команда для индексации генома Columba livia:
makeblastdb -in GCF_036013475.1_bColLiv1.pat.W.v2_genomic.fna -dbtype nucl
16S рРНК – это РНК-компонент 30S субъединицы прокариотической рибосомы. Функции 16S рРНК: 3'-конец 16S рРНК связывается с белками S1 и S21, которые участвуют в инициации синтеза белка, взаимодействие с 23S, способствуя связыванию двух рибосомных субъединиц (50S и 30S), поддержание структуры малой субъединицы рибосомы.
23S рРНК – это РНК-компонент 50S субъединицы прокариотической рибосомы. Функции 23S рРНК: играет важную роль в связывании тРНК в P-участке большой рибосомной субъединицы, поддержание структуры большой субъединицы рибосомы.
Для поиска гомологов 16S и 23S рРНК я выбрала программу blastn, так как нужно искать среди неблизкородственных организмов гомологи по некодирующим белок последовательностям. Для последовательности 16S рРНК выдача содержала 6 находок (5 гомологов), для последовательности 23S рРНК – 37 (10 гомологов, если отберем по высокому e-value (можно было отсечь по этому критерию находки еще при формировании запроса на выравнивание) и посмотрим внимательно на то, что в некотрых случаях нашлись разные гомологи на одну нуклеотидную запись (смотрим координаты и понимаем: можем объединить выравнивания в нахождение одного гомолога или нет)).
Использованные команды:blastn -task blastn -query 16S.fasta -db GCF_036013475.1_bColLiv1.pat.W.v2_genomic.fna -outfmt 7 -out 16Stbl.out
blastn -task blastn -query 23S.fasta -db GCF_036013475.1_bColLiv1.pat.W.v2_genomic.fna -outfmt 7 -out 23Stbl.out
Для 16S рРНК довольно ожидаемо нашелся гомолог 18S рРНК, также из аннотированных вышли странные находки, но их e-value был очень высок, поэтому не взяла их во внимание (например, транскрипт erythrocyte membrane protein band 4.1 like 4A).
Для 23S рРНК довольно ожидаемо нашелся гомолог 28S рРНК, также из аннотированных вышли странные находки, но их e-value был очень высок, поэтому не взяла их во внимание (например, транскрипт xylosyltransferase 1 isoform X1).