
В практиуме мне выдан список из 36ти ID генов человека, связанных с метаболизмом жирных кислот, удлинением цепей и десатурацией. Нужно его проанализировать с помощью баз данных/сервисов.
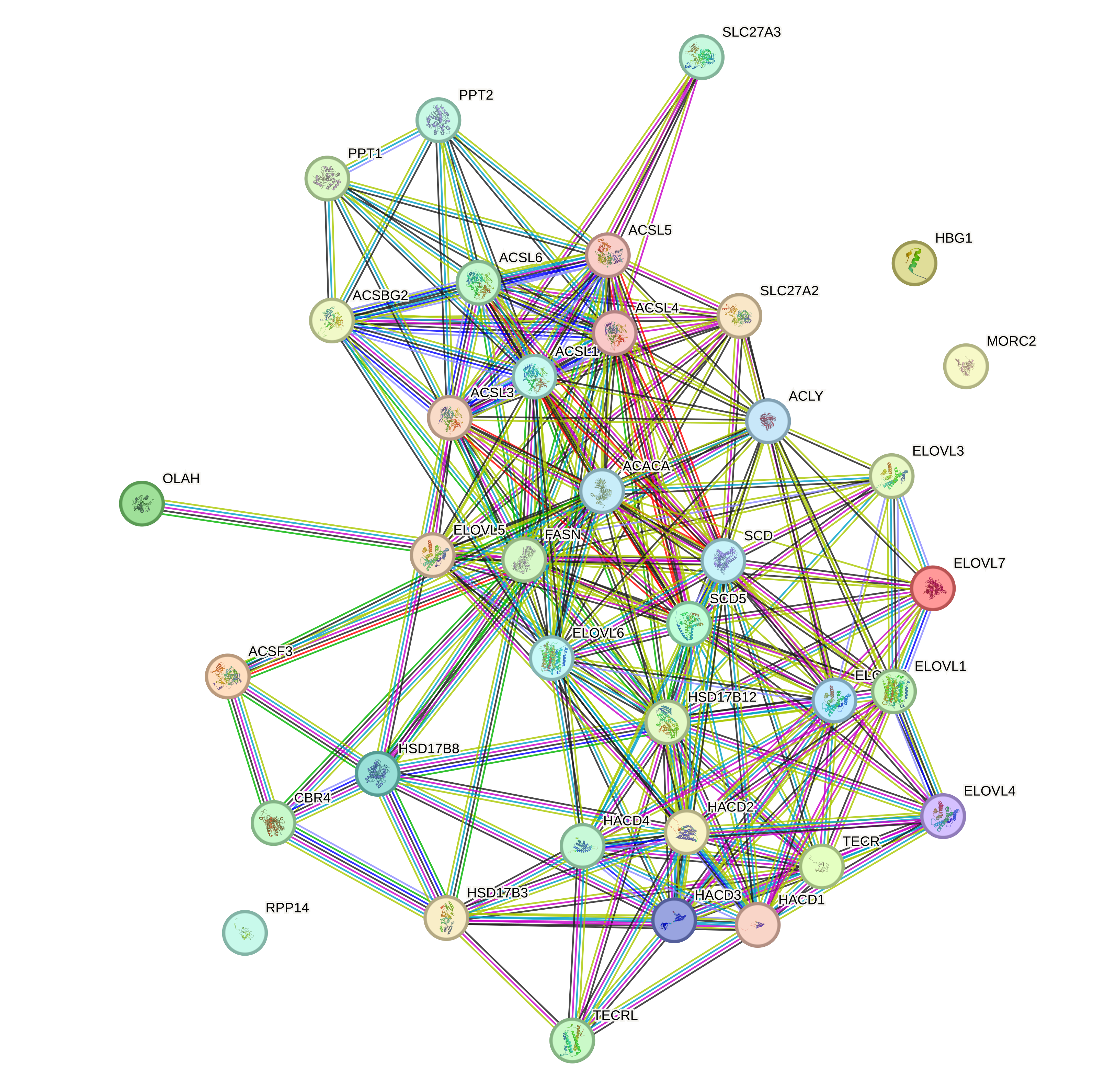
STRING – база данных для предсказывания функциональных связей между белками и визуализации кластеров. Загрузила в нее свой список белков и получила PPI сеть (Protein-Protein Interaction Network)(рис.1).
Видим, что три белка остались без взаимодействия с другими: RPP14 (субъединица рибонуклеазы P), HBG1 (субъединица гемоглобина), MORC2 (регулирует конденсацию гетерохроматина в ответ на повреждение ДНК и играет роль в подавлении транскрипции). То есть белки не участвуют в метаболизме ЖК, поэтому логично, что они не связаны с остальными белками.

|

|
| Рис. 1. PPI сеть |
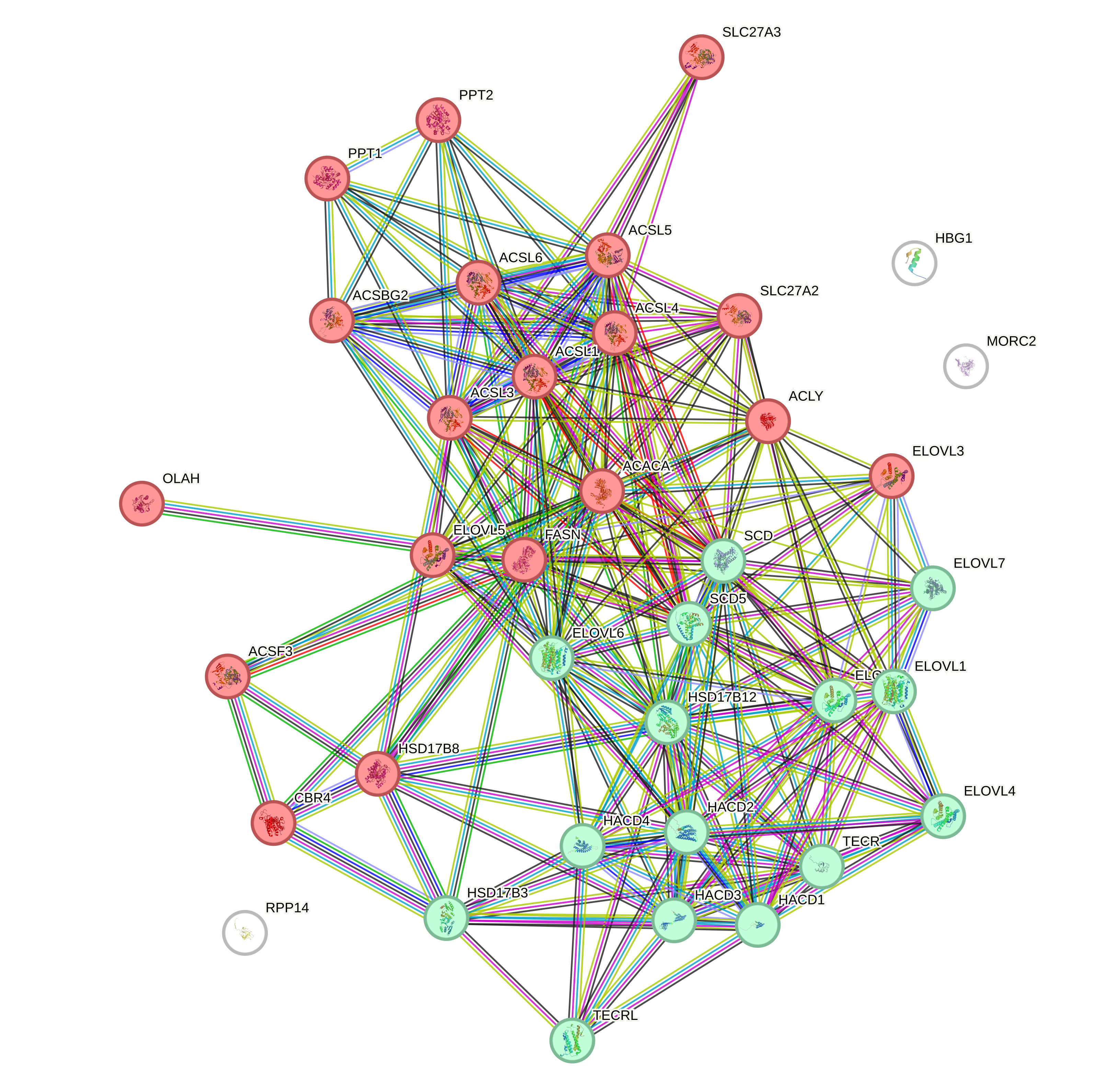
Далее я провела кластеризацию белков с помощью алгоритма MCL (Markov Cluster Algorithm) (рис. 2, рис. 3). В первый кластер вошли исключительно гены белков, участвующих в биосинтезе ацил-КоА жирных кислот. Во второй кластер также попали некоторые гены, связанные с биосинтезом ацил-КоА жирных кислот, но большинство из них кодируют белки, участвующие в биосинтезе длинноцепочечных и ненасыщенных жирных кислот. Это подчиняется биохимической логике разделения и взаимодействия этапов активации и модификации ЖК.

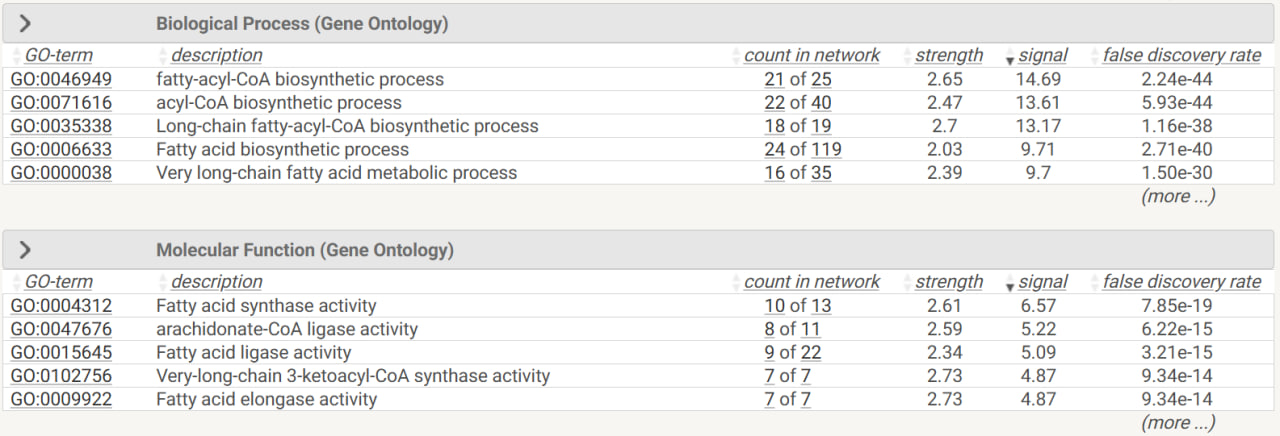
Далее посмотрела на таблицу биологических процессов и молекулярных функций белков, кодирующихся анализируемыми генами (рис. 4).
Наиболее значимые процессы: биосинтез эфира жирной кислоты с ацил-КоА (GO:0046949, GO:0071616) (задействовано >80% генов (21 из 25), 25 - общее число генов в геноме, связанных с этим термином), синтез длинноцепочечных ЖК (GO:0035338) (18 из 19 генов).
Далее решила провести в базе данных GO анализ на обогащение по биологической функции набора генов (рис.5). Получена таблица (отсортирована по p-value). Параметры запроса: точный тест Фишера для подсчёта p-value и поправка Бонферрони на множественное тестирование.
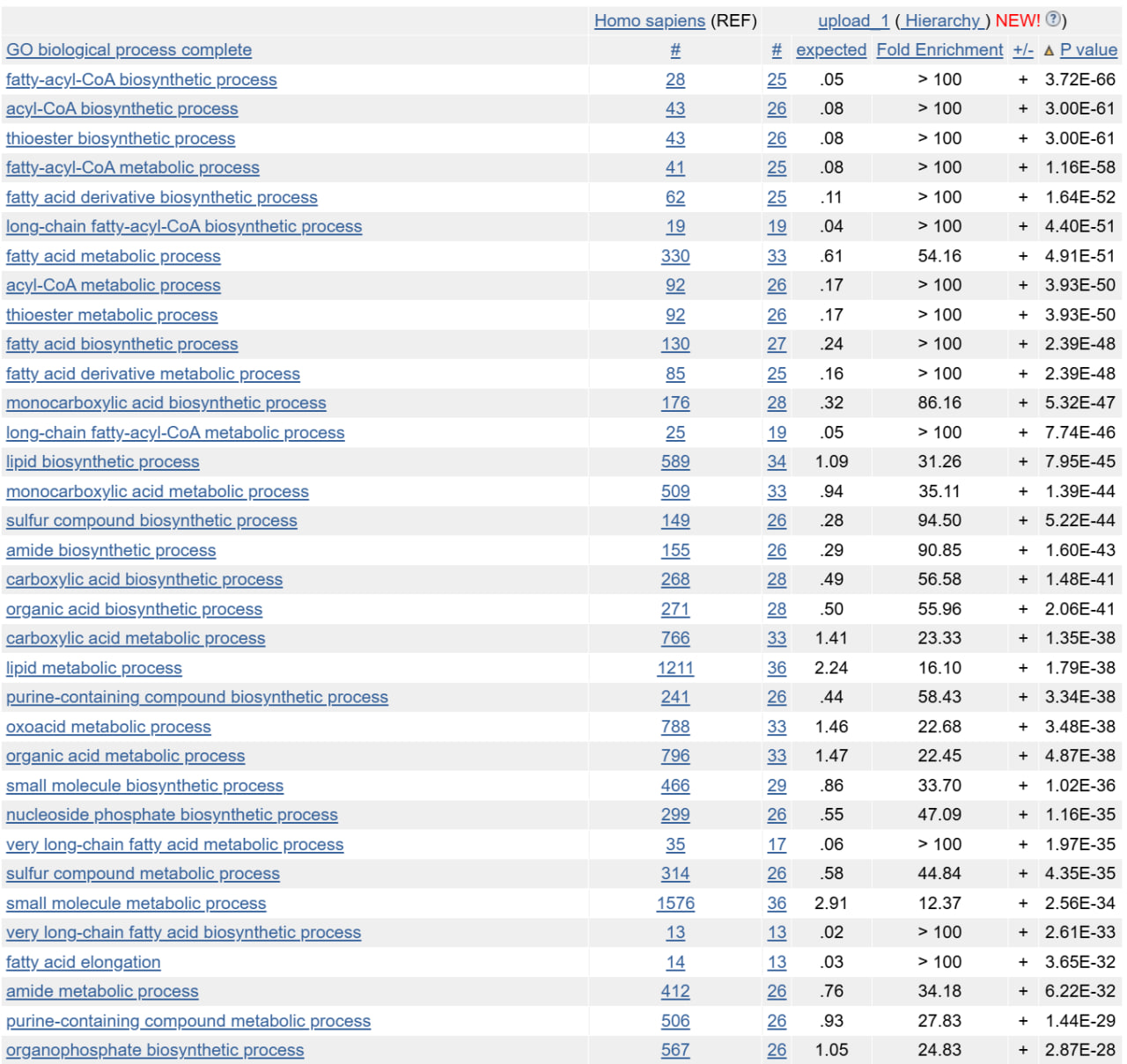
Всего было найдено 63 GO terms. Из таблицы мы видим, что основные биологические процессы в которых участвуют белки это: биосинтез эфира жирной кислоты с ацил-КоА и его метаболизм, синтез ацил-КоА, удлинение жирных кислот (насыщенных и ненасыщенных), биосинтез очень длинноцепочечных ЖК, биосинтез длинноцепочечных ЖК, биосинтез ненасыщенных ЖК, метаболизм линолевой кислоты, синтез сфинголипидов.
Human Protein Atlas – база данных, которая предоставляет информацию о протеоме человека с детализацией на уровне тканей, клеток и субклеточных структур.
Для анализа выбрала ген ELOVL1 (ген: ELOVL1 = Elongation of very long chain fatty acids protein 1, белок: ELOVL элонгаза 1 жирных кислот). Во вкладке summary видим общую информацию о гене и транскриптах (рис. 6). У выбранного гена известно 3 транскрипта.
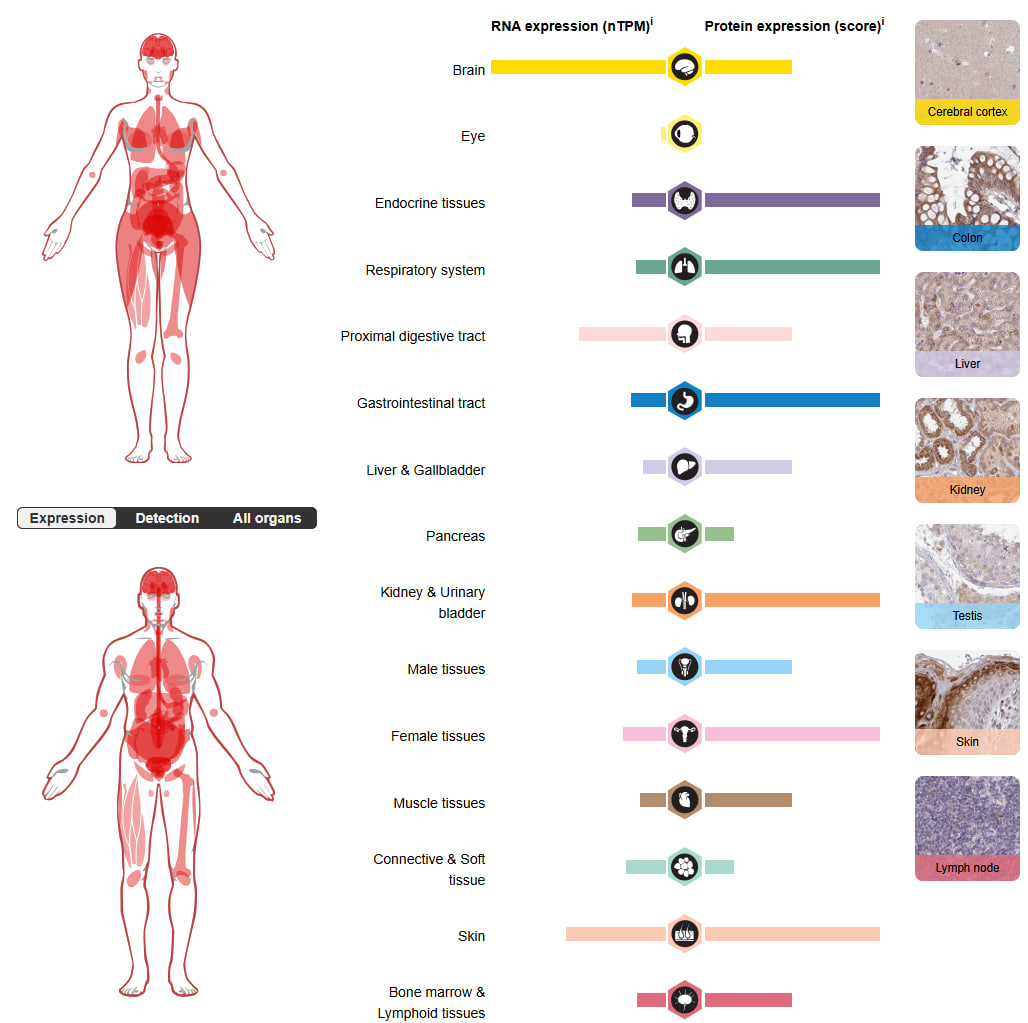
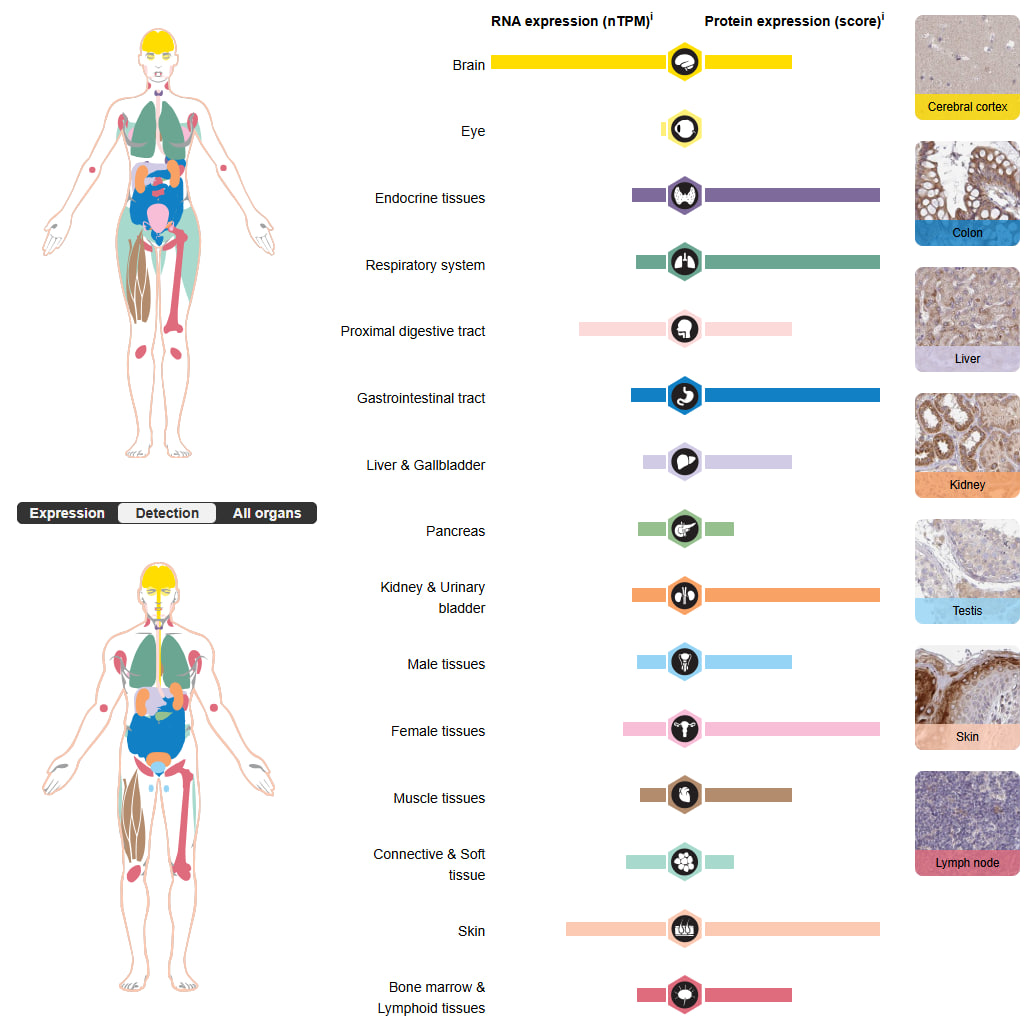
Далее посмотрела визуализацию локализации экспрессии и обнаружения транскриптов гена (рис. 7, рис. 8. Экспрессии гена не наблюдается в глазах, также низкий уровень экспрессии наблюдается в поджелудочной железе. Высокий уровень экспрессии в мозге, эндокринной ткани, дыхательной системе, ЖКТ, почках, мочевом пузыре, женской половой системе, коже. Высокий уровень экспрессии РНК наблюдается в мозге. Высокий уровень экспрессии ELOVL1 в мозге согласуется с известной ролью очень длинноцепочечных жирных кислот в формировании миелиновых оболочек нейронов.
|
|
| Рис. 7. Визуализация локализации экспрессии | Рис. 8. Визуализация обнаружения транскриптов гена |
Также посмотрела локализацию белка в клетке (рис.9). ELOVL1 локализуется на эндоплазматическом ретикулуме, что ожидаемо, так как именно в ЭПР идет элонгация цепочки ЖК.
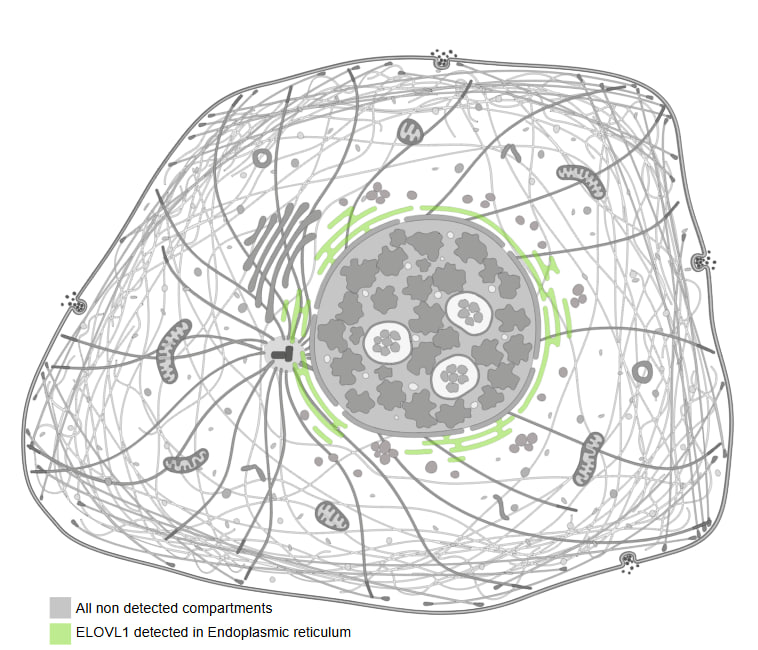
Итог: я проанализировала данный мне набор генов в трех базах данных. В STRING увидела их взаимодействия, а также выяснила, что есть 3 гена, которые не объединяются с остальными. В GO провела анализ на обогащение по биологической функции набора генов (данные практически совпали с тем, что было дано в STRING) и выяснила, что все они связаны с метаболизмом жирных кислот. В Human Protein Atlas проанализировала экспрессию белка ELOVL1 в тканях и клетке.