Составление списка гомологов
Выбраны бактерии:
- ACICJ - Acidiphilium cryptum
- BARHE - Bartonella henselae
- HAEIN - Haemophilus influenzae
- NEIMA - Neisseria meningitidis
- PASMU - Pasteurella multocida
- ROSDO - Roseobacter denitrificans
- SHEDO - Shewanella denitrificans
- THIDA - Thiobacillus denitrificans
Достоверные гомологи белка CLPX_ECOLI
Объединил протеомы всех бактерий в один файл с помощью команды:
Произвел поиск программой blastp:
Полная выдача доступна по ссылке. Был получен список из 19 находок:
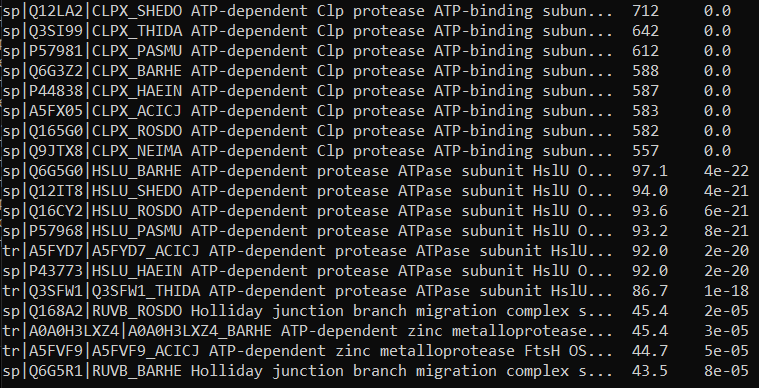
Реконструкция и визуализация
Отредактированный файл с найденными последовательностями доступен по ссылке
Выравнивание производил с помощью команды muscle:
Произвел построение дерева (файл с посл-тями был предварительно переведен в формат phylip-relaxed скриптом по ссылке) с помощью программы fastme, используя модель p-distance:
Полученная Newick формула доступна по ссылке
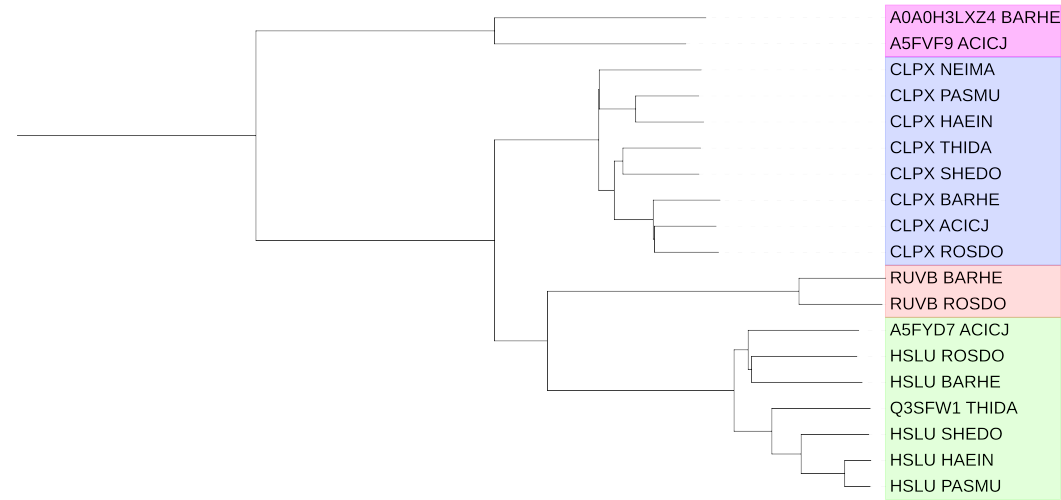
Примеры пар ортологов (рис. 1):
- CLPX_PASMU и CLPX_HAEIN
- HSLU_PASMU и HSLU_HAEIN
- RUVB_BARHE и RUVB_ROSDO
Примеры пар паралогов (рис. 1):
- HSLU_SHEDO и CLPX_SHEDO
- A5FVF9_ACICJ и CLPX_ACICJ
- RUVB_ROSDO и HSLU_ROSDO
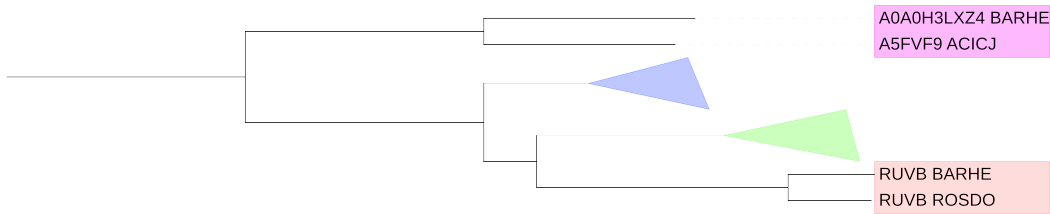
Сравнение с референсом
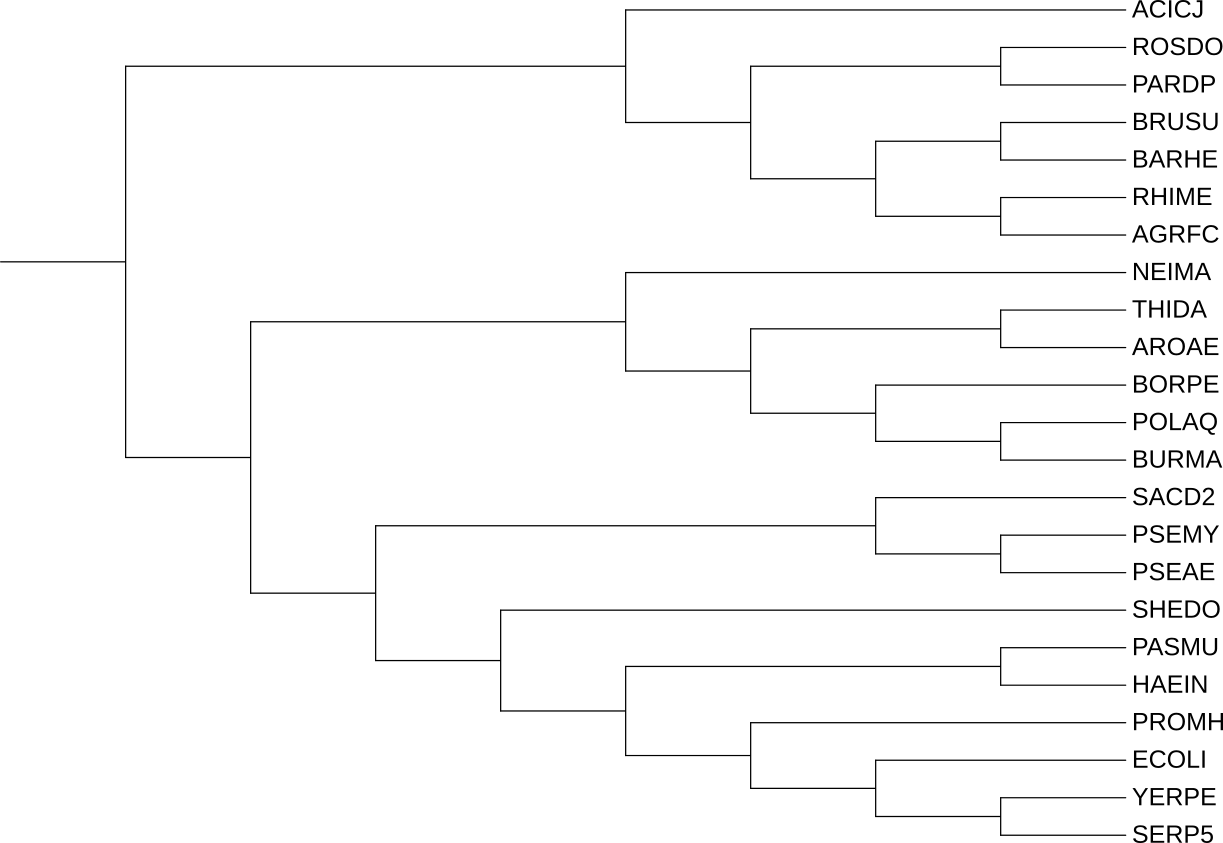
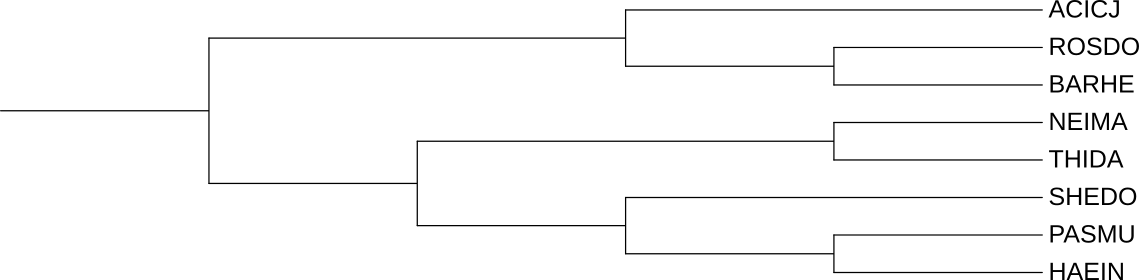
Дерево на рис. 3 является референсным. Пользоваться им для сравнения результатов не очень удобно (на дереве расположено 23 бактерии, а нас интересует только треть из них), поэтому я построил новое дерево (рис. 4) из 8 моих бактерий на основе старого дерева.
При сравнении полученного дерева гомологов (рис. 5) с новым референсным (рис. 4) можно легко заметить, что ветвь с HSLU белками (выделена зеленым) построена правильно. От референса данная ветвь отличается лишь отсутствием бактерии NEIMA. Ветвь достоверных ортологов CLPX окрашена красным, т. к. её устройство не совпадает с референсом. Правильно выделены в подгруппу только CLPX_PASMU и CLPX_HAEIN. Маленькие ветви не стоит принимать во внимание - они, конечно же, обладая всего двумя листами (каждая), построиться могли лишь одним способом.
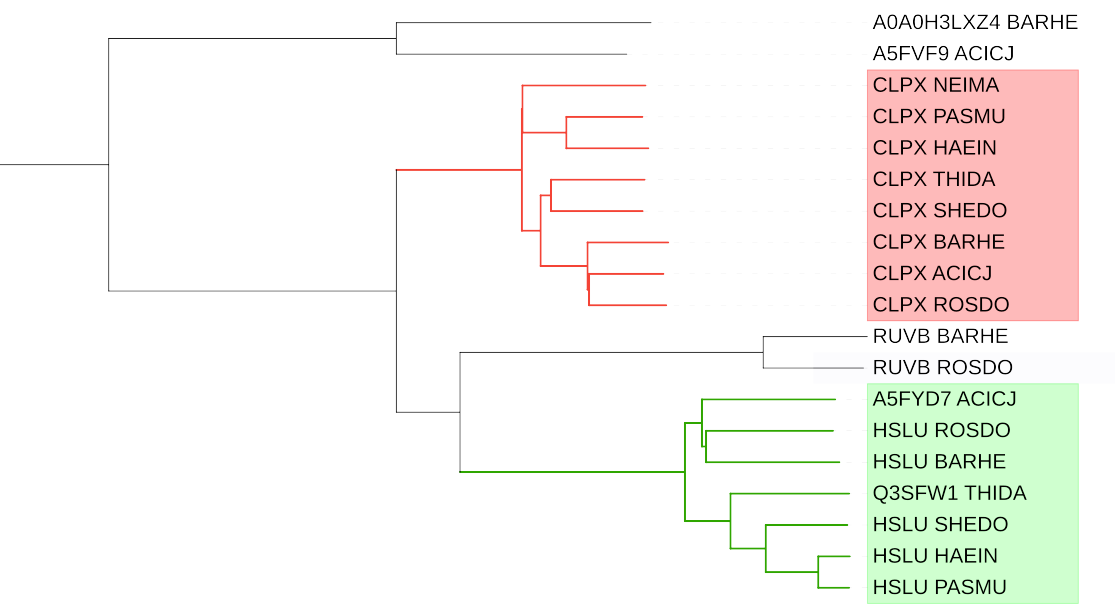
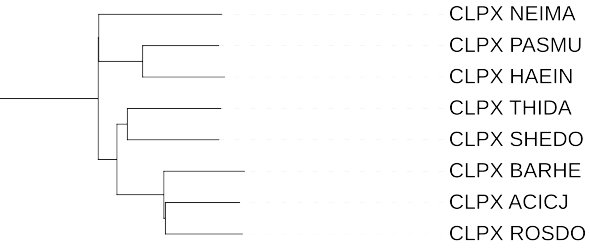
Подробнее рассмотрим неправильную ветвь:
- Правильно объединены PASMU и HAEIN
- CLPX_BARHE, CLPX_ACICJ и CLPX_ROSDO, судя по дереву, крайне незначительно отличаются друг от друга. Но iTOL не делает трихотомий, и поэтому программа предпочла сделать ошибку.
- Ошибочно объединились в одну кладу SHEDO и BARHE.
- NEIMA вместе с кладой PASMU-HAEIN объединены в одну кладу. В этом месте iTOL тоже мог бы поставить трихотомию (с другими пятью бактериями) - отличия между белками не очень велики.
Выводы
С помощью программы blastp удалось найти гомологов и относительно удачно расположить их на дереве. Возникли проблемы с расположением белков CLPX, зато группа бактерий идеально распределилась по HLSU и пары других белков, которые, видимо, не были аннотированы и не имеют мнемоники. Можно убедиться с помощью базы данных UniProt в том, что данные белки также являются продуктами гена hslU (в соответствующих организмах) и выполняют роль АТФ-зависимой АТФазной субъединицы (как и их аннотированные в Swiss-Prot гомологи).