 BLAST
BLAST BLAST
BLAST|
Решалась задача: поиск гомологов белка тимидилат синтазы (thymidylate synthase) из организма
Deinococcus maricopensis. Поиск вёлся по базе данных Refseq_protein, диапазон поиска - 5000 результатов, максимальное E-value по умолчанию - 10, хотя вероятность гомологии таких белков очень мала. Было найдено 2387 находок. Они распределялись по организмам следующим образом:
Можно заметить, что процент идентичных остатков в "худшей" находке превышает данный параметр для находки из середины. Это объясняется тем, что в последней находки очень маленький участок последовательности имеет сходство с запросом, но относительно длины этого участка в нём содержится много идентичных остатков.
Сравнение находок по всему банку белков и по бактериямЯ добавила в параметры поиска - поиск только по бактериальным белкам (с порогом E-value < 0,001). Было найдено 1632 белка. Я взяла находку, которая встречалась при прошлом запуске blast. Score для неё не изменился, как и процент идентичных колонок. А вот E-value уменьшился с 5e-134 до 4e-134. Но это та же находка, что и при предыдущем запуске, т.к. она имеет такой же код доступа (Accession number). E-value оценивает не похожесть находки на заданный белок, а количество находок, имеющих больший score. А раз я уменьшила диапазон поиска и общее количество находок стало меньше, то и E-value уменьшился. При этом остальные параметры не изменились, поскольку выравнивание данной последовательности относительно заданного белка не изменилось.Для этой же находки (она принадлежит организму Deinococcus phoenicis) я построила карту локального сходства (Рис.1). Она указывает на сходство участков 5-231 искомого белка и 22-249 выбранной находки. 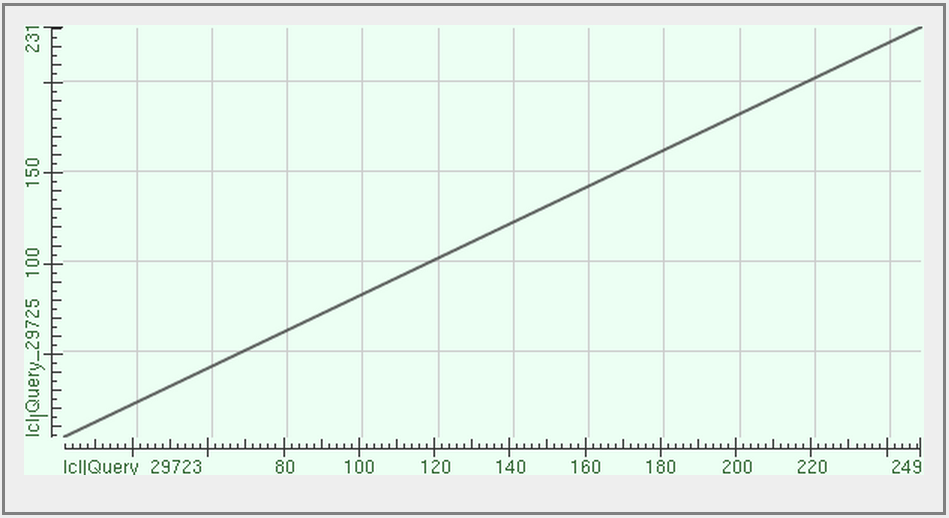
Поиск по своей базе данныхЯ создала базу данных из выравнивания, которое использовала для освоения программы Jalview. Был проведён поиск по моей базе данных белка тимидилат синтазы (thymidylate synthase). В результате похожие мотивы были найдены в двух последовательностях.
|
«Назад Дальше» |
|||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
© Колупаева А.Л. 2014 |