 Чтение последовательностей по Сэнгеру
Чтение последовательностей по Сэнгеру Чтение последовательностей по Сэнгеру
Чтение последовательностей по Сэнгеру
По результатам хроматограмм из капиллярного секвенатора по Сэнгеру и последовательности, сгенерированной программой, получить последовательность фрагмента ДНК
»Выравнивание последовательностей »Цепь ДНК В программе Chromas (Lite) были выровняны относительно друг друга хроматограммы прямой и обратной цепей ДНК. С помощью команды reverse + complement была получена реверсированная последовательность, комплементарная обратной. Команда find отмечала одинаковые участки в двух последовательностях, с ориентировкой на которые затем строилось выравнивание в программе Jalview. После того, как основная часть двух последовательностей выравнена, необходимо обратить внимание на участки несоответствия. Так на участке 109 - 115 (отсчёт оснований по выравненной цепи ДНК) наблюдается пятно краски на хроматограмме прямой цепи (Рис.1). На обратной же цепи этот участок сомнений не вызывает. На участке 150 - 151 на прямой цепи помехи мешают различить сигналы от оснований, а на обратной помех нет. 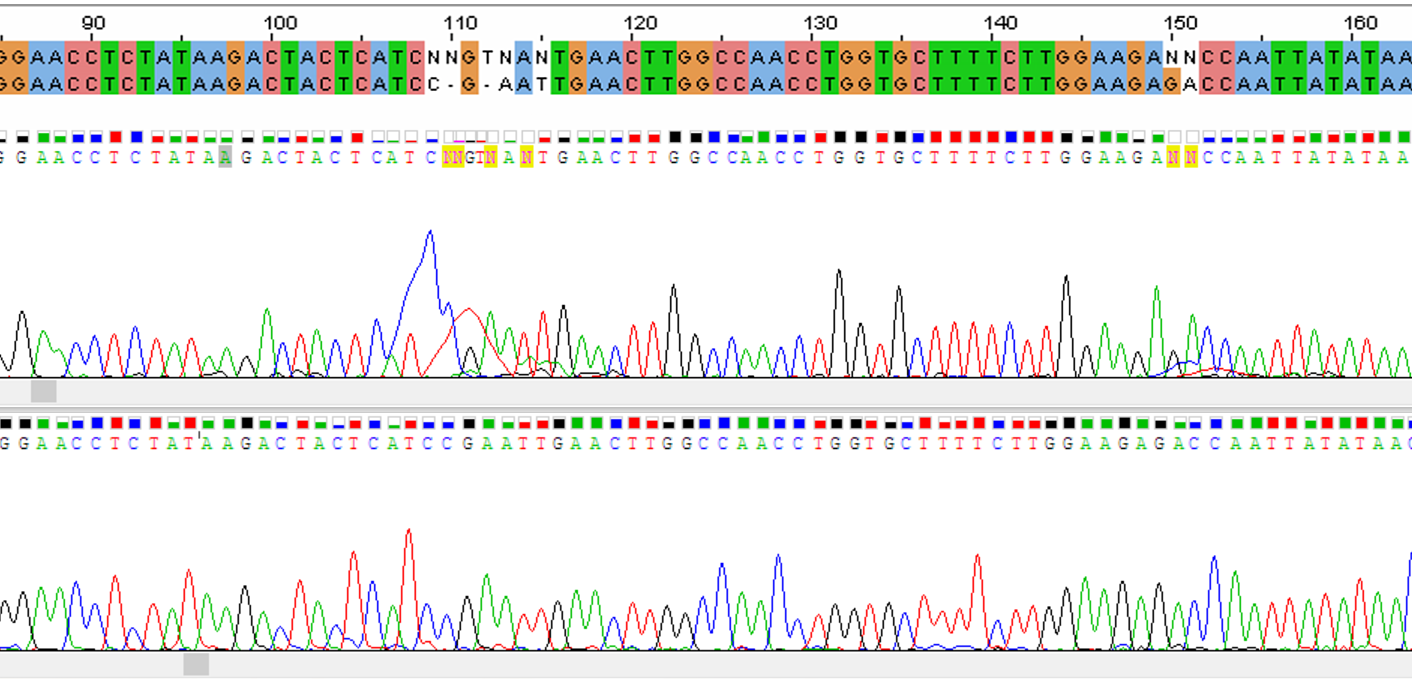
Также в последовательностях было несколько мест, где программа поставила "N" (т.е. неопределённый нукеотид) над некоторым нуклеотидом, а все его соседи определены. Это связано с тем, что сигнал от нуклеотида хоть и враже, но не сильно превосходит уровень шума, который по какой-то причине проявился на месте конкретного нуклеотида и отличается от общего уровня шума (Рис.2, 3, 4).
Такое изображение может свидетельстввать о существовании полиморфизма для данных нуклеотидов. Хотя в случаях на рисунках 2 и 3 такое маловероятно, поскольку рядом есть пики шума сравнимой высоты. На рисунке 4 также наблюдаются дргие сравнительно большие шумы рядом с позицией "N", но здесь помимо этого есть довольно большой пик гуанина, перекрывающийся с тимином 381, по высоте сравнимый с другими пиками гуанина.Можно предположить, что в позиции 381 сссуществует полиморфизм, выражающийся в замене тимина на гуанин. Начало прямой цепи (Рис.5) и конец обратной (Рис.6) явяются нечитаемыми, поскольку секвенировались в начале. Эти концы необходимо удалить, так как никакой информации они не несут. Координаты нечитаемых концов (по прямой последовательности): Начало: 0 - 16 Конец: 653 - 702 Хотя программа уже не определяет некоторые основания до 653 на обратной последовательности, но визуально их можно различить, и они совпадают с таковыми на прямой.
В целом, хроматограмма имеет довольно высокое качество, поскольку сила шума была не сравнима с силой сигнала, пики сигнала разделены и хорошо выражены. Однако было одно пятно краски на прямой цепи и сложно определяемый участок близко к концу обратной цепи. Рассмотрим нечитаемый участок и выясним, почему нельз извлечь из него информацию. 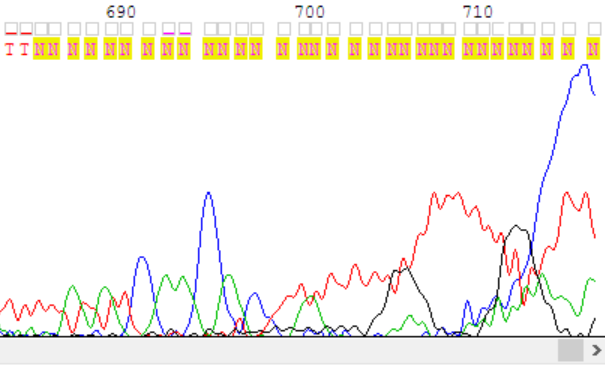
По цвету можно определить, сигнал какого нуклеотида наблюдается на хроматограмме. Но выяснить, в какой последовательности они идут не представляется возможным, поскольку одни пики леат поверх других.Также нельзя разобрать, сигнал скольки тиминов изображён на участках 683 - 690 и 698 - 716, так как пики не разделены. К тому же, невозможно разобрать, какой сигнал яляется шумовым, а какой истинным, ведь все сигналы одного уровня. Всё, что можно заключить из этой хроматограммы, это то, что в этом участке ДНК есть аденин, тимин, цитозин и гуанин, но для анализа последовательности эта информация не имеет практического смысла. |
«Назад Дальше» |
|||||||||||||||||
|
© Колупаева А.Л. 2014 |