Гомологичные белки и выравнивания
Сравнение выравниваний программами Mafft, T-Coffee и Muscle
В данном задании было проведено сравнение множественных выравниваний белков с мнемоникой EFEB из практикума 9, выполненных разными программами.
Сравнение осуществлялось по идентичным блокам в разных выравниваниях. Полученные выравнивания можно посмотреть в проекте Jalview.
Попарная визуализация идентичных блоков доступна по ссылкам (программа VerAlign) :
Muscle+Mafft,
Mafft+T-Coffee
Далее представлено описание идентичных блоков в выравниваниях :
| Блок | Mafft+Muscle | Mafft+T-Coffee |
|---|---|---|
| 1 | (12-31)=(12-31) | (1-2)=(1-2) |
| 2 | (65-438)=(62-435) | (13-31)=(13-31) |
| 3 | - | (36-39)=(36-39) |
| 4 | - | (65-124)=(64-123) |
| 5 | - | (127-438)=(126-437) |
Процент выровненных колонок при использовании Mafft : 96.58%, длина выравнивания = 438
Процент выровненных колонок при использовании Muscle : 97.24%, длина выравнивания = 435
Процент выровненных колонок при использовании T-Coffee : 96.80%, длина выравнивания = 437
Для поиска идентичных блоков была использована программа за авторством Ксении Кирцовой.
Выравнивание по совмещению структур и сравнение его с выравниванием программой Mafft
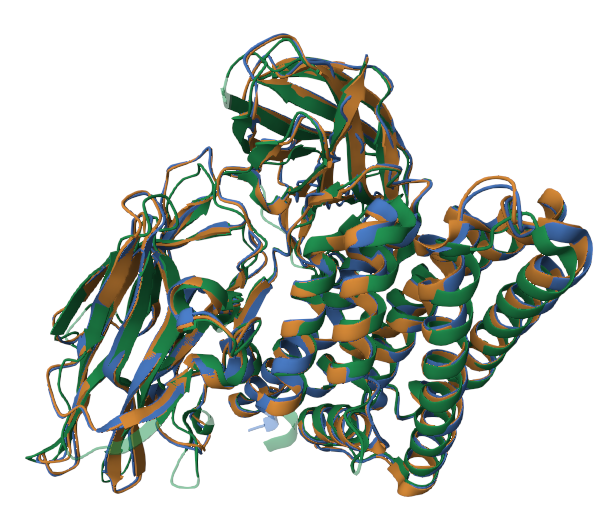
Для проведения выравнивания путем совмещения пространственных структур было выбрано семейство PF00555 Endotoxin_M, а в нем белки с pdb-кодами 6lfp, 1dlc, 3eb7.
Выравнивание было выполнено на сайте PDB (Analyze -> Pairwise Structure Alignment), в качестве референсного белка был выбран 6lfp
В Jalview было произведено выравнивание этих же белков с помощью Mafft. При сравнении вышеупомянутых выравниваний с помощью программы из задания 2
был выявлен один идентичный блок (469-470) PDB (длина выравнивания - 600, процент выровненных колонок - 98.17%) и (485-486) Mafft (длина выравнивания
- 615, процент выровненных колонок - 95.77%)
Наиболее серьезное видимое отличие в совмещенных структурах - отсутсвие небольшого участка выравнивания с Nконца у белка 6lfb.
Полученные выравнивания можно посмотреть в проекте Jalview

Описание программы Mafft
Mafft - программа, используемая для создания множественных выравниваний аминокислотных или нуклеотидных последовательностей. Первая версия была создана Казутакой Като в 2002 году, последняя версия вышла в 2022 году.
Алгоритм :
Прогрессивная часть : составить приблизительную матрицу расстояний, предварительно составив парные выравнивания, построить направляющее дерево и
выровнять последовательности в соответствии с порядком ветвления.
Итеративная часть : Построить множественное выравнивание -> Разделить его на две группы -> Перевыровнять две группы. Повторить.
Точность прогрессивного выравнивания повышается с помощью метода итеративного уточнения. Итеративное уточнение повторяется
до тех пор, пока не перестанет улучшаться показатель WSP (сумма весов пар) или количество циклов не достигнет 1000.
Параметры : матрица замен аминокислот BLOSUM62, матрица нуклеотидов, штрафы за гэпы в белках, штрафы за гэпы в РНК