Филогенетическое дерево
Отобранные организмы, их таксономия и мнемоники
| Вид | Таксономия | Мнемоника |
|---|---|---|
| Trichosurus caninus | Eumetazoa; Bilateria; Deuterostomia; Chordata; Craniata; Vertebrata; Gnathostomata; Teleostomi; Euteleostomi; Sarcopterygii; Dipnotetrapodomorpha; Tetrapoda; Amniota; Mammalia; Theria; Metatheria; Diprotodontia; Phalangeridae; Trichosurus | TRICS |
| Lepus othus | Eumetazoa; Bilateria; Deuterostomia; Chordata; Craniata; Vertebrata; Gnathostomata; Teleostomi; Euteleostomi; Sarcopterygii; Dipnotetrapodomorpha; Tetrapoda; Amniota; Mammalia; Theria; Eutheria; Boreoeutheria; Euarchontoglires; Glires; Lagomorpha; Leporidae; Lepus | LEPOT |
| Lepus brachyurus | Eumetazoa; Bilateria; Deuterostomia; Chordata; Craniata; Vertebrata; Gnathostomata; Teleostomi; Euteleostomi; Sarcopterygii; Dipnotetrapodomorpha; Tetrapoda; Amniota; Mammalia; Theria; Eutheria; Boreoeutheria; Euarchontoglires; Glires; Lagomorpha; Leporidae; Lepus | LEBR |
| Rattus norvegicus | Eumetazoa; Bilateria; Deuterostomia; Chordata; Craniata; Vertebrata; Gnathostomata; Teleostomi; Euteleostomi; Sarcopterygii; Dipnotetrapodomorpha; Tetrapoda; Amniota; Mammalia; Theria; Eutheria; Boreoeutheria; Euarchontoglires; Glires; Rodentia; Myomorpha; Muroidea; Muridae; Murinae; Rattus | RAT |
| Mus poschiavinus | Eumetazoa; Bilateria; Deuterostomia; Chordata; Craniata; Vertebrata; Gnathostomata; Teleostomi; Euteleostomi; Sarcopterygii; Dipnotetrapodomorpha; Tetrapoda; Amniota; Mammalia; Theria; Eutheria; Boreoeutheria; Euarchontoglires; Glires; Rodentia; Myomorpha; Muroidea; Muridae; Murinae; Mus; Mus | MUSPO |
| Mus musculus | Eumetazoa; Bilateria; Deuterostomia; Chordata; Craniata; Vertebrata; Gnathostomata; Teleostomi; Euteleostomi; Sarcopterygii; Dipnotetrapodomorpha; Tetrapoda; Amniota; Mammalia; Theria; Eutheria; Boreoeutheria; Euarchontoglires; Glires; Rodentia; Myomorpha; Muroidea; Muridae; Murinae; Mus; Mus | MOUSE |
| Sus philippensis | Eumetazoa; Bilateria; Deuterostomia; Chordata; Craniata; Vertebrata; Gnathostomata; Teleostomi; Euteleostomi; Sarcopterygii; Dipnotetrapodomorpha; Tetrapoda; Amniota; Mammalia; Theria; Eutheria; Boreoeutheria; Laurasiatheria; Artiodactyla; Suina; Suidae; Sus | SUSPH |
| Sus scrofa | Eumetazoa; Bilateria; Deuterostomia; Chordata; Craniata; Vertebrata; Gnathostomata; Teleostomi; Euteleostomi; Sarcopterygii; Dipnotetrapodomorpha; Tetrapoda; Amniota; Mammalia; Theria; Eutheria; Boreoeutheria; Laurasiatheria; Artiodactyla; Suina; Suidae; Sus | PIG |
| Ailuropoda melanoleuca | Eumetazoa; Bilateria; Deuterostomia; Chordata; Craniata; Vertebrata; Gnathostomata; Teleostomi; Euteleostomi; Sarcopterygii; Dipnotetrapodomorpha; Tetrapoda; Amniota; Mammalia; Theria; Eutheria; Boreoeutheria; Laurasiatheria; Carnivora; Caniformia; Ursidae; Ailuropoda | AILME |
| Ursus thibetanus | Eumetazoa; Bilateria; Deuterostomia; Chordata; Craniata; Vertebrata; Gnathostomata; Teleostomi; Euteleostomi; Sarcopterygii; Dipnotetrapodomorpha; Tetrapoda; Amniota; Mammalia; Theria; Eutheria; Boreoeutheria; Laurasiatheria; Carnivora; Caniformia; Ursidae; Ursus | URSTH |
| Ursus arctos | Eumetazoa; Bilateria; Deuterostomia; Chordata; Craniata; Vertebrata; Gnathostomata; Teleostomi; Euteleostomi; Sarcopterygii; Dipnotetrapodomorpha; Tetrapoda; Amniota; Mammalia; Theria; Eutheria; Boreoeutheria; Laurasiatheria; Carnivora; Caniformia; Ursidae; Ursus | URSAR |
Скобочная формула дерева
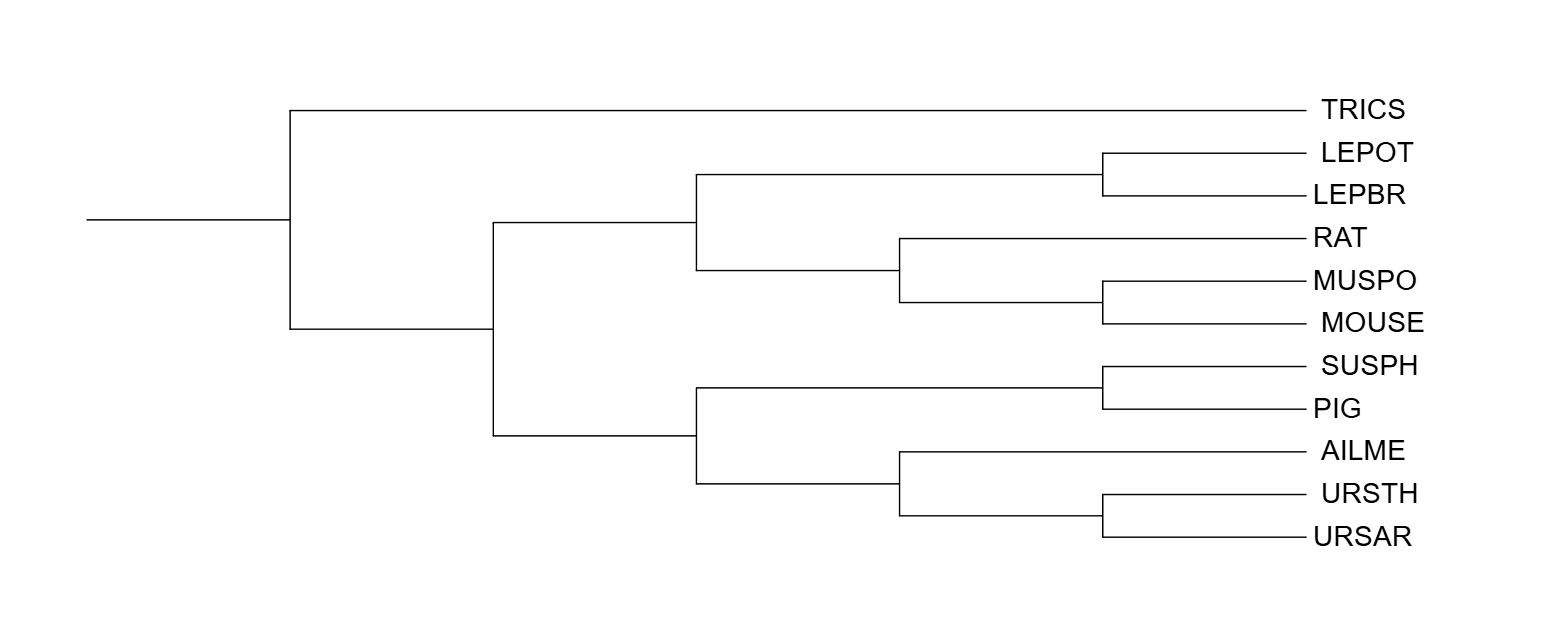
((((LEPBR, LEPOT), (RAT, (MUSPO, MOUSE))), ((PIG, SUSPH), ((URSAR, URSTH), AILME))), TRICS);
Изображение дерева
Таксономия
Реконструкция с помощью программ
Норма
LEPBR,LEPOT vs RAT, MUSPO, MOUSE
LEPBR,LEPOT, RAT, MUSPO, MOUSE vs SUSPH, PIG, AILME, URSAR, USTH
Fatsme p-distance

- У клад LEPBR,LEPOT и RAT, MUSPO, MOUSE должен быть общий предок, в данной реконструкции его нет - данная программа смоделировала общего предка LEPBR,LEPOT и SUSPH, PIG, AILME, URSAR, USTH
- Есть общий предок у клад LEPBR,LEPOT, SUSPH, PIG, AILME, URSAR, USTH и RAT, MUSPO, MOUSE (а долен быть у LEPBR,LEPOT, RAT, MUSPO, MOUSE и SUSPH, PIG, AILME, URSAR, USTH
Отличия от таксономического дерева кратко:
- LEPBR,LEPOT vs SUSPH, PIG, AILME, URSAR, USTH
- LEPBR,LEPOT, SUSPH, PIG, AILME, URSAR, USTH vs RAT, MUSPO, MOUSE
Fastme MtREV

Данное дерево реконструировано верно
IQ-Tree

- У клад LEPBR,LEPOT и RAT, MUSPO, MOUSE должен быть общий предок, в данной реконструкции его нет - зато есть общий предок у клад LEPBR,LEPOT и SUSPH, PIG, AILME, URSAR, USTH, RAT, MUSPO, MOUSE
- LEPBR,LEPOT, RAT, MUSPO, MOUSE и SUSPH, PIG, AILME, URSAR, USTH должны расходиться на разные ветви, но в данном случае разделяются RAT, MUSPO, MOUSE и SUSPH, PIG, AILME, URSAR, USTH, а веть с LEPBR,LEPOT отделяется раньше
- LEPBR,LEPOT vs SUSPH, PIG, AILME, URSAR, USTH, RAT, MUSPO, MOUSE
- RAT, MUSPO, MOUSE vs SUSPH, PIG, AILME, URSAR, USTH
Отличия от таксономического дерева кратко:
Реконструкция дерева по нуклеотидным последовательностям 12S rRNA

При реконструкции дерева по последовательностям 12S rRNA не был найден геном для организмов LEPBR, LEPOT и MUSPO.
Поэтому я взяла представителей того же рода, но другого вида - таких, чтобы у них был геном в базе данных ENA -
LEPEU, LEPYA и MUSMA.
Построеннное с помощью программы fastme дерево полностью соответсвует таксономическому. Соответственно, отличия от
деревьев, реконструированных по цитохрому В полностью аналогичны (учтем измененные виды).
Примерный код
Укоренение во внешнюю группу

Для укоренения дерево по внешней группе я выбрала TACAC (ехидна)- это животное относится к Первозверям, тогда как все остальные - Звери. Точка укоренения определилась верно - от внешней группы отделается таксономически правильное дерево.
Бутстреп - поддержка ветвей

Бутсреп нужен для оценки правдоподобности построенных ветвей - чем в большем числе реплик выделялась наша ветвь, тем выше у нее поддержка. Здесья использовала 100 реплик бутсрепа - т.е максимальное число поддержки = 100.
При использовании параметров по умолчанию я получила полностью верное дерево. Меня насторожило, что у верной ветви поддержка 51 - это значит, что данная ветвь скорее всего неверна. Поэтому я решила реконструировать дерево с помощью p-distance и посмотреть, что изменится.

P-distance выдала дерево с неверным разбиением на клады - у LEPEU, LEPYA и SUSPH, PIG, AILME, URSAR, USTH, RAT,
MUSMA, MOUSE неожиданно образовался общий предок, а у LEPEU, LEPYA и RAT, MUSMA, MOUSE общего предка нет.
Однако, у данной неверной ветви очень низкая поддержка - 49. А значит, она вероятнее всего реконструирована ошибочно.
Что и следовало доказать (если поддержка низкая, то к этой ветви нужно относиться с осторожность и недоверием...).
Отметим, что ветви с поддержкой 100 встречаются во всех реконструированных деревьях.