
Длина гена белка RIR2_ECOLI равна 1131 нуклеотида.
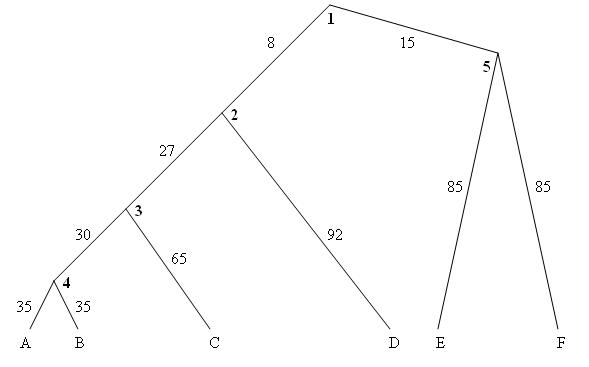
Был проведён анализ филогенетического дерева, результаты приведены в отчете.
Затем с помощью программы msbar были внесены мутации в белок RIR2_ECOLI, их количество было определено в соответствии с деревом с учётом длины гена.
Ниже представлен полный текст скрипта:
msbar RIR2_ECOLI.fasta 5_165.fasta -point 4 -count 165 -auto
msbar RIR2_ECOLI.fasta 2_88.fasta -point 4 -count 88 -auto
msbar 2_88.fasta 3_297.fasta -point 4 -count 297 -auto
msbar 2_88.fasta D_1012.fasta -point 4 -count 1012 -auto
msbar 5_165.fasta E_935.fasta -point 4 -count 935 -auto
msbar 5_165.fasta F_935.fasta -point 4 -count 935 -auto
msbar 3_297.fasta 4_330.fasta -point 4 -count 330 -auto
msbar 3_297.fasta C_715.fasta -point 4 -count 715 -auto
msbar 4_330.fasta A_385.fasta -point 4 -count 385 -auto
msbar 4_330.fasta B_385.fasta -point 4 -count 385 -auto
|
В начало страницы.
На главную страницу.