Построение профиля
Для построение профиля были выбраны последовательности, которые принадлежат к одной кладе на дереве из предыдущего практикума, а также принадлежат таксону Amniota и обладают весьма консервативной последовательностью. В качестве признака было выбрано наличие подпоследовательностей PRESV (5-9) и TQNSSQ (40-45). Команды для построения и калибровки профиля:
hmm2build profile.out sample.fasta
hmm2calibrate profile.out
Ссылка на файл списка последовательностей
Ссылка на файл с выравниванием
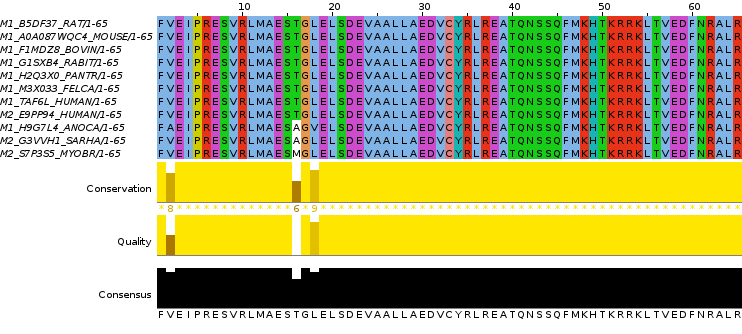
Рис.1. Изображение выравнивания последовательностей выборки
Для поиска среди белков, содержащих рабочий домен:
hmm2search profile.out PF02969whole.fasta > output.txt
Затем с Uniprot были скачаны все последовательности семейства, принадлежащие к таксону Amniota, а из PF02969whole.fasta с помощью скрипта были извлечены все последовательности, которые содержат характерные подпоследовательности, после чего находки из файла output.txt были перенесены в таблицу и обозначены в зависимости от того, принадлежат ли они подсемейству.
Построение частотной гистограммы и ROC-кривой
По полученным данным была построена гистограмма частотности находок по значениям весов:
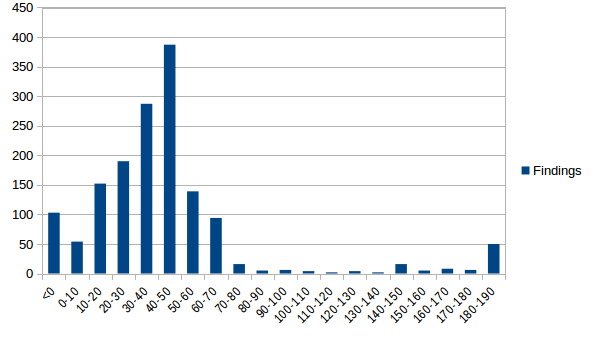
Рис.2. Гистограмма частот находок в зависимости от веса
Также построена ROC-кривая (график True Positive Rate от False Positive Rate):
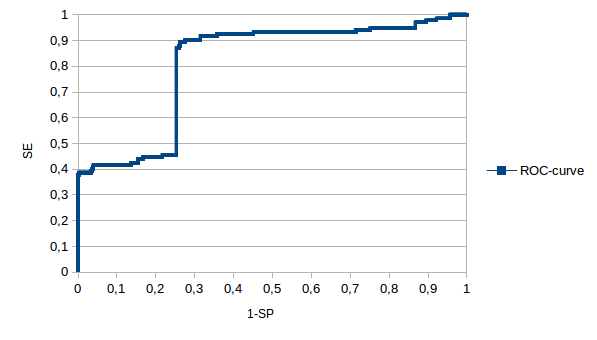
Рис.3. ROC-кривая для определения подсемейства по профилю нашей выборки
Порог для профиля был выбран по одновременному максимуму суммы специфичности и чувствительности, что можно найти по максимуму величины SE + SP - 1, которая составила 0,63 при пороге e-value 6,10E-12 и пороге веса 48,1. Специфичность при данном пороге составляет 0,74; селективность - 0,89.
Таблица 1.
| На самом деле | Принадлежит подсемейству | Не принадлежит подсемейству | Сумма |
| Выше порога по профилю | 118 | 367 | 485 |
| Ниже порога | 14 | 1031 | 1045 |
| Сумма | 132 | 1398 | 1530 |
С чем связана такая форма ROC-кривой? Скорее всего, с определением подсемейства, поскольку изначально выбраны в качестве признака два фрагмента последовательности, которая является практически целиком консервативной, соответствие профилю же считается по всей последовательности. Поэтому вполне возможно, что большое количество FP-результатов - следствие наличия замен в характерных подпоследовательностях, при общем высоком сходстве с профилем. Таким образом, если переформулировать определение нашего подсемейства, можно получить лучшую ROC-кривую. Действительно, переопределим наше подсемейство, как белки Amniota, чьи последовательности описываются регулярным выражением: "F.EIPRESVRLMAES.G.ELSDEVAALLAEDVCYRLREATQNSSQFMKHTKRRKLTVEDFNRALR", извлечем все эти последовательности скриптом, и заново построим ROC-кривую:
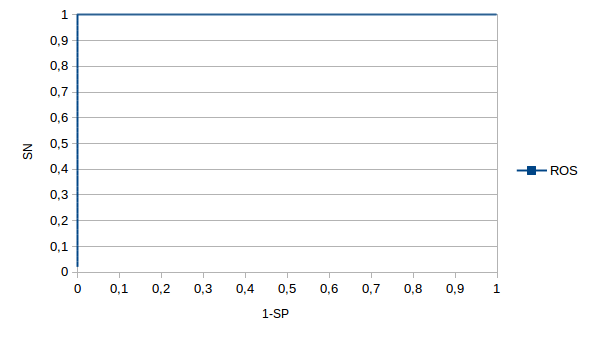
Рис.4. ROC-кривая для определения нового подсемейства по профилю.
Ссылка на таблицу с результами по новому семейству
Как можно видеть, данная кривая соответствует идеальной ROC-кривой, при которой можно определить порог, отделяющий подсемейство без ложных результатов. Таковым порогом по E-value является 4.1e-54, по весу - 188.2, показатель SN + SP - 1 здесь равен 1.
Таблица 2.
| На самом деле | Принадлежит подсемейству | Не принадлежит подсемейству | Сумма |
| Выше порога по профилю | 48 | 0 | 48 |
| Ниже порога | 0 | 1482 | 1482 |
| Сумма | 48 | 1482 | 1530 |
Таким образом, мы выделили консервативное подсемейство в весьма разнородном семействе (см. выравнивания), члены которого присутствуют в основном среди млекопитающих и 1 экземпляр известен среди рептилий (возможно, это связано с недостаточной представленностью белков рептилий в Uniprot).