Выбор эукариотического организма
Для выполнения задания я выбрал муху дрозофилу (англ. fruit fly, лат. Drosophila melanogaster). В NCBI Datasets нашлась референсная сборка генома, которая аннотированные гены и имеет качество "Chromosome".
| Параметр | Значение |
|---|---|
| GenBank Accession | GCA_000001215.4 |
| RefSeq Accession | GCF_000001215.4 |
| Уровень сборки | Chromosome |
| Размер генома | 143.7 Mb |
| Количество хромосом | 7 |
| Количество скаффолдов | 1869 |
| Количество контигов | 2441 |
| Scaffold N50 | 25.3 Mb |
| Scaffold L50 | 3 |
| Contig N50 | 21.5 Mb |
| Contig L50 | 3 |
Контиг - непрерывная последовательность нуклеотидов ДНК, собранная из чтений.
Скаффолд - последовательность из нескольких контигов, стоящих в правильном месте, в правильном направлении, но мы не знаем, что между ними находится.
N50 Contig - такая наименьшая длина контига, что >=50% сборки покрыто контигами такой и/или более длины.
L50 Contig - такое количество контигов, в которой содержится 50% всех нуклеотидов сборки
Инсулин в NCBI и ENA
Я ввел insulin[Title] и выдало 45020 записей, 4129 из которых по ДНК, 36529 - по мРНК, 12747 записей принадлежит GenBank, 32270 - RefSeq
(insulin[Title]) AND "homo sapiens"[Organism], по такому запросу выдало 5609 записей: 1472 по ДНК, 4093 по мРНК, 5355 из GenBank, 254 из RefSeq.
В ENA по запросу tax_eq(9606) AND description="insulin" выдало 9341 записей: 157 по ДНК(tax_eq(9606) AND description="insulin" AND mol_type="mrna" - запрос)
Поиск в геноме эукариота гена, кодирующего дельта субъединицу АТФ синтазы
Для поиска нужного протеина я использовал поиск по шаблону "ATP synthase, delta", нашлось две записи: изоформы А и В, для задания я использую изоформу B, АС в NCBI Proteins: NP_001259397.1, RefSeq: NM_001272468.1.

Идентификатор белка: NP_001259397.1 (ссылка на NCBI)
Идентификатор нуклеотидной записи, к которой относится белок: XM_036847100
Координаты кодирующей части белка: 112-585
Кодирующая часть гена в FASTA файле
Разные алгоритмы BLAST
Так как муха дрозофила это первичноротое животное, для задания я решил использовать поиск по таксону Кошачьи (Felidae).
Сначала я использовал blastn, чтобы найти гомологичные гены, которые ищет по нуклеотидным последовательностям.
Параметры blastn:
- Database: refseq_genomes
- Organism: Felidae (taxid:9681)
- Max target sequences: 100
- Expect treshold: 0.05
- Wordsize: 7
- Match/Mismatch Scores: 2/-3
- Gap costs: existence-5, extension-2
По таким параметрам нашлось несколько находок:

Также поищем с помощью tblastx, который переводит нуклеотидную последовательность в аминокислотную и у запроса, и у последовательностей из нуклеотидной базы данных во все 6 возможных вариантов.
Параметры tblastx:
- Database: refseq_genomes
- Organism: Felidae (taxid:9681)
- Max target sequences: 100
- Expect treshold: 0.05
- Wordsize: 2
- Matrix: BLOSSUM62
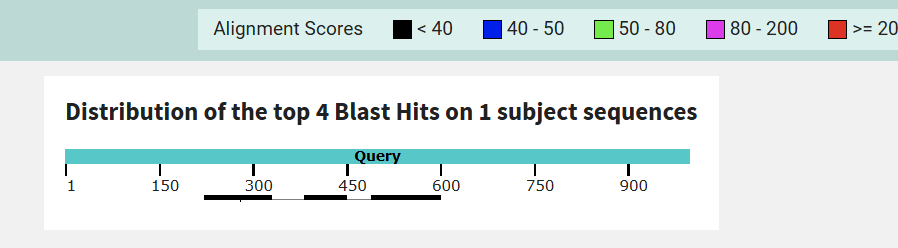
На удивление этот метод дал всего одну находку с не самым хорошим весом, которая не совпадает с находками blastn.
tblastn оставляет аминокислотную последовательность, а нуклеотидную базу данных транслирует в 6 возможных вариантов и ищет находки по им:
Параметры tblastn:
- Database: refseq_genomes
- Organism: Felidae (taxid:9681)
- Max target sequences: 100
- Expect treshold: 0.05
- Wordsize: 5
- Matrix: BLOSSUM62

Поиск в геноме мухи дрозофилы генов основных рибосомальных РНК по далекому гомологу
Сначала я создал db по геному мухи дрозофила с помощью:
makeblastdb -dbtype nucl -in gcf.fna -out dbpr8
Затем скачал файл с двумя рРНК E.coli и поместил записи в rRNA_ecoli1.fasta и rRNA_ecoli2.fasta. Затем я сначала провел поиск с помощью blastn, который не дал никаких находок:
blastn -db dbpr8 -query rRNA_ecoli1.fasta -word_size 7 -outfmt 7 -out blastn1
blastn -db dbpr8 -query rRNA_ecoli2.fasta -word_size 7 -outfmt 7 -out blastn2
В выдаче для 16S rRNA всего найдено 53 находки по таблице, но если посмотреть, то некоторые находки друг друга "продолжают", т.е. три находки могут быть одним гомологом, всего такиех гомологов таким образом получается 23. Нарисуем для 16S rRNA схему с одним таким гомологом:
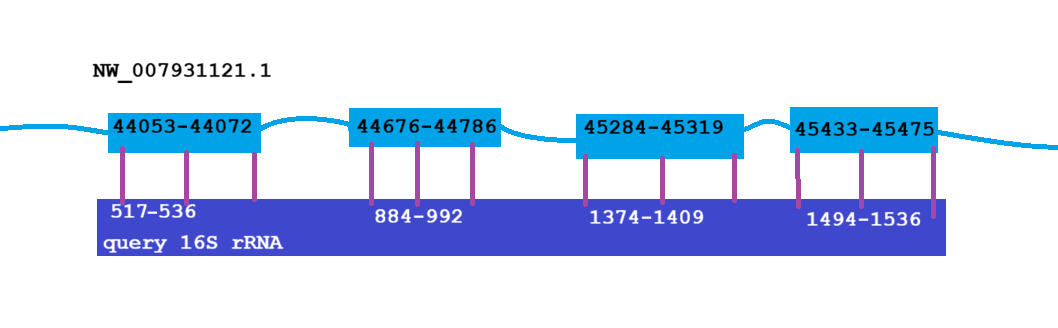
Разные "коробки" сверху - это разные находки в таблице.
Карта локального сходства
Для построение карт я выбрал вирус оспы человека (NC_001611) и обезьянью оспу (NC_063383).
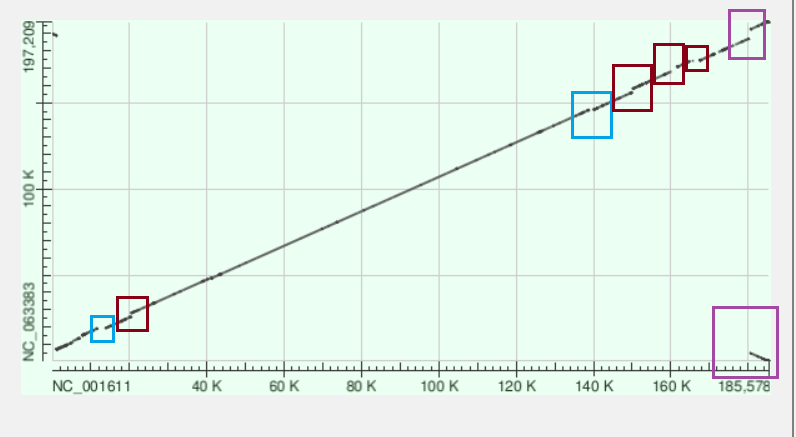
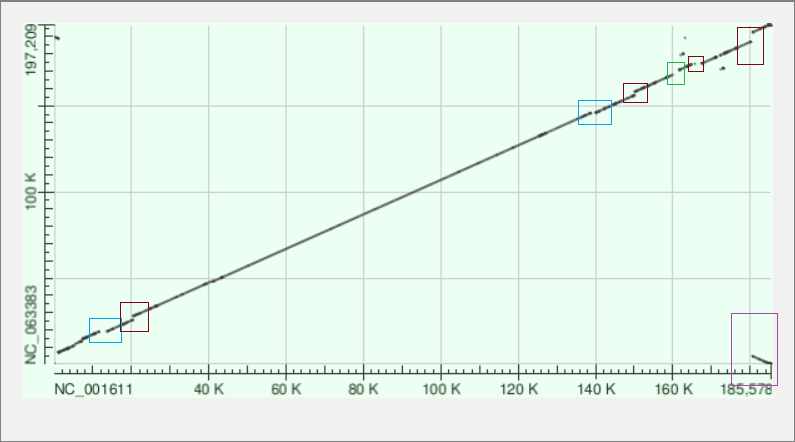
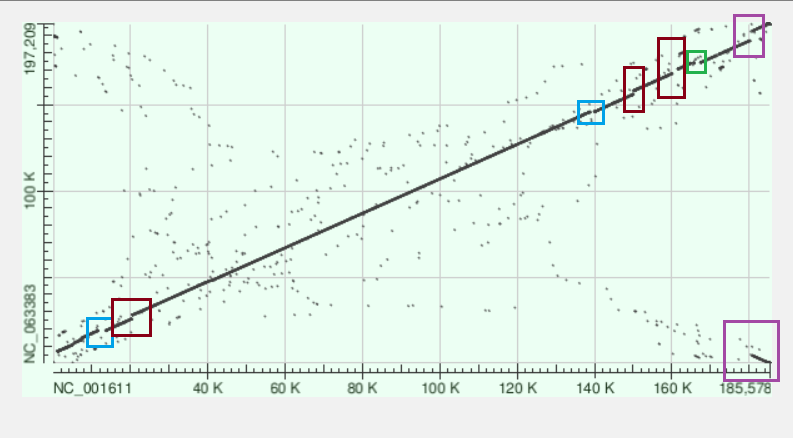
Синим я пометил вставки, красным - делеции, фиолетовым - инверсии, также на картах есть различные транслокации и дупликации очень маленьких участков (особенно на карте для tblastx).