С помощью Putty присоединимся к серверу kodomo.cmm.msu.ru и перейдем в рабочую папку term3/block1/pr3.
Для указания пути к 3DNA используем следующие команды:
export PATH=${PATH}:/home/preps/golovin/progs/X3DNA/bin
export X3DNA=/home/preps/golovin/progs/X3DNA
Далее c помощью программы fiber пакета 3DNA получаемм A-, B- и Z-форму дуплекса ДНК
(одна из нитей - gatcgatcgatcgatcgatc). (PDB файлы можно посмотреть)
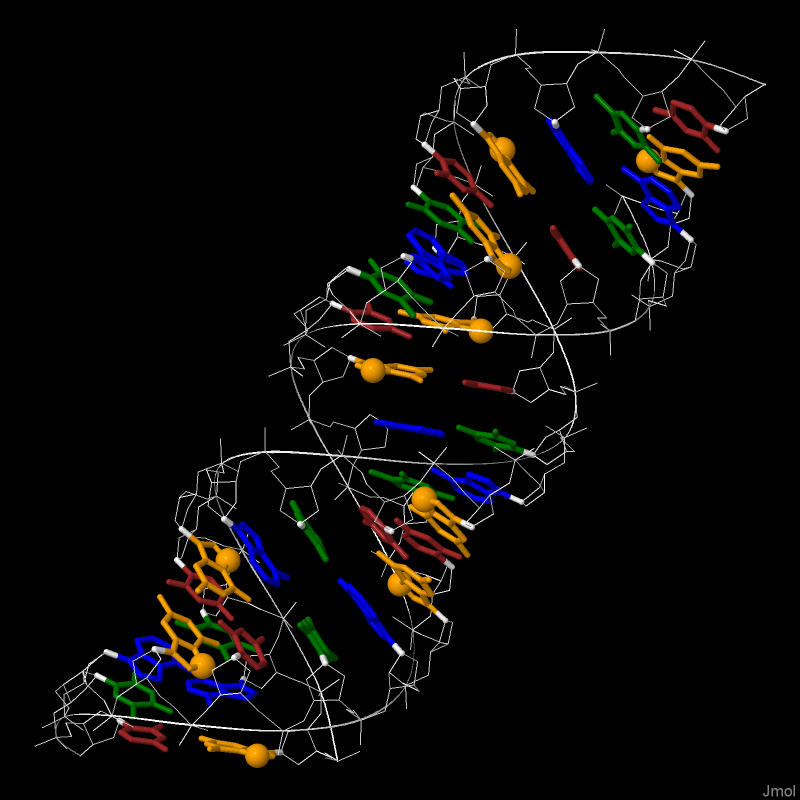
На данном рисунке, используя файл А-формы (gatc-a.pdb) были выделены:
- Нуклеотиды (wireframe=50):
- Аденин - синим
- Тимин - зеленым
- Гуанин - коричневым
- Отдельно выделены атомы (N7 во всех гуанинах)
- Цитозин - коричневым
- Сахарофосфатный остов ДНК - белым (wireframe=10; ribbons=10)
Далее в файле B-формы были соответственно отмечены большая и малая бороздки:
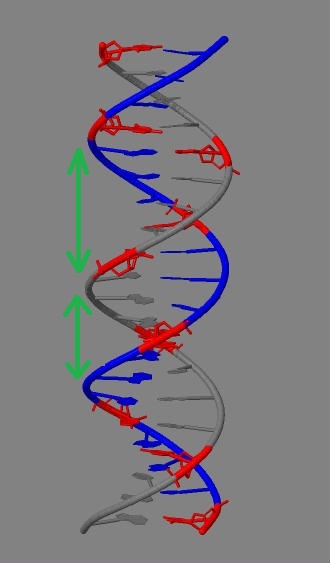 На рисунке серым и синим выделены цепочки ДНК, а красным - цитозин.
На рисунке серым и синим выделены цепочки ДНК, а красным - цитозин.
С помощью ChemSketch получено изображение цитозина (sk2-файл):
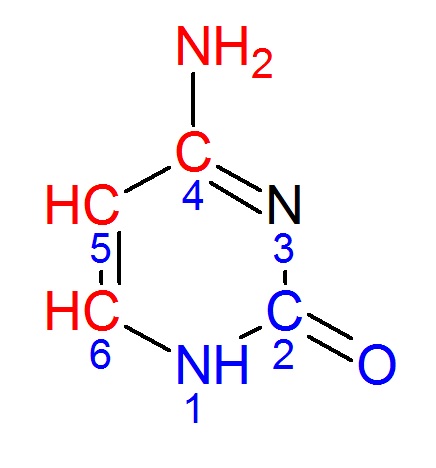
В сторону большой бороздки обращены атомы с12.c5, c12.c6, c12.с4, c12.n4
В сторону малой бороздки обращены атомы c12.c2, c12.n1, c12.o2
Остальные атомы основания не имеют четкого направления.
Затем были измерены основные спиральные параметры разных форм ДНК:
| А-форма | B-форма | Z-форма |
| Тип спирали (правая или левая) | Правая | Левая | Левая |
| Шаг спирали (A) | 28,03 | 33,75 | 43,5 |
| Число оснований на виток | 11 | 10 | 12 |
| Ширина большой бороздки | 16.81 (A6A.P-G37B.P) | 17.21 (C4A.P-A34B.P) | 18.3 (C6A.P-C12B.P) |
| Ширина малой бороздки | 7.98 (A6A.P-G29B.P) | 11.69 (T11A.P-A34B.P) | 8.68 (G9A.P-G17B.P) |
Сравнение торсионных углов в структурах А- и В-формах ДНК:
|
α (P - O5') |
β (O5' - C5') |
γ (C5' - C4') |
δ (C4' - C3') |
ε (C3' - O3') |
ξ (O3' - P) |
χ (C1' - N) |
| A-форма |
64.1 |
173 |
41.7 |
79.1 |
-147.8 |
-75.1 |
-157.2 |
| B-форма |
-29.9 |
136.4 |
31.1 |
143.4 |
-140.8 |
-160.5 |
-98.0 |
Далее значения полученных торсионных углов были сравнены со значениями из презентации:
|
α (P - O5') |
β (O5' - C5') |
γ (C5' - C4') |
δ (C4' - C3') |
ε (C3' - O3') |
ξ (O3' - P) |
χ (C1' - N) |
| A-форма |
62 |
173 |
52 |
88/3 |
178 |
-50 |
-160 |
| B-форма |
63 |
171 |
54 |
123/131 |
155 |
-90 |
-117 |
Как заметно из этих двух таблиц: значения различны. Это происходит потому что измерения проводились при помощи разных атомов.
Определение параметров структур нуклеиновых кислот с помощью программ пакета 3DNA.
Так как пакет 3DNA работает только со старым форматом PDB, потребовалось перевести файлы в старый формат при помощи команды:
remediator --old ''XXXX.pdb'' > ''XXXX_old.pdb
Далее была получена информация о структурах 1F7U и 1TRO при помощи команды:
find_pair -t XXXX_old.pdb stdout | analyze
Далее в файлах 1F7U.out и 1TRO.out была найдена информация о торсионных углах, водородных связях, стекинг-взаимодействиях и так далее.
Таблица с торсионными углами структуры 1TRO.
Из таблицы видно, что максимально отклоняется в первой цепи 4С, а во второй цепи - 34C.
В файле 1F7U_old.out была найдена информация о структуре тРНК, цветом выделены пары оснований, образующие стебли тРНК.

В этом же файле была найдена информация о неканонических парах, на таблице ниже они выделены цветом.
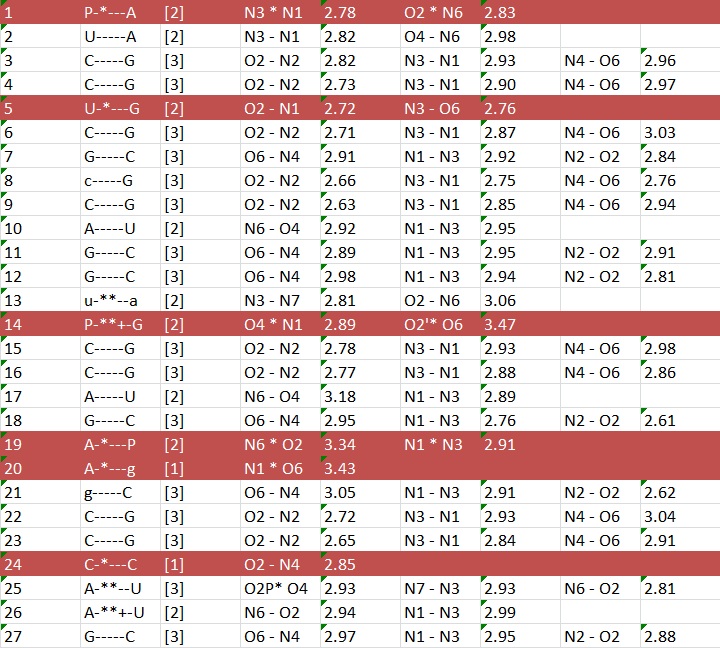
В структурах РНК встречаются так называемые стекинг-взаимодействия, возникающие между расположенными друг над другом основаниями. Сила взаимодействия определяется площадью перекрывания этих оснований. Информацию о стекинг-взаимодействиях в структуре тРНК можно найти в том же файле, см. таблицу ниже. Зеленым цветом выделены минимально перекрывающиеся основания, а голубым-максимально.
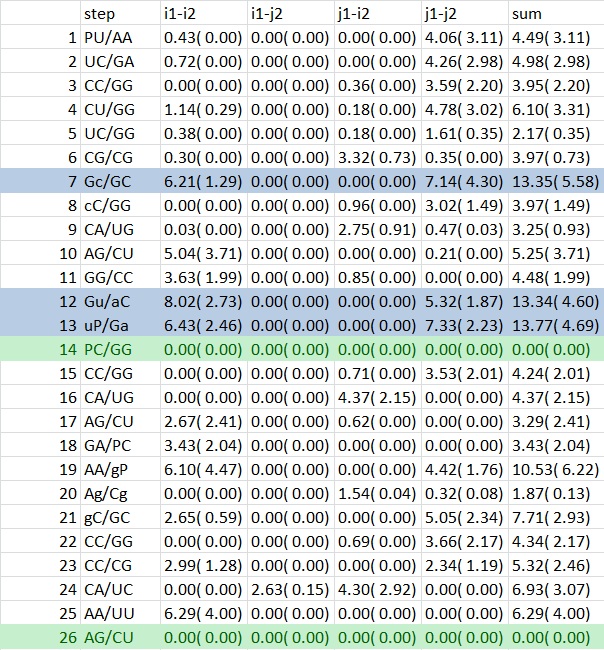
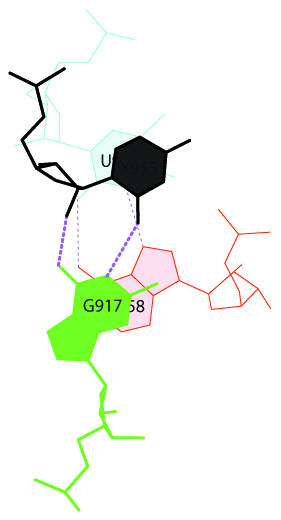
Графическое изображение минимального и максимального перекрываний можно получить с помощью команд:
ex_str -X stacking.pdb stepX.pdb
stack2img -cdolt stepX.pdb stepX.ps
Максимально перекрывающиеся основания:
Минимально перекрывающиеся основания:







 На рисунке серым и синим выделены цепочки ДНК, а красным - цитозин.
На рисунке серым и синим выделены цепочки ДНК, а красным - цитозин.




